É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um método para medir o metabolismo em Ordenado subpopulações de Comunidades celulares complexas usando Stable Isotope Tracing
Neste Artigo
Resumo
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
Resumo
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
Introdução
organismos superiores contêm comunidades complexas de tipos de células distintas que colaboram para funções mais complexas. Por exemplo, tumores contêm não só as células cancerosas, mas também fibroblastos, células que constituem os vasos sanguíneos, e muitas vezes de células imunes infiltrados 1; sangue contém uma mistura complexa de dezenas de subtipos de células imunológicas 2; e ainda linhas de células cultivadas pode ser constituída por várias subpopulações, tal como o lúmen e subtipos de células basais do cancro da mama 3. Além disso, os tipos de células distintas que coexistem podem exibir "colaboração" metabólica. Por exemplo, no cérebro, astrócitos são pensados para converter a glicose em lactato, o qual é, em seguida, "alimentado" para os neurónios que oxidam este substrato 4; Os linfócitos T são, em alguns contextos dependentes de células dendríticas adjacentes como uma fonte de cisteína 5; e as células cancerosas podem colaborar com assofibroblastos ciados em tumores 6. Para compreender o comportamento metabólico de tais sistemas, é essencial para separar e medir as actividades metabólicas dos vários tipos de células presentes.
De longe, o método mais amplamente utilizado para a separação de tipos de células é a classificação de células activadas por fluorescência. Este método é amplamente aplicável, desde que o tipo de célula ou do estado de interesse podem ser "etiquetados" utilizando anticorpos fluorescentes, a expressão de proteínas fluorescentes modificadas, ou outros corantes. Uma opção é tipos de células inicialmente separadas por meio de um classificador de células, re-cultura os tipos de células individuais obtidos, e em seguida, realizar estudos sobre o metabolismo destas culturas 7. No entanto, isto só é possível se o tipo de célula ou fenótipo é estável nas condições de cultura, e não pode captar o comportamento transiente tais como estados do ciclo celular, nem a cooperação metabólica em co-culturas. Para tais casos, o metabolismo deve ser medida directamente no assimcélulas rted. Este é um desafio desde a célula procedimento de triagem submete células a tensões que possam falsear o seu metabolismo 8, e estamos cientes de apenas alguns estudos que tomam esta abordagem 9, 10. Em particular, verificou-se que os principais metabolitos, tais como aminoácidos pode vazar a partir de células mantidas em tampão de separação de células, de modo que as medições de abundância metabolito absoluta não são fiáveis 11 (embora comparação relativa entre as fracções ordenadas podem ainda ser valiosos).
Para contornar esses problemas, nós rotulamos células com isótopos estáveis antes de triagem, e focar os MIDs em metabólitos celulares, em vez de abundâncias de metabólitos. Desde MIDs são formados em escalas de tempo mais longos, devem ser menos afetados pela exposição a curto prazo às condições de classificação. Nós quantificar MIDs usando espectrometria de massa de alta resolução-scan completo, que é sensível o suficiente para fornecer data em centenas de metabólitos a partir de cerca de 500.000 células classificadas, necessitando de cerca de 30-60 minutos de tempo de separação de células. Uma comparação entre um "simulada" classificados de controlo (células passados através do instrumento de separação de células, sem qualquer gating população específica) e extracção metabolito directamente a partir da placa de cultura é feita para garantir que os médios observados são representativos daqueles na cultura original. Dependendo da escolha dos marcadores de isótopos estáveis, várias vias metabólicas podem ser estudada com este método.
Protocolo
1. metabolito Extração
- Extração de prato
- células de cultura em uma placa de 6 poços em triplicado em isótopos marcados meios de cultura estáveis + suplementos dialisados (soro ou outros suplementos de crescimento) até que as células se tornam 75% confluentes.
NOTA: Aqui cultura de células HeLa, durante 48 h em RPMI contendo 40% U- 13 C-glicose e 70% U- 13 C, 15 N2-glutamina e 5% de FBS dialisado (Soro Bovino Fetal). FBS dialisado é usado para se livrar dos pequenos metabólitos peso molecular que possam contaminar os meios de comunicação rotulados. células em suplemento dialisada antes da experiência real, a cultura é recomendada para garantir as células estão a crescer normalmente no meio. Suplementos são dialisados em solução 0,15 M NaCl durante a noite usando tubo de diálise pele de cobra. - No dia de meios de cultura de extração de descarte, lavar os poços duas vezes com HBSS 500 mL fria e depois descartá-lo.
NOTA: Aqui utilizar HBSS contendo 40% de U-13 C-glucose uma vez que também é no meio de cultura. - Adicionar 600 uL de metanol a 100% pré-arrefecido em gelo seco.
- Colocar a cápsula para secar gelo e remover o material celular com um raspador de células.
- Cuidadosamente pipetar os extractos de células para um tubo de microcentrífuga e armazenar a -80 ° C até a análise por espectrometria de massa.
- células de cultura em uma placa de 6 poços em triplicado em isótopos marcados meios de cultura estáveis + suplementos dialisados (soro ou outros suplementos de crescimento) até que as células se tornam 75% confluentes.
- Extracção de células classificadas simulados
- células de cultura em um prato de 100 mm de triplicado em isótopos marcados meios de cultura + suplementos dialisados estáveis.
NOTA: Aqui cultura de células HeLa, durante 48 horas em placa de 100 mm, porque um grande número de células (~ 4 x 10 6) são necessários para obter 500.000 células separadas. Este número de células HeLa extracto foi necessária para obter uma boa medição de metabolitos. O meio de cultura utilizado foi o meio RPMI contendo 40% U- 13 C-glucose e 70% U- 13 C, 15 N2-glutamina e 5% de FBS dialisada. - No dia de extracção, meios de cultura de descarte, lavar os poços wom 1,5 mL quente HBSS e depois descartá-lo.
- Separe as células por adição de 1,5 ml de tripsina / EDTA durante 4 min a 37 ° C. Execute as seguintes etapas em 4 ° C ou em gelo.
- Desactivar tripsina, adicionando 3 mL de gelo HBSS frio (Solução Salina Equilibrada de Hank) + suplemento de diálise.
- Recolher as células num tubo de 15 ml e centrifugar a 750 xg durante 3 min.
- Ressuspender o sedimento em suplemento + EDTA 1 mM + HBSS dialisado a uma concentração de 1-2 x 10 6 células / ml, passar através de 40 uM coadores de células para a obtenção de células individuais, e transferir para um tubo de 5 ml.
- Ordenação de células através de separação de células de propagação única para singletos, e células de centrífuga classificados a 750 xg durante 3 min a 4 ° C.
NOTA: Aqui classificar células HeLa a uma taxa de 1.000 eventos / s, com pressão instrumento de 27 psi, e usando um bico de 100 mm. As células HeLa são grandes para que eles devem ser classificadas em uma taxa de eventos lento e grande bico para obter pellet tão intacto quanto possível. Manter as células em blocos frios throughout triagem para diminuir o metabolismo. Uma descrição completa do processo de classificação é descrito anteriormente 12. - Descartar o sobrenadante e ressuspender o sedimento em 50 mL de gelo frio de dH 2 O para se obter um pelete homogénea antes da adição de metanol. A adição de metanol frio directamente para sedimentar forma um sedimento sólido que é difícil para ressuspender.
- Extrair metabolitos por adição de 540 uL de metanol mantido em gelo seco e manter extractos a -80 ° C até a cromatograf ia líquida - espectroscopia de massa de alta resolução (LC-HRMS) análise.
- células de cultura em um prato de 100 mm de triplicado em isótopos marcados meios de cultura + suplementos dialisados estáveis.
- Extracção do ciclo celular de células classificadas
- células de cultura em um prato de 100 mm de triplicado em meio de cultura + suplementos dialisados.
NOTA: Aqui cultura células HeLa contendo geminin Fucci verde (-MAG1 hGem) da sonda 13, que permite a triagem de G1 (negativo) e células SG2M (positivas). As células podem ser cultivadas em meios rastreio de isótopos estáveis para umaé tempo considerado necessário. Aqui temos cultivados-los por 46 h em meio não marcado, então nós mudaram para RPMI contendo 40% de U- 13 C-glicose e 70% U- 13 C, 15 N2-Glutamina 2 h antes de iniciar a triagem. Isto foi feito a fim de ser capaz de estudar as fases do ciclo celular que requer um pulso curto rotulagem. - No dia da extracção, meios de cultura de descarte, lavar os poços com 1,5 mL HBSS quente e, em seguida, descartá-lo.
- Separe as células por adição de 1,5 ml de tripsina / EDTA durante 4 min a 37 ° C. Execute as seguintes etapas em 4 ° C ou em gelo.
- Desactivar tripsina, adicionando 3 mL gelada HBSS + suplemento de dialisado.
- Recolher as células num tubo de 15 ml e centrifugar a 750 xg durante 3 min.
- Ressuspender o sedimento em suplemento + EDTA 1 mM + HBSS dialisado a uma concentração de 1-2 x 10 6 células / ml, passar através de 40 uM coadores de células para a obtenção de células individuais, e transferir para um tubo de 5 ml.
- Classificar células através do classificador de células gAting a entrada de detritos e dupletos, em seguida, gating para o marcador de células de interesse, e células de centrífuga classificados em 750 xg durante 3 min a 4 ° C.
NOTA: Aqui classificar células HeLa a uma taxa de 1.000 eventos / s, com pressão instrumento de 27 psi, e usando um bico de 100 mm. Manter as células em blocos de frio ao longo de triagem para diminuir a taxa de metabolismo. - Descartar o sobrenadante e ressuspender o sedimento em 50 mL de gelo frio de dH 2 O para se obter um pelete homogénea antes da adição de metanol. A adição de metanol frio directamente para sedimentar forma um sedimento sólido que é difícil para ressuspender.
- Extrair os metabolitos por adição de 540 uL de metanol mantido em gelo seco e manter extractos a -80 ° C até a análise por LC-HRMS.
- células de cultura em um prato de 100 mm de triplicado em meio de cultura + suplementos dialisados.
2. Análise de Espectrometria de Massa
Nota: Aqui nós descrevemos o protocolo para a análise de extratos de células em um sistema LC-HRMS. Quaisquer métodos metabolômicos para análise de extractos celulares pode ser usado. verificação completaA análise pode ser útil na detecção de uma ampla variedade de metabolitos.
- Calibrar o instrumento utilizando uma mistura de calibração de referência espectrometria de massa.
- extractos celulares descongelamento em gelo durante 30 min e agitar com vortex durante 15 s.
- Transferir 100 uL de extracto celular para um filtro de centrifugação e centrifugar durante 10 min a 13000 xg, a 4 ° C.
- Injectar 12,5 uL do filtrado para o sistema de LC-HRMS.
- metabolitos separados utilizando cromatografia líquida zwitteriónico Interacção Hidrofílico (ZIC-HILIC) coluna (150 mm x 4,6 mm, 5 um de tamanho de partícula) equipado com uma coluna de guarda ZIC-HILIC (20 mm x 2,1 milímetros) usando um gradiente de eluição de 0,1% fórmico ácido em água (solvente a) e acetonitrilo (solvente B). Iniciar a eluição em gradiente a 20% de solvente A e aumentar até 80% em 17 min. Manter esta percentagem durante 4 min, com um fluxo de 400 mL min -1 e a temperatura da coluna e da bandeja da amostra a 23 ° C e 4 ° C, respectivamente.
- Use um iINSTRUMENTO acoplado à separação cromatográfica para a detecção de metabolitos, um electropulverização aquecida (H-ESI II) em ambos os modos positivos e negativos como fonte de ionização, e um modo de aquisição de varrimento total em um poder de resolução de massa de 70000 completa largura meia altura (FWHM) ( m / z 200).
- Use azoto (pureza> 99,995%) para o gás de revestimento e gás auxiliar a um caudal de 45 e 10 AU (unidades arbitrárias) e ajustar a temperatura do vaporizador a 350 ° C e a tensão de electrospray a 4 kV no modo positivo e -3,5 kV no modo negativo.
Análise 3. Dados
- Selecione um número de metabolitos para os quais existam normas e que mostram picos de boa qualidade nas amostras. picos de boa qualidade tem alta relação sinal-ruído. É importante verificar a qualidade do pico e certifique-se de não incluir falsos isótopos. picos isotópicos, que diferem entre si em forma e / ou tempo de retenção são susceptíveis falsa.
- Para amostras marcados com isótopos calcular isotopomer de massa (MI) fracções, dividindo a área do pico de cada MI com áreas totais dos picos de todas as IMs.
- Calcular as fracções de carbono / azoto marcados, enriquecimento de 13 C e 15 N, respectivamente, conforme
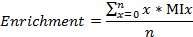
onde n é o número total de átomos de carbono (ou azotos, respectivamente) no metabolito, e misturar a fracção é MI de x.
NOTA: Todos os cálculos podem ser realizados utilizando linguagens de programação.
Resultados
Como um exemplo, aqui descrevemos uma experiência investigar o metabolismo das células HeLa classificados de acordo com a fase do ciclo celular. Para identificar uma grande variedade de metabolitos centrais em ambos os átomos de carbono e átomos de azoto, que as células cultivadas durante 48 h, usando U- 13 C-glicose e U- 13 C, 15 N-glutamina como marcadores. Para obter MIDs ricos para a experiência de validação, que escolheu uma mistura de 40% U-...
Discussão
O nosso método é baseado no princípio de que em MIDs metabolitos celulares reflectir a "história" de actividades metabólicas de uma célula. Isto faz com que seja possível investigar actividades metabólicas na subpopulação de células, uma vez que ocorreram na comunidade complexo de células, antes do processo de separação de células. Em contraste, as áreas dos picos de metabólitos diferem acentuadamente entre os extratos de células classificadas e extração direta da placa de cultura
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
Materiais
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
Referências
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados