Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Une méthode pour mesurer le métabolisme dans des communautés triées sous-populations cellulaires complexes Utilisation Stable Isotope Tracing
Dans cet article
Résumé
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
Résumé
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
Introduction
les organismes supérieurs contiennent des communautés complexes de types cellulaires distincts qui collaborent pour réaliser des fonctions plus complexes. Par exemple, les tumeurs contiennent non seulement les cellules cancéreuses, mais aussi des fibroblastes, des cellules qui constituent les vaisseaux sanguins et de cellules immunitaires souvent infiltrats 1; sang contient un mélange complexe de plusieurs dizaines de sous - types de cellules immunitaires 2; et même des lignées cellulaires cultivées peuvent être constituées de plusieurs sous - populations, comme l'luminale et les sous - types de base de cellules de cancer du sein 3. De plus, les types de cellules distinctes qui coexistent peut présenter métabolique "collaboration". Par exemple, dans le cerveau, les astrocytes sont pensés pour convertir le glucose en lactate qui est ensuite «alimenté» pour les neurones qui oxydent ce substrat 4; Les lymphocytes T sont dans certains contextes , qui dépendent des cellules dendritiques adjacentes en tant que source de cystéine 5; et les cellules cancéreuses peuvent collaborer avec des assofibroblastes ciés dans les tumeurs 6. Pour comprendre le comportement métabolique de tels systèmes, il est nécessaire de séparer et de mesurer les activités métaboliques des différents types présents de cellules.
De loin, la méthode la plus largement utilisée pour séparer les types de cellules est le tri cellulaire activé par fluorescence. Ce procédé est largement applicable, à condition que le type de cellule ou d'un état d'intérêt peuvent être "marqués" en utilisant des anticorps fluorescents, l'expression de protéines fluorescentes modifiées, ou d'autres colorants. Une option est de types de cellules initialement séparées par un trieur de cellules, re-culture les types cellulaires individuels obtenus, puis effectuer des études sur le métabolisme de ces cultures 7. Toutefois, cela est seulement possible si le type de cellule ou d'un phénotype est stable dans des conditions de culture, et ne peuvent pas capturer le comportement transitoire tels que les états du cycle cellulaire, ni la coopération métabolique dans les co-cultures. Pour de tels cas, le métabolisme doit être mesurée directement sur tantcellules rted. Cela est difficile car la cellule procédure de tri soumet les cellules à des contraintes qui peuvent fausser leur métabolisme 8, et nous sommes conscients de seulement quelques études qui suivent cette approche 9, 10. En particulier, nous avons constaté que les principaux métabolites tels que les acides aminés peuvent fuir à partir de cellules conservées dans un tampon de tri cellulaire, de sorte que les mesures de l' abondance absolue des métabolites ne sont plus fiables 11 (bien que la comparaison relative entre fractions triées peut encore être utile).
Pour contourner ces problèmes, nous appelons les cellules avec des isotopes stables avant le tri, et de se concentrer sur les MIDs en métabolites cellulaires, plutôt que les abondances métabolites. Depuis MIDs sont formés sur des échelles de temps plus longues, ils devraient être moins affectés par l'exposition à court terme à des conditions de tri. Nous quantifions MIDs utilisant-balayage complet à haute résolution spectrométrie de masse, ce qui est assez sensible pour fournir data sur des centaines de métabolites à partir de environ 500.000 cellules triées, nécessitant environ 30-60 min de la cellule de temps à trier. Une comparaison entre un "mock triée" contrôle (cellules transmises à travers l'instrument de trieur de cellules sans gating une population spécifique) et l'extraction des métabolites directement à partir de la boîte de culture est faite pour veiller à ce que les MIDs observés sont représentatifs de ceux de la culture d'origine. En fonction du choix des traceurs isotopiques stables, diverses voies métaboliques peuvent être étudiés par cette méthode.
Protocole
1. Métabolite Extraction
- Extraction du plat
- les cellules de culture dans une plaque à 6 puits en triple dans isotope marqué les milieux de culture stables + suppléments dialysés (sérum ou autres suppléments de croissance) jusqu'à ce que les cellules deviennent 75% confluentes.
Remarque: ici , la culture des cellules HeLa pendant 48 h dans un milieu RPMI contenant 40% U- 13 C-glucose et 70% U- 13 C, 15 N2-glutamine et 5% de FBS dialyse (sérum bovin fœtal). Dialysée FBS est utilisé pour se débarrasser des petits métabolites de poids moléculaire qui pourrait contaminer les médias étiquetés. La culture de cellules en supplément dialysée avant l'expérience réelle est recommandée pour assurer les cellules se développent normalement dans le milieu. Les suppléments sont dialysées dans 0,15 M de NaCl solution durant la nuit en utilisant un tube de dialyse de la peau de serpent. - Au jour de l'extraction défausse des milieux de culture, rincer les puits deux fois avec HBSS 500 ul froide, puis jetez-le.
REMARQUE: Ici utiliser HBSS contenant 40% U-13C-glucose car il est aussi dans le milieu de culture. - Ajouter 600 pi à 100% de méthanol pré-refroidi sur de la glace sèche.
- Transférez le plat pour sécher la glace et enlever de la matière cellulaire avec un grattoir cellulaire.
- Pipetter prudemment les extraits cellulaires à un tube de microcentrifugeuse et conserver à -80 ° C jusqu'à l'analyse par spectrométrie de masse.
- les cellules de culture dans une plaque à 6 puits en triple dans isotope marqué les milieux de culture stables + suppléments dialysés (sérum ou autres suppléments de croissance) jusqu'à ce que les cellules deviennent 75% confluentes.
- Extraction de cellules triées simulacres
- les cellules de la culture dans une boîte de 100 mm en triple en isotopes étiquetés milieux de culture + suppléments dialysés stables.
NOTE: Voici la culture des cellules HeLa pendant 48 h à 100 mm plat car un nombre élevé de cellules (~ 4 x 10 6) sont nécessaires pour obtenir 500.000 cellules triées. Ce nombre de HeLa extrait de cellules a été nécessaire pour obtenir une bonne mesure des métabolites. Le milieu de culture utilisé était RPMI contenant 40% U- 13 C-glucose et 70% U- 13 C, 15 N2-glutamine et 5% de FBS dialyse. - Au jour de l'extraction, les milieux de culture défausse, rincer les puits with chaud 1,5 ml de HBSS, puis jetez-le.
- Détacher les cellules en ajoutant 1,5 trypsine mL / EDTA pendant 4 minutes à 37 ° C. Effectuez les étapes suivantes dans 4 ° C ou dans de la glace.
- Désactiver la trypsine en ajoutant 3 ml de glace HBSS froid (solution saline équilibrée de Hank) + supplément dialysée.
- Recueillir les cellules dans un tube de centrifugeuse de 15 ml et à 750 g pendant 3 min.
- Reprendre le culot dans HBSS + dialysée supplément + 1 mM EDTA à une concentration de 1-2 x 10 6 cellules / mL, passent à travers 40 um crépines de cellules pour obtenir des cellules individuelles, et transférer dans un tube de 5 ml.
- Trier les cellules par trieur de cellules gating seulement pour singulets, et les cellules de centrifugation triées à 750 g pendant 3 min à 4 ° C.
NOTE: Voici trier les cellules HeLa à un taux de 1000 événements / s, avec la pression de l'instrument 27 psi, et en utilisant une buse de 100 um. Les cellules HeLa sont grandes de sorte qu'ils doivent être triés à un taux d'événement lent et large buse pour obtenir culot aussi intact que possible. Gardez les cellules dans des blocs froids throughout tri pour diminuer le métabolisme. Une description détaillée de la procédure de tri est décrit précédemment 12. - Jeter le surnageant et remettre le culot dans 50 ul de la glace dH froide 2 O pour obtenir un culot homogène avant d' ajouter du méthanol. L'addition de méthanol froid directement à granulés forme une pastille solide qui est difficile à remettre en suspension.
- Extrait métabolites en ajoutant 540 ul de méthanol maintenu dans la glace sèche et de garder des extraits à -80 ° C jusqu'à ce que la chromatographie liquide - spectrométrie de masse à haute résolution (LC-HRMS) analyse.
- les cellules de la culture dans une boîte de 100 mm en triple en isotopes étiquetés milieux de culture + suppléments dialysés stables.
- Extraction du cycle cellulaire des cellules triées
- les cellules de la culture dans une boîte de 100 mm en triple dans les milieux de culture + suppléments dialysés.
NOTE: Voici la culture des cellules HeLa contenant Géminine Fucci Green (mag1-hGem) sonde 13 qui permet le tri de G1 (négatif) et SG2M cellules (positives). Les cellules peuvent être cultivées dans des milieux d'isotopes stables pour un traçages longtemps que nécessaire. Ici , nous les avons mis en culture pendant 46 h dans des milieux non marqué, nous sommes passés à RPMI contenant 40% U- 13 C-glucose et 70% U- 13 C, 15 N2-glutamine 2 h avant de commencer le tri. Cela a été fait afin d'être en mesure d'étudier les phases du cycle cellulaire qui exige un étiquetage à impulsions courtes. - Au jour de l'extraction, les milieux de culture défausse, rincer les puits avec 1,5 mL HBSS chaud puis le jeter.
- Détacher les cellules en ajoutant 1,5 trypsine mL / EDTA pendant 4 minutes à 37 ° C. Effectuez les étapes suivantes dans 4 ° C ou dans de la glace.
- Désactiver la trypsine en ajoutant 3 ml de glace froide HBSS + supplément dialysée.
- Recueillir les cellules dans un tube de centrifugeuse de 15 ml et à 750 g pendant 3 min.
- Reprendre le culot dans HBSS + dialysée supplément + 1 mM EDTA à une concentration de 1-2 x 10 6 cellules / mL, passent à travers 40 um crépines de cellules pour obtenir des cellules individuelles, et transférer dans un tube de 5 ml.
- Trier les cellules à travers le trieur de cellules gAting les débris et les doublets, puis gating pour le marqueur de cellule d'intérêt, et les cellules de centrifugation triées à 750 g pendant 3 min à 4 ° C.
NOTE: Voici trier les cellules HeLa à un taux de 1000 événements / s, avec la pression de l'instrument 27 psi, et en utilisant une buse de 100 um. Gardez les cellules dans des blocs froids tout au long de tri pour diminuer le taux de métabolisme. - Jeter le surnageant et remettre le culot dans 50 ul de la glace dH froide 2 O pour obtenir un culot homogène avant d' ajouter du méthanol. L'addition de méthanol froid directement à granulés forme une pastille solide qui est difficile à remettre en suspension.
- Extraire les métabolites en ajoutant 540 ul de méthanol maintenu dans la glace sèche et de garder des extraits à -80 ° C jusqu'à l'analyse LC-HRMS.
- les cellules de la culture dans une boîte de 100 mm en triple dans les milieux de culture + suppléments dialysés.
2. Analyse de spectrométrie de masse
Note: Nous décrivons ici le protocole d'analyse des extraits de cellules sur un système LC-HRMS. Toutes les méthodes de la métabolomique pour l'analyse d'extraits cellulaires peuvent être utilisés. Scan completl'analyse pourrait être utile pour la détection d'un large éventail de métabolites.
- Calibrer l'appareil en utilisant un étalonnage mix de référence de la spectrométrie de masse.
- des extraits cellulaires Décongeler sur la glace pendant 30 minutes et le vortex pendant 15 s.
- Transférer 100 pl de l'extrait cellulaire à un filtre et centrifuger pendant 10 min à 13 000 xg à 4 ° C.
- Injecter 12,5 ul du filtrat sur le système LC-HRMS.
- métabolites distincts à l'aide d'une interaction hydrophile zwitterionique chromatographie liquide (ZIC-HILIC) colonne (150 mm x 4,6 mm, 5 um de taille de particule) équipé d'une colonne de garde ZIC-HILIC (20 mm x 2,1 mm) en utilisant un gradient d'élution de 0,1% acide formique l'acide dans l'eau (solvant A) et de l'acétonitrile (solvant B). Démarrer l'élution en gradient à 20% de solvant A et augmenter jusqu'à 80% en 17 min. Maintenir ce pourcentage pendant 4 minutes avec un débit de 400 ul min -1 et la température de la colonne et le plateau d'échantillon à 23 ° C et 4 ° C, respectivement.
- Utilisez un iNSTRUMENT couplée à la séparation chromatographique pour la détection de métabolites, d'un électrospray chauffé (H-ESI II) dans les deux modes positif et négatif en tant que source d'ionisation, et un mode d'acquisition scan complet à un pouvoir de résolution en masse de 70 000 largeur à mi-hauteur (FWHM) ( m / z 200).
- Utilisez l'azote (pureté> 99,995%) pour le gaz de gaine et gaz auxiliaire à un débit de 45 et 10 au (unités arbitraires) et régler la température de vaporisation à 350 ° C et la tension d'électronébulisation à 4 kV en mode positif et -3,5 kV en mode négatif.
Analyse 3. Données
- Sélectionnez un certain nombre de métabolites pour lesquels des normes sont disponibles et qui montrent de bons pics de qualité dans les échantillons. De bons pics de qualité ont élevé le rapport signal sur bruit. Il est important de vérifier la qualité de pointe et assurez-vous de ne pas inclure de faux isotopes. pics isotopiques qui diffèrent dans la forme et / ou le temps de rétention sont susceptibles faux.
- Pour les échantillons marqués par des isotopes calculer isotopomère masse (MI) fractions en divisant la zone de pointe de chaque MI avec des zones totales de pointe de tous les MIs.
- Calculer les fractions carbone / azote marqué, l' enrichissement de 13 C et 15 N, respectivement,
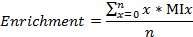
où n est le nombre total de carbones (ou azotes, respectivement) dans le métabolite, et MIx est la fraction MI de x.
NOTE: Tous les calculs peuvent être effectués en utilisant des langages de programmation.
Résultats
A titre d'exemple, nous décrivons ici une expérience d'instruction du métabolisme des cellules HeLa triés en fonction de la phase du cycle cellulaire. Étiqueter un large éventail de métabolites centraux sur les deux atomes de carbone et des atomes d' azote, on a cultivé les cellules pendant 48 heures en utilisant U- 13 C-glucose et U- 13 C, 15 N-glutamine comme traceurs. Pour obtenir MID riche pour l'expérience de validation, on a...
Discussion
Notre méthode est basée sur le principe que MIDs dans les métabolites cellulaires reflètent l ' «histoire» des activités métaboliques d'une cellule. Ceci permet d'étudier les activités métaboliques dans les sous- population de cellules, car ils se sont produits dans le milieu complexe de cellules, préalablement à la procédure de tri cellulaire. En revanche, les zones de pointe de métabolites diffèrent sensiblement entre les extraits de cellules triées et extraction directe à partir de la bo...
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
matériels
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
Références
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon