È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un metodo per misurare il metabolismo in Ordinati sottopopolazioni di cellule Comunità complessi utilizzando Stable Isotope Tracing
In questo articolo
Riepilogo
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
Abstract
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
Introduzione
organismi superiori contengono comunità complesse di tipi cellulari diversi che collaborano per realizzare le funzioni più complesse. Ad esempio, i tumori contengono non solo le cellule cancerose, ma anche fibroblasti, cellule che costituiscono i vasi sanguigni e cellule immunitarie spesso infiltrati 1; sangue contiene una miscela complessa di decine di sottotipi di cellule immunitarie 2; e anche le linee cellulari in coltura possono consistere di più sottopopolazioni, come il lume e sottotipi basali delle cellule di carcinoma mammario 3. Inoltre, tipi di cellule distinte che coesistono può esibire metabolica "collaborazione". Ad esempio, nel cervello, astrociti sono pensati per convertire il glucosio in lattato, che è poi "alimentato" ai neuroni che ossidano il substrato 4; I linfociti T sono in alcuni contesti dipendenti cellule dendritiche adiacenti come fonte di cisteina 5; e le cellule tumorali possono collaborare con Assofibroblasti ciati nei tumori 6. Per comprendere il comportamento metabolico di tali sistemi, è essenziale per separare e misurare le attività metaboliche dei vari tipi di cellule presenti.
Di gran lunga il metodo più utilizzato per separare tipi cellulari è fluorescenza-attivato cell sorting. Questo metodo è ampiamente applicabile, a condizione che il tipo di cellula o lo stato di interesse possono essere "etichettati" con anticorpi fluorescenti, espressione di proteine fluorescenti ingegnerizzate, o altri coloranti. Una possibilità è quella di tipi inizialmente separati cellule attraverso un cell sorter, ri-cultura dei singoli tipi di cellule ottenute, e quindi eseguire studi sul metabolismo di queste culture 7. Tuttavia, ciò è possibile solo se il tipo di cellula o fenotipo è stabile in condizioni di coltura, e non possono catturare comportamento transitorio come stati del ciclo cellulare, né la cooperazione metabolica in co-coltura. Per tali casi, il metabolismo deve essere misurata direttamente su cosìcellule ari. Questa è una sfida dal momento che la cellula di smistamento procedura sottopone cellule alle sollecitazioni che possono falsare il loro metabolismo 8, e siamo consapevoli di solo pochi studi che prendono questo approccio 9, 10. In particolare, abbiamo trovato che i principali metaboliti come aminoacidi può fuoriuscire dalle cellule tenuti in memoria di cella di smistamento, in modo che le misurazioni dell'abbondanza metabolita assoluto non sono più affidabili 11 (anche se il confronto relativo tra le frazioni ordinate può essere ancora valido).
Per aggirare questi problemi, etichettiamo le cellule con isotopi stabili prima di ordinamento e concentriamo sui MID di metaboliti cellulari, piuttosto che abbondanze di metaboliti. Dal momento che i MID si formano su scale temporali più lunghi, essi dovrebbero essere meno influenzati da esposizione a breve termine a condizioni di ordinamento. Abbiamo quantificare MID utilizzando la spettrometria di massa ad alta risoluzione full-scan, che è abbastanza sensibile per fornire data su centinaia di metaboliti a partire da circa 500.000 cellule ordinati, che richiede circa 30-60 minuti di tempo l'ordinamento delle cellule. Un confronto tra un "finto allineati" controllo (cellule passati attraverso lo strumento sorter cella senza gating alcuna specifica popolazione) ed estrazione metabolita direttamente dal piatto di coltura è fatto per assicurare che le MIDs osservati sono rappresentativi di quelli nella cultura originale. A seconda della scelta di isotopi stabili, varie vie metaboliche possono essere studiate con questo metodo.
Protocollo
1. Estrazione Metabolite
- Estrazione da piatto
- cellule di coltura in una piastra da 6 pozzetti in triplicato in isotopi etichettato terreni di coltura stabile + supplementi dializzati (siero o altri integratori di crescita) fino a quando le cellule diventano il 75% confluenti.
NOTA: Qui coltura cellule HeLa per 48 ore in RPMI contenente 40% di U- 13 C-glucosio e il 70% U 13 C, 15 N2-glutammina e 5% dializzato FBS (siero fetale bovino). Dializzato FBS viene usato per liberarsi dei piccoli metaboliti peso molecolare che possano contaminare i media etichettati. Si raccomanda coltura di cellule in appendice dializzato prima della esperimento reale per garantire cellule crescono normalmente in mezzo. I supplementi sono dializzati in 0,15 M di NaCl soluzione durante la notte utilizzando pelle di serpente tubo di dialisi. - Al giorno di terreni di coltura di estrazione degli scarti, lavare i pozzi due volte con 500 microlitri HBSS freddo e poi disfarsene.
NOTA: Qui utilizzare HBSS contenente il 40% U-13 C-glucosio essendo anche nei mezzi di coltura. - Aggiungere 600 ml metanolo 100% pre-raffreddata in ghiaccio secco.
- Trasferire il piatto di ghiaccio secco e rimuovere il materiale delle cellule con un raschietto cellulare.
- pipetta con attenzione gli estratti di cellule in una provetta e conservare a -80 ° C fino al momento dell'analisi spettrometria di massa.
- cellule di coltura in una piastra da 6 pozzetti in triplicato in isotopi etichettato terreni di coltura stabile + supplementi dializzati (siero o altri integratori di crescita) fino a quando le cellule diventano il 75% confluenti.
- Estrazione di cellule ordinati finti
- cellule di coltura in un piatto 100 millimetri in triplice copia in isotopi etichettato terreni di coltura + supplementi dializzati stabili.
NOTA: Qui coltura cellule HeLa per 48 h in piatto 100 millimetri perché un elevato numero di celle (~ 4 x 10 6) sono necessarie per ottenere 500.000 cellule ordinati. Questo numero di HeLa estratto cellule è stato richiesto per ottenere una buona misura di metaboliti. I terreni di coltura utilizzato era RPMI contenente 40% di U- 13 C-glucosio e il 70% U-13 C, 15 N2-glutammina e il 5% dializzato FBS. - Al giorno d'estrazione, terreni di coltura scarti, lavare i pozzi wesimo 1.5 mL caldo HBSS e poi disfarsene.
- Staccare cellule aggiungendo 1,5 ml di tripsina / EDTA per 4 min a 37 ° C. Effettuare le seguenti operazioni a 4 ° C o nel ghiaccio.
- Disattivare tripsina con l'aggiunta di 3 ml di ghiaccio HBSS freddo (soluzione salina bilanciata di Hank) + supplemento dializzato.
- Raccogliere cellule in una provetta da centrifuga da 15 ml e 750 xg per 3 min.
- Risospendere il pellet in HBSS + dializzata supplemento + 1 mM EDTA ad una concentrazione di 1-2 x 10 6 cellule / ml, passano attraverso 40 micron filtri cellulari per ottenere cellule singole, e trasferire in una provetta da 5 ml.
- cellule ordinare attraverso cell sorter gating solo per canottiere, e le cellule centrifugare ordinato a 750 xg per 3 min a 4 ° C.
NOTA: qui Trova cellule HeLa a una velocità di 1.000 eventi / s, con la pressione dello strumento 27 psi, e l'utilizzo di un ugello 100 micron. cellule HeLa sono grandi quindi dovrebbero essere ordinati ad un tasso di eventi lento e grande bocchetta per ottenere pellet più intatto possibile. Mantenere le cellule in blocchi freddi throughout ordinamento per diminuire il metabolismo. Una descrizione approfondita della procedura di selezione è descritto in precedenza 12. - Eliminare il surnatante e risospendere il pellet in 50 microlitri di ghiaccio dH freddo 2 O per ottenere un pellet omogeneo prima di aggiungere metanolo. L'aggiunta di metanolo freddo direttamente al pellet forma un pellet solido che è difficile da risospendere.
- Estrarre metaboliti aggiungendo 540 ml di metanolo mantenuto in ghiaccio secco e mantenere estratti a -80 ° C fino cromatografia liquida - spettrometria di massa ad alta risoluzione (LC-HRMS) analisi.
- cellule di coltura in un piatto 100 millimetri in triplice copia in isotopi etichettato terreni di coltura + supplementi dializzati stabili.
- Estrazione del ciclo cellulare allineati cellule
- cellule di coltura in un piatto 100 millimetri in triplice copia in cultura dei media + supplementi dializzati.
NOTA: Qui cellule HeLa coltura contenenti Geminin Fucci verde (MAG1-hGem) sonda 13 che consente per l'ordinamento di G1 (negativo) e le cellule SG2M (positive). Le celle possono essere coltivate in un supporto stabile isotopi traccia per uns tempo necessario. Qui li abbiamo in coltura per 46 ore in supporti senza etichetta, allora abbiamo passato a RPMI contenente 40% di U- 13 C-glucosio e il 70% U-13 C, 15 N2-glutammina 2 ore prima di iniziare l'ordinamento. Ciò è stato fatto in modo da essere in grado di studiare le fasi del ciclo cellulare che richiede un breve impulso di etichettatura. - Al giorno d'estrazione, terreni di coltura scarti, lavare i pozzetti con 1,5 ml caldo HBSS e poi disfarsene.
- Staccare cellule aggiungendo 1,5 ml di tripsina / EDTA per 4 min a 37 ° C. Effettuare le seguenti operazioni a 4 ° C o nel ghiaccio.
- Disattivare tripsina con l'aggiunta di 3 ml di ghiaccio freddo HBSS + supplemento dializzato.
- Raccogliere cellule in una provetta da centrifuga da 15 ml e 750 xg per 3 min.
- Risospendere il pellet in HBSS + dializzata supplemento + 1 mM EDTA ad una concentrazione di 1-2 x 10 6 cellule / ml, passano attraverso 40 micron filtri cellulari per ottenere cellule singole, e trasferire in una provetta da 5 ml.
- cellule ordinare attraverso il cell sorter gDELL'INDICE DI i detriti e doppietti, quindi gating per il marcatore di cellule; e centrifugare le cellule filtrate a 750 xg per 3 min a 4 ° C.
NOTA: qui Trova cellule HeLa a una velocità di 1.000 eventi / s, con la pressione dello strumento 27 psi, e l'utilizzo di un ugello 100 micron. Mantenere le cellule in blocchi fredde tutto l'ordinamento per diminuire il tasso di metabolismo. - Eliminare il surnatante e risospendere il pellet in 50 microlitri di ghiaccio dH freddo 2 O per ottenere un pellet omogeneo prima di aggiungere metanolo. L'aggiunta di metanolo freddo direttamente al pellet forma un pellet solido che è difficile da risospendere.
- Estrarre i metaboliti aggiungendo 540 ml di metanolo mantenuto in ghiaccio secco e mantenere estratti a -80 ° C fino all'analisi LC-HRMS.
- cellule di coltura in un piatto 100 millimetri in triplice copia in cultura dei media + supplementi dializzati.
Analisi 2. Spettrometria di Massa
Nota: Qui si descrive il protocollo per l'analisi di estratti cellulari su un sistema LC-HRMS. Qualsiasi metodo metabolomica per analisi di estratti cellulari possono essere usati. Scansione completaanalisi potrebbe essere utile per rilevare una vasta gamma di metaboliti.
- Calibrare lo strumento utilizzando un mix di calibrazione di riferimento spettrometria di massa.
- estratti cellulari Scongelare in ghiaccio per 30 minuti e vortex per 15 s.
- Trasferire 100 microlitri dell'estratto cellulare ad un filtro rotativo e centrifugare per 10 minuti a 13.000 xga 4 ° C.
- Iniettare 12,5 ml di filtrato nel sistema LC-HRMS.
- metaboliti separate utilizzando una zwitterionici interazione idrofilica cromatografia liquida (ZIC-HILIC) della colonna (150 mm x 4.6 mm 5 micron dimensione delle particelle) dotati di una colonna di guardia ZIC-HILIC (20 mm × 2,1 millimetri) con un gradiente di eluizione dello 0,1% formico L'acido in acqua (a solvente) e acetonitrile (solvente B). Avviare il gradiente di eluizione a 20% di solvente A e aumentare fino al 80% in 17 min. Mantenere questa percentuale durante 4 minuti con un flusso di 400 ml min -1 e temperatura della colonna e vassoio di campione a 23 ° C e 4 ° C, rispettivamente.
- Utilizzare un iNstrument accoppiato alla separazione cromatografica per il rilevamento metaboliti, un elettrospray riscaldata (H-ESI II) sia in modalità positivi e negativi come sorgente di ionizzazione, e una modalità di acquisizione scansione completa ad una massa risolvere potenza di 70.000 Figura Larghezza tempo massimo (FWHM) ( m / z 200).
- Utilizzare azoto (purezza> 99,995%) per il gas guaina e gas ausiliario ad una portata di 45 e 10 au (unità arbitrarie) e temperatura vaporizzatore impostata a 350 ° C e la tensione electrospray a 4 kV in modalità positiva e -3.5 kV in modalità negativa.
Analisi 3. Dati
- Selezionare un numero di metaboliti per i quali sono disponibili gli standard e che mostrano picchi di qualità di campioni. picchi di buona qualità hanno un elevato rapporto segnale-rumore. E 'importante verificare la qualità di picco e assicurarsi di non includere falsi isotopi. picchi isotopici che differiscono in forma e / o tempo di ritenzione sono probabilmente false.
- Per i campioni marcati con isotopi calcolano isotopomero di massa (MI) frazioni dividendo l'area del picco di ogni MI con superficie totale di picco di tutti i MIS.
- Calcolare le frazioni di carbonio / azoto etichettati, arricchimento del 13 C e 15 N, rispettivamente, come
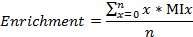
dove n è il numero totale di atomi di carbonio (o atomi di azoto, rispettivamente) nel metabolita, e Mix è la frazione MI di x.
NOTA: Tutti i calcoli possono essere eseguiti utilizzando linguaggi di programmazione.
Risultati
Come esempio, qui si descrive un esperimento indagare il metabolismo delle cellule HeLa selezionati secondo fase del ciclo cellulare. Per etichettare una vasta gamma di metaboliti centrali su entrambi i carboni e atomi di azoto, abbiamo coltivato le cellule per 48 h utilizzando U- 13 C-glucosio e U- 13 C, 15 N-glutammina come traccianti. Per ottenere MID ricche per l'esperimento di validazione, abbiamo scelto qui una miscela di 40% U- 13 C-...
Discussione
Il nostro metodo si basa sul principio che MIDs in metaboliti cellulari riflettono la "storia" delle attività metaboliche di una cella. Questo rende possibile indagare attività metaboliche in sottopopolazione di cellule, come si è verificato nel complesso comunità di cellule, prima della procedura di cell sorting. Al contrario, le aree di picco di metaboliti differiscono notevolmente tra estratti di cellule ordinati e estrazione diretta dal piatto della cultura 11. In parte ciò è ...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
Materiali
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
Riferimenti
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon