Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein Verfahren zur Messung des Metabolismus in sortierter Subpopulationen von komplexen Zell Gemeinschaften Verwendung stabiler Isotopen-Tracing
In diesem Artikel
Zusammenfassung
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
Zusammenfassung
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
Einleitung
Höhere Organismen enthalten komplexe Gemeinschaften von verschiedenen Zelltypen, die über komplexere Funktionen zu bringen, zusammenzuarbeiten. Zum Beispiel enthalten Tumoren nicht nur Krebszellen, sondern auch Fibroblasten, Zellen , die Blutgefäße darstellen, und oft Immunzellen infiltriert 1; Blut enthält eine komplexe Mischung von Dutzenden von Immunzellsubtypen 2; und auch kultivierte Zelllinien können aus mehreren Subpopulationen bestehen, wie beispielsweise der luminalen und basalen Subtypen von Brustkrebszellen 3. Darüber hinaus verschiedene Zelltypen, die metabolische "Zusammenarbeit" zeigen nebeneinander bestehen können. Beispielsweise im Gehirn, sind Astrozyten gedacht Glukose zu Laktat zu konvertieren, die "gefüttert" zu Neuronen ist dann , dass dieses Substrat 4 zu oxidieren; T - Lymphozyten sind in einigen Zusammenhängen abhängig von benachbarten dendritischen Zellen als eine Quelle für Cystein 5; und Krebszellen können mit asso zusammenarbeitengehörigen Fibroblasten in Tumoren 6. Um das metabolische Verhalten solcher Systeme zu verstehen, ist es wichtig, zu trennen und die Stoffwechselaktivitäten der verschiedenen Zelltypen zu messen.
Bei weitem das am häufigsten verwendete Verfahren zur Abtrennung von Zelltypen ist die Fluoreszenz-aktivierte Zellsortierung. Dieses Verfahren ist breit anwendbar, vorausgesetzt, dass der Zelltyp oder Zustand von Interesse kann unter Verwendung von fluoreszierenden Antikörpern, die Expression von gentechnisch fluoreszierende Proteine oder andere Farbstoffe "markiert" werden. Eine Möglichkeit besteht darin , zunächst getrennte Zelltypen durch einen Zellsortierer, wieder Kultur die einzelnen Zelltypen erhalten, und dann Metabolismus - Untersuchungen dieser Kulturen 7 durchführen. Dies ist jedoch nur möglich, wenn der Zelltyp oder Phänotyps in Kulturbedingungen stabil ist, und nicht die Übergangsverhalten, wie beispielsweise Zellzykluszustände noch die metabolischen Kooperation in Kokulturen erfassen kann. Für solche Fälle müssen Stoffwechsel direkt auf so gemessen werden,rtet Zellen. Das ist eine Herausforderung , da die Zelle Verfahren Sortierfächer Zellen zu Spannungen, die ihren Stoffwechsel 8 verzerren, und wir sind nur wenige Studien bewusst diesen Ansatz 9, 10. Insbesondere haben wir die Hauptmetaboliten wie Aminosäuren gefunden in Zellsortierpuffer gehalten von Zellen austreten kann, so dass die Messungen des absoluten Metabolitenfluss sind nicht mehr zuverlässig 11 (obwohl relativen Vergleich zwischen sortierten Fraktionen immer noch wertvoll sein kann).
Um diese Probleme zu umgehen, wir beschriften Zellen mit stabilen Isotopen vor der Sortierung und konzentrieren sich auf die MIDs in zellulären Metaboliten, anstatt Metaboliten Abundanzen. Da MIDs über längere Zeitskalen gebildet werden, sollten sie weniger durch Sortierung Bedingungen kurzfristige Exposition. Wir quantifizieren MIDs Full-Scan-hochauflösende Massenspektrometrie, die empfindlich genug ist, da bietenta auf Hunderten von Metaboliten von rund 500.000 sortierten Zellen beginnen, was etwa 30-60 Minuten von Zellsortierung Zeit. Ein Vergleich zwischen einem "mock sortiert" Kontrolle (Zellen durch den Zellsortierer Instrument weitergegeben, ohne spezifische Bevölkerungs Gating) und Metaboliten Extraktion direkt aus der Kulturschale gewährleisten gemacht, dass die beobachteten MIDs sind repräsentativ für diejenigen, die in der ursprünglichen Kultur. Je nach Wahl der stabilen Isotopen Tracern können verschiedene Stoffwechselwege mit diesem Verfahren untersucht werden.
Protokoll
1. Metabolit Extraction
- Extraktion aus Gericht
- Kultur-Zellen in einer 6-Well-Platte in dreifacher Ausführung in einem stabilen Isotop Kulturmedien + dialysiert Ergänzungen (Serum oder anderen Wachstums Ergänzungen), bis die Zellen werden 75% konfluent.
HINWEIS: Hier Kultur HeLa - Zellen für 48 h in RPMI mit 40% U- 13 C-Glucose und 70% U- 13 C, 15 N2-Glutamin und 5% dialysiertem FBS (Fetal Bovine Serum). Dialysierter FBS wird verwendet, der mit geringem Molekulargewicht Metaboliten, um loszuwerden, die den markierten Medien kontaminieren könnten. Kultivieren von Zellen in dialysiert Ergänzung vor dem realen Experiment wird empfohlen Zellen wachsen üblicherweise in dem Medium zu gewährleisten. Zuschläge sind in 0,15 M NaCl-Lösung über Nacht unter Verwendung von Schlangenhaut Dialyseschlauch dialysiert. - Am Tag der Kulturmedien Extraktion zu verwerfen, spülen Vertiefungen zweimal mit 500 & mgr; l kaltem HBSS und es dann zu verwerfen.
Hinweis: Verwenden Sie hier HBSS 40% U-13 C-Glucose , da es auch in den Kulturmedien. - In 600 & mgr; l 100% Methanol vorgekühlt auf Trockeneis.
- Übertragen Sie die Schüssel Eis zu trocknen und Zellmaterial mit einem Zellschaber entfernen.
- Sorgfältig Pipette die Zellextrakte auf einem Mikrozentrifugenröhrchen und bei -80 ° C bis zur Massenspektrometrie-Analyse.
- Kultur-Zellen in einer 6-Well-Platte in dreifacher Ausführung in einem stabilen Isotop Kulturmedien + dialysiert Ergänzungen (Serum oder anderen Wachstums Ergänzungen), bis die Zellen werden 75% konfluent.
- Die Extraktion von Mock sortierten Zellen
- Kultur-Zellen in einer 100-mm-Schale in dreifacher Ausführung in einem stabilen Isotop Kulturmedien + dialysierten ergänzt.
HINWEIS: Hier Kultur HeLa - Zellen für 48 h in 100 - mm - Schale , da eine hohe Anzahl von Zellen (~ 4 × 10 6) 500.000 sortierten Zellen zu erhalten , benötigt. Diese Anzahl von Zellen, HeLa-Extrakt wurde erforderlich gute Messung von Stoffwechselprodukten zu erhalten. Das Kulturmedium verwendet wurde , war RPMI , enthaltend 40% U- 13 C-Glucose und 70% U- 13 C, 15 N2-Glutamin und 5% dialysiertem FBS. - Am Tag der Extraktion, Discard Kulturmedien, spülen Brunnen with 1,5 ml warmem HBSS und es dann zu verwerfen.
- Abzulösen Zellen durch Zugabe von 1,5 ml Trypsin / EDTA für 4 min bei 37 ° C. Führen Sie die folgenden Schritte in 4 ° C oder in Eis.
- Deaktivieren Trypsin durch Zugabe von 3 ml eiskaltem HBSS (Hanks Balanced Salt Solution) + dialysiert Ergänzung.
- Sammeln Zellen in einem 15 ml-Röhrchen und zentrifugiert bei 750 × g für 3 min.
- Das Pellet in HBSS + dialysiert Ergänzung + 1 mM EDTA in einer Konzentration von 1 bis 2 x 10 6 Zellen / ml, durch 40 & mgr; m Zelle Siebe passieren zu einzelnen Zellen zu erhalten, und in ein 5 ml - Röhrchen.
- Sortier Zellen durch Gating Zellsortierer nur für Singuletts und zentrifugieren sortierten Zellen bei 750 × g für 3 min bei 4 ° C.
HINWEIS: Hier sortieren HeLa-Zellen mit einer Rate 1.000 Veranstaltungen / s, mit Instrumentendruck 27 psi und unter Verwendung einer 100 & mgr; m Düse. HeLa-Zellen sind groß, so sollten sie in einem langsamen Ereignisrate und große Düse sortiert werden als intakte Pellet wie möglich zu erhalten. Halten Sie die Zellen in kalten Blöcke throughout Sortieren des Metabolismus zu verringern. Eine ausführliche Beschreibung der Sortierverfahren wird zuvor 12 beschrieben. - Überstand verwerfen und das Pellet in 50 & mgr; l eiskaltem dH 2 O , um eine homogene Pellets zu erhalten , bevor Zugabe von Methanol. Zugabe von kaltem Methanol direkt zu Pellets bildet eine feste Pellet, das schwer zu resuspendieren.
- Extrahieren Sie Metaboliten durch Zugabe von 540 & mgr; l Methanol in Trockeneis gehalten und halten Extrakte bei -80 ° C bis zur Flüssigchromatographie - hochauflösender Massenspektrometrie (LC-HRMS) Analyse.
- Kultur-Zellen in einer 100-mm-Schale in dreifacher Ausführung in einem stabilen Isotop Kulturmedien + dialysierten ergänzt.
- Die Extraktion von Zellzyklus - sortierten Zellen
- Kultur-Zellen in einer 100-mm-Schale in dreifacher Ausführung in Kulturmedien + dialysiert Ergänzungen.
HINWEIS: Hier Kultur HeLa - Zellen Geminin Fucci Green (mag1-hGem) Sonde 13 , die für die Sortierung von G1 (negativ) ermöglicht und SG2M (positive) Zellen enthält. Die Zellen können in einem stabilen Isotop Tracing Medien für eine kultiviert werdens lange wie erforderlich. Hier haben wir kultiviert sie für 46 h in unmarkierten Medien, dann haben wir umgeschaltet RPMI mit 40% U- 13 C-Glucose und 70% U- 13 C, 15 N 2-Glutamin 2 h vor dem Start zu sortieren. Dies wurde getan, um in der Lage sein, die Zellzyklusphasen zu untersuchen, die eine kurze Pulsmarkierung erfordert. - Am Tag der Extraktion, Discard Kulturmedien, spülen Brunnen mit 1,5 ml warmem HBSS und es dann zu verwerfen.
- Abzulösen Zellen durch Zugabe von 1,5 ml Trypsin / EDTA für 4 min bei 37 ° C. Führen Sie die folgenden Schritte in 4 ° C oder in Eis.
- Deaktivieren Trypsin durch Zugabe von 3 ml eiskaltem HBSS + dialysierten ergänzen.
- Sammeln Zellen in einem 15 ml-Röhrchen und zentrifugiert bei 750 × g für 3 min.
- Das Pellet in HBSS + dialysiert Ergänzung + 1 mM EDTA in einer Konzentration von 1 bis 2 x 10 6 Zellen / ml, durch 40 & mgr; m Zelle Siebe passieren zu einzelnen Zellen zu erhalten, und in ein 5 ml - Röhrchen.
- Sortier Zellen durch den Zellsortierer gtriebs aus Schutt und Dubletts, dann für die Zellmarker von Interesse Gating und zentrifugieren sortierten Zellen bei 750 × g für 3 min bei 4 ° C.
HINWEIS: Hier sortieren HeLa-Zellen mit einer Rate 1.000 Veranstaltungen / s, mit Instrumentendruck 27 psi und unter Verwendung einer 100 & mgr; m Düse. Halten Sie die Zellen in kalten Blöcke im gesamten Sortier die Stoffwechselrate zu verringern. - Überstand verwerfen und das Pellet in 50 & mgr; l eiskaltem dH 2 O , um eine homogene Pellets zu erhalten , bevor Zugabe von Methanol. Zugabe von kaltem Methanol direkt zu Pellets bildet eine feste Pellet, das schwer zu resuspendieren.
- Extrahieren Sie die Metaboliten durch Zugabe von 540 & mgr; l Methanol in Trockeneis gehalten und halten Extrakte bei -80 ° C bis LC-HRMS-Analyse.
- Kultur-Zellen in einer 100-mm-Schale in dreifacher Ausführung in Kulturmedien + dialysiert Ergänzungen.
2. Massenspektrometrie-Analyse
Hinweis: Hier haben wir das Protokoll zur Analyse von Zellextrakten auf einem LC-HRMS-System zu beschreiben. Jegliche metabolomics Verfahren zur Analyse von Zellextrakten verwendet werden. Kompletter SuchlaufAnalyse könnte zum Erfassen einer Vielzahl von Stoffwechselprodukten nützlich sein.
- Kalibrieren Sie das Gerät eine Massenspektrometrie Referenzkalibrierungsmischung verwendet wird.
- Thaw Zellextrakten auf Eis für 30 min und Vortex für 15 s.
- Transfer von 100 & mgr; l des Zellextraktes auf ein Spinfilter und Zentrifuge für 10 min bei 13000 × g bei 4 ° C.
- Injizieren 12,5 & mgr; l des Filtrats auf das LC-HRMS-System.
- Separate Metaboliten ein zwitterionischer hydrophiler Wechselwirkungen FLÜSSIGCHROMATOGRAPHIE (ZIC-HILIC) Säule (150 mm x 4,6 mm, 5 & mgr; m Partikelgröße) mit einem ZIC-HILIC-Schutzsäule (20 mm × 2,1 mm) unter Verwendung einer Gradientenelution von 0,1% Ameisensäure ausgestattet Säure in Wasser (Lösungsmittel A) und Acetonitril (Lösungsmittel B). Starten Sie die Gradientenelution bei 20% Lösungsmittel A und erhöhen in 17 min bis zu 80%. Aufrechterhaltung dieser Prozentsatz während 4 min mit einem Strom von 400 & mgr; l min -1 und die Säulentemperatur und die Probenschale bei 23 ° C und 4 ° C.
- Verwenden Sie ein instrument zur chromatographischen Trennung gekoppelt Metaboliten Detektion einer beheizten Elektrosprüh (H-ESI II) in sowohl positive als auch negative Betriebsarten als Ionisationsquelle und einen Scan Erfassungsmodus bei einem Massenauflösungsvermögen von 70.000 Full Width Half Maximum (FWHM) ( m / z 200).
- Verwenden Sie Stickstoff (Reinheit> 99,995%) für den Mantel Gas und Hilfsgas mit einer Strömungsgeschwindigkeit von 45 und 10 au (willkürliche Einheiten), und stellen Verdampfertemperatur bei 350 ° C und der Elektrospannung bei 4 kV im positiven Modus und -3,5 kV in negativen Modus.
3. Datenanalyse
- Wählen Sie eine Reihe von Metaboliten, für die Normen verfügbar sind und die zeigen eine gute Qualität Spitzen in den Proben. Gute Qualität Spitzen haben hohes Signal-Rausch-Verhältnis. Es ist wichtig, Spitzenqualität zu überprüfen und sicherstellen, dass keine falschen Isotope enthalten. Isotopen-Peaks, die in Form und / oder Retentionszeit unterscheiden, sind wahrscheinlich falsch.
- Für isotopenmarkierten Proben berechnen Masse Isotopomer (MI) Fraktionen, die durch den Spitzenbereich jedes MI mit Gesamtpeakflächen aller Infarkte zu teilen.
- Berechnen der markierten Kohlenstoff / Stickstoff - Fraktionen, Anreicherung von 13 C und 15 N, jeweils als
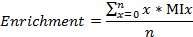
wobei n die Gesamtzahl der Kohlenstoffatome (oder Stickstoffe, respectively) in dem Metaboliten und mischen ist der MI Bruchteil x.
HINWEIS: Alle Berechnungen können mit Programmiersprachen durchgeführt werden.
Ergebnisse
Als Beispiel beschreiben wir ein Experiment den Metabolismus von HeLa-Zellen Untersuchung der Zellzyklus-Phase, sortiert nach. Um eine breite Palette von zentralen Metaboliten auf beiden Kohlenstoffe und Stickstoffe beschriften, kultiviert wir Zellen für 48 Stunden unter Verwendung von U- 13 C-Glucose und U- 13 C, 15 N-Glutamin als Tracer. Um reich MIDs für die Validierung Experiment zu erhalten, haben wir hier eine Mischung aus 40% U- 13 C-G...
Diskussion
Unsere Methode basiert auf dem Prinzip, dass MIDs in zellulären Metaboliten, die die "Geschichte" des metabolischen Aktivitäten einer Zelle widerspiegeln. Dies macht es möglich, Stoffwechselaktivitäten in Subpopulation von Zellen zu untersuchen, wie sie in der komplexen Gemeinschaft der Zellen erfolgte vor der Zellsortierung Verfahren. Im Gegensatz dazu unterscheiden sich die Peakflächen von Metaboliten deutlich zwischen Extrakten von sortierten Zellen und direkte Extraktion aus der Kulturschale
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
Materialien
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
Referenzen
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten