Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un método para medir el metabolismo en muestras ordenadas subpoblaciones de Comunidades celulares complejas utilizando isótopos estables de Búsquedas
En este artículo
Resumen
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
Resumen
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
Introducción
Los organismos superiores contienen comunidades complejas de distintos tipos de células que colaboran para llevar a cabo las funciones más complejas. Por ejemplo, los tumores contienen no sólo las células cancerosas, pero también fibroblastos, células que constituyen los vasos sanguíneos, y la célula inmune a menudo infiltra 1; sangre contiene una mezcla compleja de los subtipos de células inmunes 2 docenas; e incluso líneas de células cultivadas pueden consistir en múltiples subpoblaciones, como el luminal y subtipos basales de las células de cáncer de mama 3. Por otra parte, los tipos de células distintas que coexisten puede exhibir "colaboración" metabólica. Por ejemplo, en el cerebro, se cree que los astrocitos para convertir la glucosa a lactato, que es entonces "alimentado" a las neuronas que oxidan este sustrato 4; Los linfocitos T son en algunos contextos dependientes de células dendríticas adyacentes como fuente de cisteína 5; y las células cancerosas pueden colaborar con Assofibroblastos ciadas en los tumores 6. Para entender el comportamiento metabólico de tales sistemas, es esencial para separar y medir las actividades metabólicas de los diversos tipos de células presentes.
Con mucho, el método más ampliamente utilizado para la separación de tipos de células es la clasificación de células activadas por fluorescencia. Este método es ampliamente aplicable, siempre que el tipo de célula o estado de interés pueden ser "etiquetados" usando anticuerpos fluorescentes, la expresión de proteínas fluorescentes de ingeniería, u otros colorantes. Una opción es tipos de células inicialmente separadas a través de un clasificador de células, volver a la cultura de los tipos de células individuales obtenidas, y a continuación, realizar estudios de metabolismo de estos cultivos 7. Sin embargo, esto sólo es factible si el tipo de célula o fenotipo es estable en las condiciones de cultivo, y no pueden capturar comportamiento transitorio como los estados del ciclo celular, ni la cooperación metabólica en co-cultivos. Para tales casos, el metabolismo se debe medir directamente en lorted células. Este es un reto ya que el procedimiento de clasificación de células somete a las células a las tensiones que pueden distorsionar su metabolismo 8, y somos conscientes de sólo unos pocos estudios que toman este enfoque 9, 10. En particular, hemos encontrado que los principales metabolitos tales como aminoácidos puede gotear de las células mantenidas en un tampón de clasificación de células, por lo que las mediciones de la abundancia metabolito absoluta ya no son fiables 11 (aunque comparación relativa entre las fracciones clasificadas aún puede ser valioso).
Para evitar estos problemas, etiquetamos células con isótopos estables antes de la clasificación, y nos centramos en las MID en los metabolitos celulares, en lugar de las abundancias de metabolitos. Dado que los MID se forman durante periodos de tiempo más largos, deben ser menos afectados por la exposición a corto plazo a las condiciones de clasificación. Se cuantifica utilizando MID-análisis completo de espectrometría de masas de alta resolución, que es lo suficientemente sensible como para proporcionar data en cientos de metabolitos a partir de alrededor de 500.000 células clasificadas, que requieren aproximadamente 30-60 minutos de tiempo de clasificación de células. Una comparación entre un "mock" ordenadas de control (células pasan a través del instrumento clasificador de células sin gating cualquier población específica) y la extracción de metabolito directamente de se hace la placa de cultivo para asegurar que los MIDs observados son representativos de los de la cultura original. Dependiendo de la elección de los trazadores de isótopos estables, diversas vías metabólicas pueden ser estudiadas con este método.
Protocolo
1. Extracción de metabolitos
- Extracción del plato
- células de cultivo en una placa de 6 pocillos por triplicado en la etiqueta de isótopos estables medios de cultivo + suplementos dializados (suero u otros suplementos de crecimiento) hasta que las células se convierten en el 75% de confluencia.
NOTA: Aquí células HeLa cultivo durante 48 h en RPMI que contiene 40% de U 13 C-glucosa y 70% de U 13 C, 15 N2-glutamina y 5% de FBS dializado (Suero Bovino Fetal). FBS dializado se utiliza para deshacerse de los pequeños metabolitos de peso molecular que puedan contaminar los medios de etiquetado. Se recomienda células en suplemento de dializado antes del experimento real, el cultivo para asegurar células están creciendo normalmente en el medio. Los suplementos se dializan en solución 0,15 M de NaCl durante la noche utilizando tubos de diálisis de piel de serpiente. - En el día de la extracción de los medios de cultivo de descarte, enjuague pocillos dos veces con HBSS 500 l fría y luego descartarlo.
NOTA: Aquí utilizar HBSS que contiene 40% U-13 C-glucosa, ya que también se encuentra en el medio de cultivo. - Añadir 600 l 100% de metanol previamente enfriado en hielo seco.
- Transferir el plato a hielo seco y eliminar el material celular con un rascador de células.
- cuidadoso pipeteado los extractos de células a un tubo de microcentrífuga y se almacena a -80 ° C hasta el análisis de espectrometría de masas.
- células de cultivo en una placa de 6 pocillos por triplicado en la etiqueta de isótopos estables medios de cultivo + suplementos dializados (suero u otros suplementos de crecimiento) hasta que las células se convierten en el 75% de confluencia.
- La extracción de células clasificadas simulacros
- células de cultivo en una placa de 100 mm por triplicado en el marcado con un isótopo medios de cultivo + suplementos dializados estables.
NOTA: A continuación, las células HeLa cultivo durante 48 h en placas de 100 mm debido a un elevado número de células (~ 4 x 10 6) son necesarios para obtener 500.000 células clasificadas. Se requiere este número de células HeLa extracto para obtener una buena medición de los metabolitos. El medio de cultivo utilizado fue RPMI que contiene 40% de U 13 C-glucosa y 70% de U 13 C, 15 N2-Glutamina y 5% de FBS dializado. - En el día de la extracción, los medios de cultivo de descarte, enjuague pozos wITH 1,5 ml HBSS caliente y luego descartarlo.
- Separar las células mediante la adición de 1,5 ml de tripsina / EDTA durante 4 minutos a 37 ° C. Realice los siguientes pasos en 4 ° C o en hielo.
- Desactivar la tripsina mediante la adición de 3 ml de hielo frío HBSS (solución salina equilibrada de Hank) + suplemento de dializado.
- Recoger las células en un tubo de centrífuga de 15 ml y a 750 xg durante 3 min.
- Resuspender el precipitado en el suplemento EDTA dializada HBSS + + 1 mM a una concentración de 1-2 x 10 6 células / ml, pasan a través de tamices de 40 micras de células para obtener células individuales, y transferir a un tubo de 5 ml.
- Ordenar a través de las células compuerta de clasificación de células única para interiores, y centrifugar las células ordenados a 750 xg durante 3 min a 4 ° C.
NOTA: Aquí ordenar células HeLa a una velocidad de 1.000 eventos / s, con la presión del instrumento 27 psi, y el uso de una boquilla de 100 micras. Las células HeLa son grandes, así que deben ser ordenados a un ritmo lento y eventos boquilla grande para obtener pellets lo más intacta posible. Mantener las células en bloques de frío throughout clasificación para disminuir el metabolismo. Una descripción exhaustiva de procedimiento de clasificación se ha descrito previamente 12. - Desechar el sobrenadante y resuspender el precipitado en 50 l de hielo frío dH 2 O para obtener una pastilla homogénea antes de la adición de metanol. La adición de metanol frío directamente a pellet forma un sedimento sólido que es difícil de resuspender.
- Extraer los metabolitos mediante la adición de 540 l de metanol mantenido en hielo seco y mantener extractos a -80 ° C hasta que la cromatografía líquida - espectrometría de masas de alta resolución (LC-HRMS) análisis.
- células de cultivo en una placa de 100 mm por triplicado en el marcado con un isótopo medios de cultivo + suplementos dializados estables.
- Extracción de ciclo celular ordenada células
- células de cultivo en una placa de 100 mm por triplicado en medios de cultivo + suplementos dializados.
NOTA: A continuación, las células HeLa de cultivo que contenían geminina Fucci Verde (MAG1-hGem) de la sonda 13, que permite la clasificación de G1 (negativo) y células SG2M (positivos). Las células pueden cultivarse en medios de isótopos estables para el rastreo de unas largo como sea necesario. Aquí los hemos cultivado durante 46 h en medios sin etiqueta, entonces hemos cambiado a RPMI que contiene 40% U-13 C-glucosa y 70% de U-13 C, 15 N2-glutamina 2 h antes de iniciar la clasificación. Esto se hizo con el fin de poder estudiar las fases del ciclo celular que requiere un etiquetado de pulso corto. - En el día de la extracción, los medios de cultivo de descarte, enjuague pocillos con 1,5 ml de HBSS caliente y luego descartarlo.
- Separar las células mediante la adición de 1,5 ml de tripsina / EDTA durante 4 minutos a 37 ° C. Realice los siguientes pasos en 4 ° C o en hielo.
- Desactivar la tripsina mediante la adición de 3 ml de hielo frío HBSS + suplemento de dializado.
- Recoger las células en un tubo de centrífuga de 15 ml y a 750 xg durante 3 min.
- Resuspender el precipitado en el suplemento EDTA dializada HBSS + + 1 mM a una concentración de 1-2 x 10 6 células / ml, pasan a través de tamices de 40 micras de células para obtener células individuales, y transferir a un tubo de 5 ml.
- Ordenar las células a través del separador de células gAting los desechos y dobletes, entonces compuerta para el marcador de célula de interés, y centrifugar las células ordenados a 750 xg durante 3 min a 4 ° C.
NOTA: Aquí ordenar células HeLa a una velocidad de 1.000 eventos / s, con la presión del instrumento 27 psi, y el uso de una boquilla de 100 micras. Mantener las células en bloques de frío en toda la clasificación para disminuir la tasa de metabolismo. - Desechar el sobrenadante y resuspender el precipitado en 50 l de hielo frío dH 2 O para obtener una pastilla homogénea antes de la adición de metanol. La adición de metanol frío directamente a pellet forma un sedimento sólido que es difícil de resuspender.
- Extraer los metabolitos mediante la adición de 540 l de metanol mantenido en hielo seco y mantener extractos a -80 ° C hasta su análisis LC-HRMS.
- células de cultivo en una placa de 100 mm por triplicado en medios de cultivo + suplementos dializados.
Análisis 2. La espectrometría de masa
Nota: A continuación se describe el protocolo para el análisis de extractos de células en un sistema LC-HRMS. Los métodos para el análisis de la metabolómica de extractos celulares pueden ser utilizados. Análisis completoanálisis podría ser útil para detectar una amplia gama de metabolitos.
- Calibrar el instrumento utilizando una mezcla de masa de calibración de referencia espectrometría.
- extractos de células descongelación en hielo durante 30 minutos y agitar durante 15 s.
- Transferir 100 l de extracto celular a un filtro de giro y se centrifuga durante 10 min a 13.000 xg a 4 ° C.
- Inyectar 12.5 l del filtrado en el sistema de LC-HRMS.
- metabolitos separados utilizando una cromatografía líquida zwitteriónico interacción hidrofílica (ZIC-HILIC) columna (150 mm x 4,6 mm, 5 micras de tamaño de partícula) equipado con una columna de guardia ZIC-HILIC (20 mm x 2,1 mm) utilizando un gradiente de elución de 0,1% fórmico ácido en agua (disolvente a) y acetonitrilo (disolvente B). Iniciar el gradiente de elución en 20% de disolvente A y aumentar hasta un 80% en 17 min. Mantener este porcentaje durante 4 min con un flujo de 400 l min -1 y temperatura de la columna y la bandeja de la muestra a 23 ° C y 4 ° C, respectivamente.
- Utilice un iNSTRUMENTO acoplado a la separación cromatográfica para la detección de metabolitos, un electrospray calentado (H-ESI II) en ambos modos positivo y negativo como fuente de ionización, y un modo de adquisición de exploración completa en un poder de resolución de masa de 70.000 Ancho completo la mitad del máximo (FWHM) ( m / z 200).
- El uso del nitrógeno (pureza> 99,995%) para el gas envolvente y el gas auxiliar a una velocidad de flujo de 45 y 10 au (unidades arbitrarias) y establecer temperatura del vaporizador a 350 ° C y la tensión de electrospray en 4 kV en modo positivo y -3,5 kV en el modo negativo.
Análisis 3. Datos
- Seleccionar una serie de metabolitos para las que las normas están disponibles y que muestran picos de gran calidad de las muestras. picos de buena calidad tienen una alta relación señal-ruido. Es importante verificar la calidad máxima y asegúrese de no incluir isótopos falsas. picos isotópicos que difieren en forma y / o tiempo de retención es probable falsa.
- Para las muestras marcadas por isótopos calculan isotopomer masa (MI) fracciones dividiendo el área del pico de cada MI con áreas totales máximas de todos los infartos de miocardio.
- Calcular las fracciones de carbono / nitrógeno etiquetados, el enriquecimiento de 13 C y 15 N, respectivamente, como se
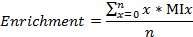
donde n es el número total de átomos de carbono (o nitrógenos, respectivamente) en el metabolito, y la mezcla es la fracción MI de x.
NOTA: Todos los cálculos se pueden realizar utilizando lenguajes de programación.
Resultados
A modo de ejemplo, aquí se describe un experimento que investiga el metabolismo de las células HeLa ordenados según la fase del ciclo celular. Para etiquetar una amplia gama de metabolitos centrales de ambos carbonos y nitrógenos, cultivamos las células durante 48 h utilizando U- 13 C-glucosa y U- 13 C, 15 N-glutamina como trazadores. Para obtener MIDs ricos para el experimento de validación, que aquí elegimos una mezcla de 40% de U 13 C...
Discusión
Nuestro método se basa en el principio de que los MID de metabolitos celulares reflejan la "historia" de las actividades metabólicas de una célula. Esto hace que sea posible para investigar las actividades metabólicas en subpoblación de células, ya que se produjeron en el complejo comunidad de células, antes del procedimiento de clasificación de células. Por el contrario, áreas de los picos de metabolitos difieren notablemente entre los extractos de células clasificadas y extracción directa de la p...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
Materiales
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
Referencias
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados