このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
安定同位体のトレースを使用した複雑な細胞のコミュニティのソートされた亜集団で代謝を測定する方法
要約
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
要約
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
概要
高等生物は、より複雑な機能をもたらすために協力異なる細胞型の複雑なコミュニティを含みます。例えば、腫瘍は、癌細胞だけでなく、線維芽細胞、血管を構成する細胞だけでなく、を含み、多くの場合、免疫細胞は、1浸潤します;血液は、免疫細胞サブタイプ2の数十の複雑な混合物が含まれています。さらには培養細胞株は、乳癌細胞3の管腔および基底サブタイプなどの複数の亜集団、から構成されてもよいです。また、共存する異なる細胞型は、代謝、「コラボレーション」を示すことができます。例えば、脳内で、アストロサイトは、この基板4を酸化ニューロンへのその後「摂食」である乳酸にグルコースを変換するために考えられています。 Tリンパ球は、システイン5の供給源として、隣接する樹状細胞に依存して、いくつかの状況です。そして癌細胞はASSOと協力することができます腫瘍6でciated線維芽細胞。このようなシステムの代謝挙動を理解するために、存在する種々の細胞型の代謝活動を分離して測定することが不可欠です。
はるか細胞型を分離するために最も広く使用される方法は、蛍光活性化細胞選別です。この方法は広く適用可能であり、目的の細胞型または状態は、蛍光抗体、操作された蛍光タンパク質の発現、又は他の染料を使用して、「標識された」ことができることを条件とします。 1つのオプションは、次にセルソーター、得られた再培養個々の細胞型、及び貫通最初は別個の細胞タイプは、これらの培養物7の代謝試験を行うことです。細胞型または表現型は、培養条件で安定であり、そのような細胞周期状態を、また、共培養における代謝協力として過渡的な挙動を捕捉することができない場合は、これは実現可能です。このような場合のために、代謝がそうで直接測定されなければなりませんRTED細胞。セルは手順をソートするので、これは困難である彼らの新陳代謝8を歪めることがストレスに細胞を施し、そして我々はこのアプローチ9、10を取るだけ少数の研究を認識しています。特に、我々は絶対的な代謝産物の存在量の測定は、もはや信頼性があるように、アミノ酸等の主要な代謝産物は、細胞ソーティングバッファに保持した細胞から漏れないことを見出した11(ソートされた画分の間の相対的な比較は依然として価値があるかもしれません)。
これらの問題を回避するために、我々は、ソートする前に、安定同位体で細胞を標識し、細胞代謝産物ではなく、代謝物の存在量でのMIDに焦点を当てます。 MIDは長い時間スケール上に形成されているので、それらは以下のソート条件に短期暴露の影響を受けなければなりません。我々は、DAを提供するのに十分な感度でフルスキャンの高分解能質量分析を使用してのMIDを定量化セルソーティング時間の約30〜60分を必要とする50万選別した細胞から始まる代謝物の何百も、上のta。培養皿から直接制御(細胞は、任意の特定の集団をゲーティングすることなく、セルソーター器を通過した)および代謝物の抽出を「モックがソート」との比較は観察のMIDは、元の培養中のものの代表であることを保証するために行われます。安定同位体トレーサーの選択に応じて、様々な代謝経路は、この方法で研究することができます。
プロトコル
1.代謝物の抽出
- 皿からの抽出
- 安定同位体標識された培養培地+透析サプリメント(血清または他の成長サプリメント)で三連で6ウェルプレートで培養細胞は、細胞が75%コンフルエントになるまで。
注:U- 13 C-グルコース40%を含むRPMI中で48時間と70%U- 13 C、15 N2-グルタミンおよび5%透析FBS(ウシ胎児血清)のためにここでは、培養HeLa細胞。透析FBSは、標識されたメディアを汚染する可能性がある低分子量代謝物を取り除くために使用されます。実際の実験の前に透析したサプリメントで細胞を培養する培地で細胞を正常に増殖していることを確認することをお勧めします。サプリメントは、ヘビ肌透析チューブを用いて、0.15 M NaCl溶液で一晩透析します。 - 抽出廃棄培地の日に、500μLの冷HBSSで二回、ウェルを洗浄し、それを捨てます。
注:ここでは40%を含むHBSSを使用U-13 C-グルコースは培地もあるからです。 - 追加600μLの100%メタノールをドライアイス上で予備冷却しました。
- ドライアイスに皿を転送し、セルスクレーパーで細胞材料を除去します。
- 注意深く質量分析まで-80℃でマイクロ遠心チューブとストアに細胞抽出物をピペット。
- 安定同位体標識された培養培地+透析サプリメント(血清または他の成長サプリメント)で三連で6ウェルプレートで培養細胞は、細胞が75%コンフルエントになるまで。
- モック選別細胞の抽出
- 安定同位体標識された培養培地+透析サプリメントで三重100 mmディッシュで培養細胞。
注:100ミリメートル皿で48時間ここで、培養HeLa細胞の細胞の数が多い(〜4×10 6)を 50万選別された細胞を得るために必要とされるからです。 HeLa細胞抽出物のこの数は、代謝物の良好な測定を得るために必要でした。培地は、40%U- 13 Cグルコース、70%U- 13 C、15 N 2グルタミン及び5%透析FBSを含むRPMIを用いました。 - 抽出、廃棄培地の日に、Wウェルをすすぎますi番目の1.5mLの温かいHBSSし、それを捨てます。
- 37℃で4分間、1.5 mLのトリプシン/ EDTAを加えることによって、細胞を切り離します。 4°Cまたは氷で次の手順を実行します。
- 3 mLの氷冷HBSS(ハンクス液)+透析サプリメントを追加することにより、トリプシンを無効にします。
- 3分間750×gで15 mLチューブと遠心分離機で細胞を収集します。
- 濃度1〜2×10 6細胞/ mLのHBSS +透析サプリメント+ 1mMのEDTAでペレットを再懸濁し、単一細胞を得るために40μmのセルストレーナーを通過し、5 mLのチューブに移します。
- ソートのみ一重のためのセルソーター・ゲーティングを介して細胞、および4℃で3分間、750×gで遠心分離選別細胞。
楽器圧力27 psiのと速度千のイベント/秒でここソートHeLa細胞、および100μmのノズルを使用します。注。 HeLa細胞は、それらが可能な限り無傷のペレットを得るために遅い事象率と大ノズルでソートされるべき大です。寒さのブロック内のセルを保つthroughou代謝を減少させるために選別T。手順ソートの完全な説明は、以前12に記載されています。 - 上清を捨て、メタノールを加える前に、均一なペレットを得るために、50μLの氷冷のdH 2 Oにペレットを再懸濁。直接ペレットへの冷メタノールの添加は、再懸濁しにくい固体ペレットを形成しています。
- ドライアイスに保た540μLのメタノールを添加することにより、代謝産物を抽出し、液体クロマトグラフィーするまで-80℃で抽出を維持 - 高分解能質量分析(LC-HRMS)分析。
- 安定同位体標識された培養培地+透析サプリメントで三重100 mmディッシュで培養細胞。
- 細胞周期選別された細胞の抽出
- 培養培地+透析サプリメントで三重100 mmディッシュで培養細胞。
注:GemininのFucciグリーンG1(負)とSG2M(陽性)細胞の選別を可能にします(MAG1-hGem)プローブ13を含む。ここで、培養したHeLa細胞。細胞は用の安定同位体のトレース培地で培養することができます必要に応じて長いですよ。ここでは、我々は40%U- 13 C-グルコースおよび70%U- 13 C、ソート開始する前に、15 N2-グルタミン2時間を含むRPMIに切り替えた、標識されていない培地で46時間のためにそれらを培養しています。これは、短いパルス標識を必要とする細胞周期段階を研究することができるようにするために行きました。 - 抽出、廃棄培地の日に、1.5 mLの温かいHBSSでウェルを洗浄し、それを捨てます。
- 37℃で4分間、1.5 mLのトリプシン/ EDTAを加えることによって、細胞を切り離します。 4°Cまたは氷で次の手順を実行します。
- 3 mLの氷冷HBSS +透析サプリメントを追加することにより、トリプシンを無効にします。
- 3分間750×gで15 mLチューブと遠心分離機で細胞を収集します。
- 濃度1〜2×10 6細胞/ mLのHBSS +透析サプリメント+ 1mMのEDTAでペレットを再懸濁し、単一細胞を得るために40μmのセルストレーナーを通過し、5 mLのチューブに移します。
- セルソーターグラムをソート細胞その後、破片やダブレットをating関心の細胞マーカーのためのゲーティング、および遠心選別細胞を4℃で3分間、750×gで。
楽器圧力27 psiのと速度千のイベント/秒でここソートHeLa細胞、および100μmのノズルを使用します。注。代謝率を減少させるためにソートを通して冷たいブロック内のセルを保管してください。 - 上清を捨て、メタノールを加える前に、均一なペレットを得るために、50μLの氷冷のdH 2 Oにペレットを再懸濁。直接ペレットへの冷メタノールの添加は、再懸濁しにくい固体ペレットを形成しています。
- ドライアイスに保た540μLのメタノールを加えて代謝物を抽出し、LC-HRMS分析まで-80℃で抽出物を保持します。
- 培養培地+透析サプリメントで三重100 mmディッシュで培養細胞。
2.質量分析
注:ここでは、LC-HRMSシステム上の細胞抽出物を分析するためのプロトコルについて説明します。細胞抽出物の分析のための任意のメタボロミクスの方法を用いることができます。フルスキャン分析は、代謝物の広い範囲を検出するのに有用である可能性があります。
- 質量分析基準較正ミックスを使用して、機器のキャリブレーションを行います。
- 15秒間30分間ボルテックス氷上で細胞抽出物を解凍します。
- 4℃で13,000×gで10分間スピンフィルターや遠心分離機に細胞抽出物の100μLを転送します。
- LC-HRMSシステムにろ液の12.5μLを注入します。
- 0.1%のギ酸の勾配溶出を使用して、ZIC-HILICガードカラム(2.1ミリメートル×20ミリメートル)を装着した双性イオン性親水性相互作用液体クロマトグラフィー(ZIC-HILIC)カラム(150ミリメートル×4.6ミリメートル、5μmの粒子サイズ)を使用して、個別の代謝物水中での酸(溶媒A)およびアセトニトリル(溶媒B)。溶剤Aの20%で勾配溶出を開始し、17分で80%まで増加。それぞれ、4 400μL分の流速で分-1および23℃と4℃のカラム温度とサンプルトレイ中にこの割合を維持します。
- 私を使用してください代謝物の検出、イオン化源として正と負の両方のモードで加熱エレクトロスプレー(H-ESI II)、70,000半値幅(FWHM)の質量分解能でフルスキャン取得モードのためのクロマトグラフィー分離に結合nstrument( M / Z 200)。
- 45と10のauの流量(任意単位)と設定し、350℃での気化温度とポジティブモードと-3.5 kVの4 kVのでエレクトロスプレー電圧でシースガスおよび補助ガスを窒素(純度> 99.995パーセント)を使用しますネガティブモードインチ
3.データ解析
- 規格が利用可能であり、試料中の良質なピークを示しているために代謝産物の数を選択します。良質のピークは、高い信号対雑音比を有しています。ピークの品質を検証し、偽の同位体を含まないことを確認することが重要です。形状および/または保持時間が異なる同位体ピークはおそらくfalseです。
- 同位体標識されたサンプルについて(質量アイソトポマーを計算MI)すべてのMIの総ピーク面積と各MIのピーク面積を分割することによって画分。
- それぞれ、13 Cおよび15 Nの濃縮をラベルされた炭素/窒素画分を計算します
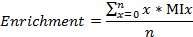
ここで、nは、代謝物中の炭素の総数(または窒素、それぞれ)は、ミックスは、xのMI分率です。
注:すべての計算は、プログラミング言語を用いて行うことができます。
結果
一例として、ここでは、細胞周期の段階に応じてソートされたHeLa細胞の代謝を調べる実験を記載します。炭素と窒素の両方に中央代謝物の広い範囲を標識するために、我々はU- 13 C-グルコースおよびトレーサーとしてU- 13 C、15 N-グルタミンを使用して48時間、細胞を培養しました。同位体の中間レベルはより多様MIパターン14を
ディスカッション
我々の方法は、細胞代謝産物中のMIDは、細胞の代謝活性の「歴史」を反映している原理に基づいています。これは、細胞の複雑なコミュニティで生じたような従来の細胞選別手順と、細胞の亜集団における代謝活性を調査することができます。これとは対照的に、代謝物のピーク面積は、ソートされた細胞と培養皿11から直接抽出の抽出物との間で著しく異なります。部分?...
開示事項
著者らは、開示することは何もありません。
謝辞
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
資料
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
参考文献
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved