需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
一种用于在复杂的细胞群落稳定性同位素示踪排序亚群测量方法代谢
摘要
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
摘要
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
引言
高等生物包含协作带来更复杂的功能不同的细胞类型的复杂的社区。例如,肿瘤不仅含有癌细胞,而且成纤维细胞,在构成血管,并经常免疫细胞浸润1细胞;血液中含有数十免疫细胞亚型2的复杂混合物;甚至培养的细胞系可以由多个亚群,如管腔和乳腺癌细胞3的基底亚型。此外,不同的细胞类型共存能够表现出代谢"合作"。例如,在大脑中,星形胶质细胞被认为是葡萄糖转化为乳酸,然后将其"喂"到氧化该衬底4的神经元; T淋巴细胞是在一些依赖于相邻的树突细胞作为半胱氨酸5的源上下文;和癌细胞可能与阿索协作肿瘤6 ciated成纤维细胞。为了理解这种系统的代谢行为,至关重要的是,以分离和测量存在于各种细胞类型的代谢活动。
迄今为止用于分离细胞类型的最常用的方法是荧光激活细胞分选。此方法是广泛适用的,只要该细胞类型或感兴趣的状态可以"标记"使用荧光抗体,工程化的荧光蛋白的表达,或其他的染料。一种选择是最初分离的细胞类型通过细胞分选仪,再培养得到的个别细胞类型,然后执行这些培养7的代谢研究。然而,如果细胞类型或表型是在培养条件下稳定的,并且不能捕获瞬态行为,如细胞周期状态,也不会在共培养物的代谢合作,这是唯一可行的。对于这样的情况下,代谢必须直接上,以便测量rted细胞。这是具有挑战性的,因为细胞分选过程科目细胞可能扭曲他们的新陈代谢8的压力,而我们都知道只有少数的研究采用这种方法9,10。特别是,我们已经发现,主要代谢产物如氨基酸可以从保持在细胞分拣缓冲液细胞泄漏,使绝对代谢物丰度的测量结果不再可靠11(虽然排序级分之间的相对比较,仍可能是有价值的)。
为了规避这些问题,我们的标签细胞之前,分拣稳定同位素,并专注于细胞代谢产物,而不是代谢产物丰度的MID产品。由于MID产品则经过较长时间尺度上形成的,就应该少受短期接触到排序的条件。我们用量化的全扫描高分辨率质谱,这是非常敏感地提供移动互联网设备达TA数以百计的代谢物从约50选细胞出发,大约需要30-60分钟的细胞分选的时间。一个之间的比较"模拟排序"控制(通过细胞分选仪仪器没有选通任何特定人口传递细胞)和代谢物提取直接从培养皿制成,以确保所观察到的MID的是代表那些在原始培养物。取决于稳定同位素示踪剂的选择,各种代谢途径可以用这种方法进行研究。
研究方案
1.提取代谢物
- 从盘中提取
- 在稳定同位素标记的培养基+透析补充剂(血清或其他生长补充剂)一式三份一6孔板培养细胞直至细胞成为75%汇合。
注意:在此培养HeLa细胞在RPMI 48小时含有40%U- 13 C-葡萄糖和70%U- 13 C,15 N 2 -谷氨酰胺和5%透析的FBS(胎牛血清)。透析FBS用来摆脱可能污染标记的媒体小分子量代谢产物。培养在之前的实际实验透析补充细胞建议确保细胞在培养基中正常生长。补充剂在0.15M NaCl的溶液透析使用蛇皮透析管过夜。 - 在萃取丢弃培养基的当天,漂洗孔两次用500微升冷的HBSS,然后丢弃它。
注意:这里使用含40%HBSS U-13 C-葡萄糖,因为它也是在培养基中。 - 添加600μL的100%甲醇中预冷干冰上。
- 传送盘干冰和除去细胞材料用细胞刮刀。
- 仔细吸取细胞提取物到微量离心管中并储存在-80℃直至质谱分析。
- 在稳定同位素标记的培养基+透析补充剂(血清或其他生长补充剂)一式三份一6孔板培养细胞直至细胞成为75%汇合。
- 模拟排序细胞的提取
- 培养细胞在100毫米的菜一式三份的稳定同位素标记的培养基+透析补充剂。
注:这里的文化HeLa细胞在100mm皿48小时,因为(〜4×10 6)需要的细胞大量获得500000排序细胞。被要求HeLa细胞提取物这个数目,以获得代谢产物的良好的测量。所用的培养基是RPMI含40%U- 13 C葡萄糖和70%U- 13 C,15 N2谷氨酰胺和5%FBS透析。 - 在萃取后,弃去培养基的一天,冲洗井瓦特第i个1.5毫升温HBSS,然后丢弃它。
- 通过在37℃下加入1.5毫升胰蛋白酶/ EDTA 4分钟分离细胞。执行在4℃或在冰以下步骤。
- 通过加入3毫升冰冷的HBSS(Hank氏平衡盐溶液)+透析补充停用胰蛋白酶。
- 收集在15毫升管中并离心细胞,在750×g离心3分钟。
- 悬浮在HBSS +透析补充+ 1mM EDTA中沉淀的浓度1-2×10 6个细胞/ mL,通过40微米的细胞滤网,以获得单细胞,并转移到一个5毫升管中。
- 仅用于汗衫排序细胞通过细胞分选仪门控,并在750×g离心在4℃下3分钟的离心分选的细胞。
注:此处的排序HeLa细胞的速率1,000个事件/秒,用仪表压力27磅,并用100微米的喷嘴。 HeLa细胞是大所以他们应该在一个缓慢的事件发生率和大喷嘴进行排序,以获得尽可能完整的颗粒越好。保持所述细胞在冷块throughouŧ排序,以降低新陈代谢。排序过程的全面描述以前12中所述。 - 弃上清,悬浮颗粒在50微升冰冷的dh 2 O加入甲醇之前获得均匀的颗粒。冷甲醇除了直接沉淀形成固体沉淀,这是很难再悬浮。
- 通过加入保持在干冰540微升甲醇提取代谢物,并在-80保持萃取℃直至液相色谱 - 高分辨质谱(LC-HRMS)分析。
- 培养细胞在100毫米的菜一式三份的稳定同位素标记的培养基+透析补充剂。
- 细胞周期的提取分选的细胞
- 培养细胞在文化传媒+透析一式三份补充100毫米菜。
注:含有Geminin Fucci绿色(MAG1-hGem)探测器13,它允许为G1(负)和SG2M(阳性)细胞的分选这里培养HeLa细胞。细胞可在一个稳定的同位素示踪培养基中培养悠久的要求。在这里,我们培养他们在未标记的媒体46小时,然后我们切换到含40%U- 13 C葡萄糖和70%U- 13 C RPMI,前15 N2谷氨酰胺2小时开始排序。这是为了能够研究这需要一个短脉冲标记细胞周期阶段完成。 - 在萃取后,弃去培养基的一天,冲洗用1.5ml温暖的HBSS孔中,然后将其丢弃。
- 通过在37℃下加入1.5毫升胰蛋白酶/ EDTA 4分钟分离细胞。执行在4℃或在冰以下步骤。
- 通过加入3毫升冰冷的HBSS +透析的补充停用胰蛋白酶。
- 收集在15毫升管中并离心细胞,在750×g离心3分钟。
- 悬浮在HBSS +透析补充+ 1mM EDTA中沉淀的浓度1-2×10 6个细胞/ mL,通过40微米的细胞滤网,以获得单细胞,并转移到一个5毫升管中。
- 排序细胞通过细胞分选仪克阿婷出碎屑和双峰,然后选通为感兴趣的细胞标记物,并离心分选的细胞在750×g离心在4℃下3分钟。
注:此处的排序HeLa细胞的速率1,000个事件/秒,用仪表压力27磅,并用100微米的喷嘴。保持在冷块中的细胞在整个排序以降低代谢速率。 - 弃上清,悬浮颗粒在50微升冰冷的dh 2 O加入甲醇之前获得均匀的颗粒。冷甲醇除了直接沉淀形成固体沉淀,这是很难再悬浮。
- 通过加入保持在干冰540微升甲醇提取代谢物,并在-80℃保持提取直到LC-HRMS的分析。
- 培养细胞在文化传媒+透析一式三份补充100毫米菜。
2.质谱分析
注:在这里,我们描述了协议的LC-HRMS系统上分析细胞提取物。可用于细胞提取物的分析的任何代谢的方法。全扫描分析可能是用于检测广泛的代谢物是有用的。
- 校准用质谱参考校准混合仪器。
- 在冰上解冻细胞提取物30分钟,并涡旋15秒。
- 以13,000 xg离心转移100μL的细胞提取物与一个旋转过滤器,离心10分钟,在4℃。
- 注入滤液12.5微升到在LC-HRMS的系统。
- 使用两性离子亲水相互作用液相色谱(ZIC-HILIC)柱(150毫米×4.6毫米,5微米颗粒尺寸)分离代谢物嵌合用0.1%甲酸的梯度洗脱一个ZIC-HILIC保护柱英寸(20毫米×2.1 MM)酸在水(溶剂A)和乙腈(溶剂B)。在溶剂A为20%开始梯度洗脱,并在17分钟增加至80%。保持在23℃和400微升的流分-1和柱温和样品盘4℃在4分钟该百分比,分别。
- 使用Instrument耦合到用于代谢物的检测,在正的和负的模式作为电离源加热的电(H-ESI II)中,并在70000全宽度半最大值(FWHM)的质量分辨能力完全扫描采集模式色谱分离( M / Z 200)。
- 用于鞘气和辅助气体氮气(纯度> 99.995%)以45的流速和10 AU(任意单位),并设置蒸发器温度为350℃,并在正模式和-3.5千伏4千伏的电电压在否定模式。
3.数据分析
- 选择一些代谢物的量的标准是可用的并且其显示样品中良好的质量峰。良好的质量峰具有高信号噪声比。它验证峰值质量,确保不包含虚假同位素是很重要的。这在形状和/或保留时间不同同位素峰可能假。
- 对于同位素标记的样本计算的质量同位素(MI)通过将每心肌梗死的峰面积与所有多边的总峰面积分数。
- 分别计算标记碳/氮分数,13 C和15 N,聚财,为
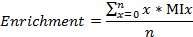
其中n是在代谢物(分别或氮,)的总碳数,混匀是x的MI分数。
注:所有计算可以使用的编程语言来执行。
结果
作为一个例子,在这里,我们描述了一种实验调查HeLa细胞根据细胞周期相排序的代谢。要标记范围广两个碳原子和氮代谢的中心,我们培养的细胞用U- 13 C葡萄糖和U- 13 C,15 N谷氨酰胺作为示踪剂48小时。要获取验证实验丰富移动互联网设备,我们在这里选择了40%的U- 13 C葡萄糖和70%U- 13 C,15 N2谷氨酰胺混合后作为同位素?...
讨论
我们的方法是基于这样的原则,在细胞代谢产物的MID反映的细胞代谢活动的"历史"。这使得有可能研究的代谢活性在细胞的亚群,因为它们发生在细胞的复杂社区,细胞分选过程之前。与此相反,代谢物的峰面积分选的细胞,并从培养皿11直接提取的提取物之间明显不同。这部分是因为不同的化学组成改变在质谱,一个所谓的"基体效应"的信号响应,但是我们还表明,氨基酸选自?...
披露声明
作者什么都没有透露。
致谢
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
材料
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
参考文献
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。