A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
שיטה למדידת מטבוליזם ב ממוינות תת-אוכלוסיות של קהילות Cell מורכבות באמצעות איתור איזוטופ יציב
In This Article
Summary
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
Abstract
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
Introduction
יצורים עילאיים להכיל קהילות שלמות מורכבות של סוגי תא ברורים כי לשתף פעולה כדי להביא פונקציות מורכבות יותר. לדוגמא, גידולים לכלול לא רק תאים סרטניים, אלא גם פיברובלסטים, תאים המהווים כלי דם, ולעתים קרובות תא חיסון מחלחל 1; דם מכיל תערובת מורכבת של עשרות תת תא חיסון 2; ואפילו שורות תאים בתרבית עשויות להיות מורכבות תת-אוכלוסיות מרובות, כגון לומינל, תת בסיס של תאי סרטן השד 3. יתר על כן, סוגי תא ברורים כי לדור בכפיפה אחת יכול להפגין מטבולית "שיתוף פעולה". לדוגמה, במוח, האסטרוציטים הם חשבו להמיר גלוקוז לקטט, אשר אז "הפד" לנוירונים כי לחמצן מצע זה 4; לימפוציטים מסוג T הם בהקשרים מסוימים תלויים תאים דנדריטים סמוכים כמקור ציסטאין 5; ותאים סרטניים יכולים לשתף פעולה עם Assoפיברובלסטים ciated בגידולים 6. כדי להבין את ההתנהגות המטבולית של מערכות כאלה, חשוב להפריד ולמדוד את הפעילות המטבולית של תאים מסוגים שונים בהווה.
ללא ספק השיטה הנפוצה ביותר להפרדת סוגי תאים היא מיון תא מופעל קרינה. שיטה זו היא החלימה רחב, ובלבד סוג התא או המדינה של עניין ניתן "שכותרתו" באמצעות נוגדני ניאון, ביטוי של חלבוני ניאון מהונדסים, או צבעים אחרים. אפשרות אחת היא סוגי תאים נפרדים בתחילה באמצעות סדרן תא, מחדש תרבות סוגי התאים הבודדים מתקבלים, ולאחר מכן לבצע מחקרים מטבוליזם של תרבויות אלה 7. עם זאת, זה אינו ריאלי רק אם הסוג או פנוטיפ התא יציב בתנאי תרבות, ולא יכול ללכוד התנהגות חולפת כמו מצבי מחזור התא, ולא את שיתוף הפעולה המטבולית שיתוף תרבויות. במקרים כאלה, מטבוליזם חייב להימדד ישירות על כךתאי rted. זה מאתגר מאז מיון תא הליך נושאים לתאים מדגיש כי עלול לעוות את חילוף החומרים שלהם 8, ואנו מודעים רק מעט מחקרי נקיטת גישה זו 9, 10. בפרט, מצאנו כי מטבוליטים גדולים כמו חומצות אמינו עלולים לדלוף מתאים שמרה במאגר מיון תא, כך שמדידות של שפע המטבוליט מוחלט הם כבר לא אמינות 11 (אם כי השוואה יחסית בין שברי מיון עדיין עשויה להיות בעלי ערך).
כדי לעקוף בעיות אלה, אנו תווית תאים עם איזוטופים יציבים לפני מיון, ולהתמקד MIDs מטבוליטים הסלולר, ולא שכיחותם המטבוליט. מאז MIDs נוצרות על סולמות זמן רב יותר, הם צריכים להיות מושפעים פחות על ידי חשיפה קצרת טווח לתנאים מיון. אנחנו לכמת MIDs באמצעות ספקטרומטריית מסה מלאה ברזולוציה גבוהה סריקה, שהוא רגיש מספיק כדי לספק data על מאה מטבוליטים החלו מתאי סביב 500,000 מסודרים, המחייב כ 30-60 דקות של זמן על מיון תא. השוואה בין "מדומה מסודרת" שליטה (תאים עברו מכשיר סדרן תא ללא gating כל אוכלוסייה ספציפית) והפקת המטבוליט ישירות מצלחת התרבות הוא עשתה כדי להבטיח כי MIDs הנצפה הם שמייצגים את התרבות המקורית. בהתאם הבחירה של קליעים נותבים איזוטופ יציב, מסלולים מטבוליים שונים ניתן ללמוד עם שיטה זו.
Protocol
1. הפקת המטבוליט
- הפקת מצלחת
- התרבות תאים בצלחת 6-היטב triplicates בתקשורת תרבות יציבה שכותרתו איזוטופ + תוספי dialyzed (סרום או תוספי גדילה אחרים) עד התאים להיות ומחוברות 75%.
הערה: כאן תאי הלה תרבות עבור 48 שעות RPMI המכילות 40% U- 13 C-גלוקוז ו -70% U- 13 C, 15 N2-גלוטמין ו -5% FBS dialyzed (בסרום שור עוברי). Dialyzed FBS משמש להיפטר מטבוליטים משקל מולקולרי קטן אשר עשוי לזהם את התקשורת שכותרתו. Culturing תאים במוסף dialyzed לפני הניסוי האמיתי מומלץ להבטיח התאים גדלים בדרך כלל בטווח הבינוני. תוספים הם dialyzed ב 0.15 פתרון M NaCl לילה באמצעות צינורות עור דיאליזת נחש. - באותו יום של התקשורת בתרבות החילוץ שנזרקו, ולשטוף בארות פעמיים עם 500 μL HBSS קר ולאחר מכן למחוק אותו.
הערה: כאן להשתמש HBSS המכיל 40% U-C-גלוקוז 13 שכן הוא גם בתקשורת ובתרבות. - להוסיף 600 μL 100% מתנול מראש מקורר על קרח יבש.
- מעבירים את צלחת לייבוש קרח ולהסיר את החומרים בתא עם מגרד תא.
- בזהירות פיפטה תמציות לתא צינור ולאחסן microcentrifuge ב -80 ° C עד לניתוח ספקטרומטריית מסה.
- התרבות תאים בצלחת 6-היטב triplicates בתקשורת תרבות יציבה שכותרתו איזוטופ + תוספי dialyzed (סרום או תוספי גדילה אחרים) עד התאים להיות ומחוברות 75%.
- הפקת תאים ממוינים מדומים
- התרבות תאים בצלחת 100 מ"מ triplicates ב + התקשורת והתרבות יציב שכותרתו איזוטופ תוספי dialyzed.
הערה: כאן תאים הלה תרבות במשך 48 שעות בצלחת 100 מ"מ כי מספר גבוה של תאים (~ 4 x 10 6) נדרשים כדי להשיג 500,000 תאים ממוינים. מספר זה של תמצית תאים הלה נדרש להשיג מדידה של מטבוליטים טוב. תקשורת התרבות המשמשת הייתה RPMI המכיל 40% U- 13 C-גלוקוז ו -70% U- 13 C, 15 N2-גלוטמין ו -5% dialyzed FBS. - באותו היום של מיצוי, תקשורת ותרבות שנזרקה, ולשטוף בארות wה- i 1.5 מ"ל חם HBSS ולאחר מכן למחוק אותו.
- ניתוק התאים על ידי הוספת 1.5 טריפסין מ"ל / EDTA במשך 4 דקות ב 37 מעלות צלזיוס. בצע את השלבים הבאים ב 4 ° C או בקרח.
- Deactivate טריפסין על ידי הוספת 3 מ"ל קרח קר HBSS (תמיסת מלח מאוזן של האנק) + תוספת dialyzed.
- איסוף תאים בתוך שפופרת צנטריפוגות 15 מ"ל ב 750 XG במשך 3 דקות.
- Resuspend גלולה ב HBSS + dialyzed תוספת + 1 mM EDTA בריכוז 1-2 x 10 6 תאים / מ"ל, לעבור דרך 40 מיקרומטר מסננות תא להשיג תאים בודדים, ולהעביר צינור 5 מ"ל.
- מיין תאים באמצעות gating סדרן התא רק singlets, ו תאים ממוינים צנטריפוגות ב 750 XG 3 דקות ב 4 ° C..
הערה: כאן מעין תאים הלה בשיעור 1,000 אירועים / s, עם psi 27 בלחץ המכשיר, ושימוש זרבובית 100 מיקרומטר. תאי הלה הם גדולים ולכן הם צריכים להיות מסודרים בקצב אירוע איטי זרבובית גדולה כדי לקבל גלולה ללא פגע ככל האפשר. שמור את התאים בבלוקים קרים throughout מיון כדי להקטין את חילוף החומרים. תיאור מפורט של הליך מיון מתואר בעבר 12. - בטל supernatant ו resuspend גלולה ב 50 μL קרח קר DH 2 O להשיג גלולה הומוגנית לפני הוספת מתנול. תוספת של מתנול קר ישירות גלול יוצרת גלולה מוצקה שקשה resuspend.
- חלץ מטבוליטים על ידי הוספת 540 μL מתנול שמרו על קרח יבש ולשמור תמציות ב -80 ° C עד כרומטוגרפיה נוזלית - ספקטרומטריית מסה ברזולוציה גבוהה ניתוח (LC-HRMS).
- התרבות תאים בצלחת 100 מ"מ triplicates ב + התקשורת והתרבות יציב שכותרתו איזוטופ תוספי dialyzed.
- הפקת מחזור התא מסודרת תאים
- התרבות תאים בצלחת 100 מ"מ triplicates בתקשורת תרבות + תוספי dialyzed.
הערה: כאן התרבות תאים הלה המכיל Geminin Fucci גרין (mAG1-hGem) בדיקה 13 המאפשר למיון של G1 (שלילי) ו SG2M (חיובי) תאים. תאים יכולים להיות מתורבתים במדיה התחקות איזוטופ יציבים עבורזה עוד נדרש. כאן יש לנו את התרבית אותם במשך 46 שעות ב מדיה ללא תווית, אז אנחנו עברנו RPMI המכיל 40% U- 13 C-גלוקוז ו -70% U- 13 C, 15 N2-גלוטמין 2 שעות לפני תחילת המיון. הדבר נעשה על מנת להיות מסוגל ללמוד את שלבי מחזור התא מחייב תיוג דופק קצר. - באותו יום של מיצוי, התקשורת והתרבות שנזרקו, ולשטוף בארות עם 1.5 מ"ל חם HBSS ולאחר מכן למחוק אותו.
- ניתוק התאים על ידי הוספת 1.5 טריפסין מ"ל / EDTA במשך 4 דקות ב 37 מעלות צלזיוס. בצע את השלבים הבאים ב 4 ° C או בקרח.
- Deactivate טריפסין על ידי הוספת 3 מ"ל קרח קר HBSS + תוספת dialyzed.
- איסוף תאים בתוך שפופרת צנטריפוגות 15 מ"ל ב 750 XG במשך 3 דקות.
- Resuspend גלולה ב HBSS + dialyzed תוספת + 1 mM EDTA בריכוז 1-2 x 10 6 תאים / מ"ל, לעבור דרך 40 מיקרומטר מסננות תא להשיג תאים בודדים, ולהעביר צינור 5 מ"ל.
- תאי מיין הדרך גרם סדרן התאating החוצה פסולת כפילויות, אז gating עבור סמן תא של עניין, תאים ממוינים צנטריפוגות ב 750 XG 3 דקות ב 4 ° C..
הערה: כאן מעין תאים הלה בשיעור 1,000 אירועים / s, עם psi 27 בלחץ המכשיר, ושימוש זרבובית 100 מיקרומטר. שמור את התאים בבלוקים קרים לאורך מיון כדי להקטין את קצב חילוף חומרים. - בטל supernatant ו resuspend גלולה ב 50 μL קרח קר DH 2 O להשיג גלולה הומוגנית לפני הוספת מתנול. תוספת של מתנול קר ישירות גלול יוצרת גלולה מוצקה שקשה resuspend.
- חלץ את מטבוליטים על ידי הוספת 540 μL מתנול שמרו על קרח יבש ולשמור תמציות ב -80 ° C עד לניתוח LC-HRMS.
- התרבות תאים בצלחת 100 מ"מ triplicates בתקשורת תרבות + תוספי dialyzed.
2. ניתוח ספקטרומטריית מסה
הערה: כאן אנו מתארים את הפרוטוקול לניתוח תמציות תא על מערכת LC-HRMS. כל שיטות metabolomics לניתוח של תמציות התא יכול לשמש. סריקה מלאהניתוח יכול להיות שימושי לצורך גילוי מגוון רחב של מטבוליטים.
- כייל את המכשיר באמצעות שילוב כיול התייחסות ספקטרומטריית מסה.
- תמציות תאי הפשרה על קרח למשך 30 דקות ו מערבולת למשך 15 שניות.
- העברת 100 μL של תמצית התא מסנן ספין צנטריפוגות במשך 10 דקות ב 13,000 XG ב 4 ° C..
- להזריק 12.5 μL של תסנין על מערכת LC-HRMS.
- מטבוליטים נפרדים באמצעות כרומטוגרפיה נוזלית אינטראקציה zwitterionic הידרופיליות (ZIC-HILIC) טור (150 מ"מ x 4.6 מ"מ, 5 מיקרומטר גודל החלקיקים) המצויידים טור השומר ZIC-HILIC (20 מ"מ × 2.1 מ"מ) באמצעות elution שיפוע של פורמית 0.1% חומצה במים (ממס) ו אצטוניטריל (B ממס). הפעל את elution שיפוע של 20% ממס A ו- להגדיל עד 80% ב -17 דקות. לשמור על אחוז זה במהלך 4 דקות עם זרימה של 400 μL דקות -1 וטמפרטורת טור ומגש מדגם ב 23 מעלות צלזיוס ו 4 ° C, בהתאמה.
- השתמש instrument מצמידים את ההפרדה chromatographic לגילוי מטבוליטים, electrospray מחוממת (H-ESI II) בשני מצבים חיוביים ושליליים כמקור יינון, לבין מצב הרכישה סריקה מלאה בכל הכוח בפתרון המוני של 70,000 מלא רוחב חצי מקסימום (FWHM) ( מ '/ z 200).
- השתמש חנקן (טוהר> 99.995%) עבור גז נדן וגז עזר בקצב זרימה של 45 ו -10 au (יחידות שרירותיות) וטמפרטורה מאדה להגדיר ב 350 ° C ואת מתח electrospray ב 4 ק במצב חיובי -3.5 kV במצב שלילי.
.3 ניתוח נתונים
- בחר מספר מטבוליטים עבורו סטנדרטים זמינים אשר מראים פסגות באיכות טובה בדגימות. יש פסגות באיכות טובות אות גבוהה יחס רעש. חשוב לוודא איכות שיא ולוודא שלא לכלול איזוטופים שווא. פסגות איזוטופים אשר נבדלות צורה ו / או זמן שמירה הן שקריות סבירות.
- לקבלת דוגמיות שכותרתו איזוטופ לחשב isotopomer מסה (MI) שברים על ידי חלוקת אזור השיא של כל MI עם אזורי שיא כוללים של כל MIS.
- חישוב שברי פחמן / חנקן שכותרתו, העשרה של 13 C ו -15 N, בהתאמה, כפי
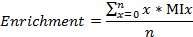
כאשר n הוא המספר הכולל של פחמנים (או חנקנים, בהתאמה) המטבוליט, ומערבבים הוא שבריר MI של x.
הערה: כל החישובים יכולים להתבצע באמצעות שפות תכנות.
תוצאות
כדוגמא, כאן אנו מתארים ניסוי חוקר את חילוף החומרים של תאים הלה ממוין לפי שלב מחזור התא. כדי לתייג מגוון רחב של מטבוליטים מרכזיים משני פחמנים חנקנים, אנו בתרבית תאים עבור 48 שעות באמצעות U- 13 C-גלוקוז U- 13 C, 15 N-גלוטמין קליעים נותבים. כדי ל...
Discussion
השיטה שלנו מבוססת על העיקרון כי MIDs מטבוליטים הסלולר לשקף את "ההיסטוריה" של פעילות מטבולית של התא. זה מאפשר לחקור פעילויות מטבוליים תת-אוכלוסייה של תאים, בעת התרחשותם בקהילה הסבוכה של תאים, לפני הליך מיון התא. לעומת זאת, באזורי שיא של מטבוליטים שונים באופן ניכר בי?...
Disclosures
החוקרים אין לי מה לחשוף.
Acknowledgements
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
Materials
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
References
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved