JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
안정 동위 원소 추적을 사용하여 복잡한 세포 공동체의 정렬 된 부분 집단에서 대사를 측정하는 방법
요약
This article describes a method for studying cellular metabolism in complex communities of multiple cell types, using a combination of stable isotope tracing, cell sorting to isolate specific cell types, and mass spectrometry.
초록
Mammalian cell types exhibit specialized metabolism, and there is ample evidence that various co-existing cell types engage in metabolic cooperation. Moreover, even cultures of a single cell type may contain cells in distinct metabolic states, such as resting or cycling cells. Methods for measuring metabolic activities of such subpopulations are valuable tools for understanding cellular metabolism. Complex cell populations are most commonly separated using a cell sorter, and subpopulations isolated by this method can be analyzed by metabolomics methods. However, a problem with this approach is that the cell sorting procedure subjects cells to stresses that may distort their metabolism.
To overcome these issues, we reasoned that the mass isotopomer distributions (MIDs) of metabolites from cells cultured with stable isotope-labeled nutrients are likely to be more stable than absolute metabolite concentrations, because MIDs are formed over longer time scales and should be less affected by short-term exposure to cell sorting conditions. Here, we describe a method based on this principle, combining cell sorting with liquid chromatography-high resolution mass spectrometry (LC-HRMS). The procedure involves analyzing three types of samples: (1) metabolite extracts obtained directly from the complex population; (2) extracts of "mock sorted" cells passed through the cell sorter instrument without gating any specific population; and (3) extracts of the actual sorted populations. The mock sorted cells are compared against direct extraction to verify that MIDs are indeed not altered by the cell sorting procedure itself, prior to analyzing the actual sorted populations. We show example results from HeLa cells sorted according to cell cycle phase, revealing changes in nucleotide metabolism.
서문
고등 생물은 더 복잡한 기능에 대해 가지고 협력 별개의 세포 유형의 복잡한 사회를 포함한다. 예를 들어, 종양은 단지 암세포 만, 섬유 아세포, 혈관을 구성하고, 주로 면역 세포가 침투 한 세포를 포함하고; 혈액은 면역 세포 아형 2의 수십의 복잡한 혼합물을 포함, 심지어 배양 세포주는 내강 및 유방암 세포 (3)의 기부 아형 여러 개체군, 구성 될 수있다. 대사 "협력"을 나타낼 수있다 공존 또한, 서로 다른 세포 유형. 예를 들어, 뇌, 성상이 기판 (4)을 산화 한 후 뉴런 "공급"인 락트산 포도당 변환 생각된다; T 림프구는 시스테인 (5)의 소스로 인접 수지상 세포에 의존하는 일부 경우에; 암 세포는 asso과 협력 할 수있다종양 6 ciated 섬유 아세포. 이러한 시스템의 대사 동작을 이해하기 위해서는 본 분리하고 다양한 종류의 세포 대사 활동을 측정하는 것이 필수적이다.
지금까지 세포 유형을 분리하기위한 가장 널리 사용되는 방법은 형광 활성화 세포 분류이다. 이 방법은 세포 유형 또는 관심 상태는 형광 항체, 조작 된 형광 단백질의 발현 또는 다른 염료를 사용하여 "표지"가 제공 될 수 넓게 적용 가능하다. 하나의 옵션은 셀 소터 통해 초기 개별 세포 유형, 다시 배양 얻어진 각 세포 유형이다하고이 배양 물 (7)의 대사 과정을 수행한다. 세포 유형 또는 표현형은 배양 조건에 안정하며, 이러한 세포주기 상태 않으며 공동 배양 물에서 대사 협력으로 과도 행동을 캡처 할 수없는 경우,이 경우에만 가능하다. 이러한 경우를 들어, 신진 대사가 너무 직접 측정해야rted 세포. 이 절차를 선별 세포 신진 대사 8 왜곡 될 응력 세포 주제 때문에 도전이며, 우리는이 방법 (9) (10)를 가지고 단지 약간의 연구 알고있다. 특히, 절대 대사 풍부 측정이 더 이상 신뢰성있게 아미노산 등 주요 대사 산물은 세포 정렬 버퍼에 보관 세포로부터 누출하지 않을 수 있음을 발견 하였다 (11) (정렬 된 분획의 상대 비교가 여전히 유용 할 수 있음).
이러한 문제를 회피하기 위해, 우리는 정렬하기 전에 안정 동위 원소와 셀 라벨, 및 세포 대사보다는 대사 산물 존재비에서의 MID에 초점을 맞 춥니 다. MID를 길게 시간 규모 상에 형성되기 때문에, 그들은 적은 정렬 조건에 단기간 노출에 의해 영향을받을 것이다. 우리는 DA를 제공하기에 충분히 민감 전체 스캔 고분해능 질량 분석법을 이용하여 MID를 정량화셀 정렬 시간의 약 30 ~ 60 분을 필요로하는 약 50 만 분류 세포에서 시작 대사 산물의 수백에 따. 배양 접시가 관찰 된 MID는 원래 배양 대표적되도록 이루어진다에서의 비교를 직접 제어 (특정 인구 게이팅없이 셀 소터 구 통과 세포) 및 대사 추출 "모의 정렬". 안정 동위 원소 추적자의 선택에 따라 다양한 대사 경로는이 방법으로 공부하실 수 있습니다.
프로토콜
1. 대사 추출
- 접시에서 추출
- 안정 동위 원소 표지 된 배양 배지 + 투석 영양 보충제 (혈청 또는 다른 성장 보조제)에서 삼중의 6 웰 플레이트에서 배양 세포는 세포는 75 %의 합류가 될 때까지.
주 : U- 13 C 40 % 글루코스를 함유하는 RPMI에서 48 시간 70 % U- 13 C, 15-N2 글루타민 및 5 % 투석 된 FBS (태아 소 혈청)은 여기 HeLa 세포 배양. 투석 FBS는 레이블이 미디어를 오염시킬 수있는 작은 분자량 대사 물질을 제거하는 데 사용됩니다. 실제 실험 이전에 투석 보충에 세포를 배양하는 세포가 배지에서 정상적으로 성장하고 확인하는 것이 좋습니다. 보충 밤새 뱀 피부 투석 튜브를 사용하여 0.15 M NaCl 용액에서 투석된다. - 추출 폐기 문화 매체의 날에, 500 μL 차가운 HBSS로 두 번 우물을 씻어 후 폐기합니다.
참고 : 여기에 HBSS 40 %를 포함하는 사용 U-13 C 글루코스가 배양 배지에도 때문이다. - 추가 600 μL의 100 % 메탄올을 드라이 아이스에 미리 냉각시켰다.
- 얼음을 건조 세포 스크레이퍼와 세포 물질을 제거 할 수있는 요리를 전송합니다.
- 조심 질량 분광 분석까지 -80 ° C에서 microcentrifuge 관 및 저장소에 세포 추출물 피펫.
- 안정 동위 원소 표지 된 배양 배지 + 투석 영양 보충제 (혈청 또는 다른 성장 보조제)에서 삼중의 6 웰 플레이트에서 배양 세포는 세포는 75 %의 합류가 될 때까지.
- 모의 분류 세포의 추출
- 안정 동위 원소 표지 된 배양 배지 + 투석 보충 교재에서 삼중의 100mm 접시에 배양 세포.
주 : 100mm 접시에 48 시간 동안 배양 여기 HeLa 세포 세포의 높은 번호 (~ 4 × 106)은 50 정렬 세포를 얻기 위해 필요하기 때문이다. 헬라 세포 추출물이 숫자는 대사 산물의 좋은 측정을 받아야했다. 사용되는 배양 배지는 40 % RPMI는 U- 13 C 글루코스 70 % U- 13 C, 15-N2 글루타민 투석 FBS, 5 %를 함유 하였다. - 추출, 폐기 문화 매체의 날에, w 우물을 씻어i 번째 1.5 mL의 따뜻한 HBSS 후 폐기합니다.
- 37 ℃에서 4 분 동안 150 mL의 트립신 / EDTA를 첨가하여 세포를 분리. 4 ° C 또는 얼음에서 다음 단계를 수행합니다.
- 3 mL의 얼음 차가운 HBSS (행크의 균형 소금 솔루션) + 투석 보충을 추가하여 트립신을 비활성화합니다.
- 3 분 동안 750 XG에 15 mL의 튜브와 원심 분리기에서 세포를 수집합니다.
- 1-2 × 10 6 세포 / ㎖가, 단일 세포를 얻기 위해 40 μm의 셀 스트레이너를 통과하고, 5 ㎖ 튜브로 전송 농도로 HBSS + 투석 보충 + 1 mM의 EDTA의 펠렛을 재현 탁.
- 정렬 만 단봉들에 대한 셀 소터 게이팅을 통해 세포, 4 ℃에서 3 분 동안 750 XG에 원심 분리기 분류 세포.
참고 : 여기에 정렬 헬라 계기 압력 27 PSI와 속도 1000 이벤트 / s로 세포, 및 100 μm의 노즐을 사용하여. 헬라 세포는 크고 그래서 그들은 가능한 한 그대로 펠렛을 얻기 위해 느린 이벤트 속도와 큰 노즐로 분류되어야한다. throughou 감기 블록 세포 유지t은 신진 대사를 감소 정렬. 정렬 절차의 철저한 설명은 이전 12 설명한다. - 뜨는을 취소하고 메탄올을 추가하기 전에 균일 한 펠렛을 얻기 위해 50 μL 얼음 차가운 DH 2 O의 펠렛을 재현 탁. 직접 펠릿을 냉 메탄올에 첨가 재현 탁 어렵다 고체 펠렛을 형성한다.
- 드라이 아이스에 보관 540 μL 메탄올을 추가하여 대사 물질을 추출하여 액체 크로마토 그래피 때까지 -80 ° C에서 추출물을 유지 - 고해상도 질량 분석 (LC-HRMS) 분석.
- 안정 동위 원소 표지 된 배양 배지 + 투석 보충 교재에서 삼중의 100mm 접시에 배양 세포.
- 세포주기의 추출 세포 정렬
- 문화 미디어 + 투석 보충 교재에서 삼중의 100mm 접시에 배양 세포.
참고 : Geminin Fucci 녹색 G1 (음)과 SG2M (양의) 세포로 정렬 할 수 있습니다 (MAG1-hGem) 탐침 (13)를 포함하는 다음 문화 HeLa 세포. 세포는 안정 동위 원소 추적 미디어에서 배양 될 수있다필요에 따라 긴이야. 여기에 우리가 레이블이없는 미디어에서 46 시간 동안을 배양 한 후, 우리는 RPMI 40 % U- (13) C-포도당 70 % U- (13) C를 함유로 전환 한 15 N2 - 글루타민 2 시간 전에 정렬 시작. 이것은 짧은 펄스 라벨링 필요한 세포주기 단계를 연구 할 수 있도록하기 위해 수행되었다. - 추출, 폐기 문화 매체의 날에, 1.5 mL의 따뜻한 HBSS와 우물을 씻어 후 폐기합니다.
- 37 ℃에서 4 분 동안 150 mL의 트립신 / EDTA를 첨가하여 세포를 분리. 4 ° C 또는 얼음에서 다음 단계를 수행합니다.
- 3 mL의 얼음 차가운 HBSS + 투석 보충을 추가하여 트립신을 비활성화합니다.
- 3 분 동안 750 XG에 15 mL의 튜브와 원심 분리기에서 세포를 수집합니다.
- 1-2 × 10 6 세포 / ㎖가, 단일 세포를 얻기 위해 40 μm의 셀 스트레이너를 통과하고, 5 ㎖ 튜브로 전송 농도로 HBSS + 투석 보충 + 1 mM의 EDTA의 펠렛을 재현 탁.
- 셀 소터 g을 통해 정렬 세포다음, 파편과 이중선을 ating 관심의 세포 마커에 대한 게이팅, 원심 분리기 분류 세포를 4 ℃에서 3 분 동안 750 XG에.
참고 : 여기에 정렬 헬라 계기 압력 27 PSI와 속도 1000 이벤트 / s로 세포, 및 100 μm의 노즐을 사용하여. 신진 대사 속도를 감소 정렬을 통해 차가운 블록 세포를 유지합니다. - 뜨는을 취소하고 메탄올을 추가하기 전에 균일 한 펠렛을 얻기 위해 50 μL 얼음 차가운 DH 2 O의 펠렛을 재현 탁. 직접 펠릿을 냉 메탄올에 첨가 재현 탁 어렵다 고체 펠렛을 형성한다.
- 드라이 아이스에 보관 540 μL 메탄올을 추가하여 대사 물질을 추출 및 LC-HRMS 분석 할 때까지 -80 ° C에서 추출물을 유지합니다.
- 문화 미디어 + 투석 보충 교재에서 삼중의 100mm 접시에 배양 세포.
2. 질량 분석 분석
참고 : 여기에 우리가 LC-HRMS 시스템에서 세포 추출물을 분석하기위한 프로토콜을 설명합니다. 세포 추출물의 분석에 대한 대사 체학 방법이 사용될 수있다. 전체 검사분석은 대사 산물의 다양한 검출에 유용 할 수있다.
- 질량 분석 참조 교정 믹스를 사용하여 기기를 보정합니다.
- 15 초 30 분, 소용돌이 얼음에 해동 세포 추출물.
- 4 ℃에서 13,000 × g으로 10 분 동안 스핀 필터 및 원심 분리하여 세포 추출물 100 ㎕를 옮긴다.
- 액정-HRMS 시스템에 여액 12.5 μL를 주입한다.
- (ZIC-HILIC) 칼럼 (150mm X 4.6 mm, 5 ㎛의 입자 크기) 양쪽이 온성 친수성 상호 작용 액체 크로마토 그래피를 이용하여 분리 된 대사 물질 0.1 % 포름산 구배 용출을 이용하여 ZIC-HILIC 가드 컬럼 (20mm mm 2.1 ×) 장착 물에 산 (용매 A) 및 아세토 니트릴 (용매 B). 용매 (A)의 20 % 구배 용출를 시작하고 17 분에서 80 %까지 증가한다. 각각 최소 400 μL의 흐름 -1 컬럼 온도 및 샘플 트레이 23 ° C에서 4 ° C와 4 분 동안이 비율을 유지한다.
- 는 I를 사용하여대사 검출 이온화 소스로서 포지티브 및 네거티브 모드 모두에서 가열 된 전기 분무 (H-ESI II) 및 70,000 전폭 반치 (FWHM)의 질량 분해능의 전체 검사를 획득 모드의 크로마토 그래피 분리에 결합 nstrument ( m / z 200).
- 플로우 (45)의 속도와 350 ° C에서 10 노소 (임의의 단위) 및 설정 증발기 온도와 긍정적 인 모드와 -3.5 kV의 4 kV로에서의 전기 분무 전압에서 시스 가스 및 보조 가스 질소 (순도> 99.995 %)를 사용하여 부정적인 모드입니다.
3. 데이터 분석
- 표준이 가능하며 샘플에서 좋은 품질의 피크를 표시하는 대사 산물의 번호를 선택합니다. 양질의 피크 대 잡음비가 높은 신호. 최고 품질을 확인하고 거짓 동위 원소를 포함하지 있는지 확인하는 것이 중요합니다. 모양 및 / 또는 체류 시간이 다른 동위 원소 피크 가능성이 false입니다.
- 동위 원소 표지 샘플 (질량 동위 원소를 계산을 위해MI) 모든 잘못의 총 피크 면적과 각 MI의 피크 면적을 나누어 분수.
- 로 각각 13 C, 15 N의 농축을 표지 탄소 / 질소 분수를 계산
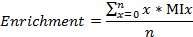
여기서 n은 대사 산물의 탄소의 총 수 (또는 질소 각각은)이며, 혼합 x의 MI 분율이다.
주 : 모든 계산은 프로그래밍 언어를 사용하여 수행 될 수있다.
결과
일례로서, 우리가 세포주기의 단계에 따라 정렬 HeLa 세포의 신진 대사를 조사하는 실험을 기술한다. 모두 탄소와 질소에 중앙 대사의 다양한 레이블을 지정하려면, 우리는 U- (13) C-포도당과 U-13 C, 추적자로 15 N 글루타민을 사용하여 48 시간 동안 세포를 배양. 동위 원소의 중간 수준보다 다양한 MI 패턴 (14)을 생성하는 경향이 검증 ...
토론
우리의 방법은 세포의 대사에서의 MID는 세포 대사 활동의 "히스토리"를 반영한다는 원리에 기초한다. 이것은 그들이 이전 셀 정렬 절차 세포의 복잡한 지역에서 발생으로 가능 세포의 하위 집단에서 대사 활동을 조사 할 수있다. 반면, 대사 산물의 피크 지역 분류 세포와 배양 접시 (11)에서 직접 추출 추출 사이에 현저하게 다르다. 일부 다른 화학 성분은 질량 분석, 소위 ...
공개
저자는 공개 아무것도 없어.
감사의 말
The authors would like to thank Dr. Anas Kamleh for valuable help with optimizing mass spectrometry methods, and Annika von Vollenhoven for assistance with cell sorting. This research was supported by the Swedish Foundation for Strategic Research (grant no. FFL12-0220) and the Strategic Programme in Cancer Research (IR, RN); the Swedish Heart-Lung Foundation (CEW, HG); and Mary Kay Foundation (JW, MJ).
자료
| Name | Company | Catalog Number | Comments |
| HBSS | Sigma | H6648 | |
| INFLUX (inFlux v7 Sorter) | BD Biosciences | ||
| U-13C-Glucose | Cambridge isotopes | 40762-22-9 / GLC-018 | |
| U-13C,15N2-Glutamine | Cambridge isotopes | CNLM-1275-H-0.1 | |
| Methanol (JT Baker), HPLC grade | VWR | BAKR8402.2500 | |
| Ultrafree - MC - VV centrifugal Filters. Durapore PVDF 0.1 µm | Millipore | UFC30VV00 | |
| Ultimate 3,000 UHPLC | Thermo Fisher scientific | ||
| Q-Exactive Orbitrap Mass spectrometer | Thermo Fisher scientific | ||
| Merk-Sequant ZIC HILIC column (150 mm x 4.6 mm, 5 µm) | Merck KGaA | 1.50444.0001 | |
| Merk-Sequant ZIC HILIC guard column (20 mm x 2.1 mm) | Merck KGaA | ||
| Acetonitrile Optima LC-MS, amber glass | Fisher Scientific | A955-212 | |
| Milli-Q water | Millipore | Produced with a Milli-Q Gradient system | |
| Myrsyra 99.5% Optima (Formic acid) | Fisher Scientific | 11423423 | |
| X100 Screw Vial 1.5 ml, 8-425 32x11.6 mm, amber, 100 units | Thermo Fisher scientific | 10560053 | |
| X100 Lock Skruv Vitt PTFE Packing 8-425 (Screw caps) | Thermo Fisher scientific | 12458636 | |
| ProteoMass LTQ/FT-Hybrid ESI Pos. Mode Cal Mix | Sigma-Aldrich | MSCAL5 | Calibration kit |
| SNAKESKIN 10K MWCO | Thermo Fisher scientific | 88245 | |
| Mathematica v.10 | Wolfram Research |
참고문헌
- Gregersen, P. K. Cell type-specific eQTLs in the human immune system. Nat. Genet. 44 (5), 478-480 (2012).
- Heppner, G. H. Tumor heterogeneity. Cancer Res. 44 (6), 2259-2265 (1984).
- Prat, A., et al. Characterization of cell lines derived from breast cancers and normal mammary tissues for the study of the intrinsic molecular subtypes. Breast Cancer Res. Treat. 142 (2), 237-255 (2013).
- Magistretti, P. J., Allaman, I. A Cellular Perspective on Brain Energy Metabolism and Functional Imaging. Neuron. 86 (4), 883-901 (2015).
- Angelini, G., et al. Antigen-presenting dendritic cells provide the reducing extracellular microenvironment required for T lymphocyte activation. Proc. Natl. Acad. Sci. U. S. A. 99, 1491-1496 (2002).
- Koukourakis, M. I., Giatromanolaki, A., Harris, A. L., Sivridis, E. Comparison of metabolic pathways between cancer cells and stromal cells in colorectal carcinomas: A metabolic survival role for tumor-associated stroma. Cancer Res. 66 (2), 632-637 (2006).
- Hollenbaugh, J. A., Munger, J., Kim, B. Metabolite profiles of human immunodeficiency virus infected CD4+ T cells and macrophages using LC-MS/MS analysis. Virology. 415 (2), 153-159 (2011).
- Richardson, G. M., Lannigan, J., Macara, I. G. Does FACS perturb gene expression. Cytom. Part A. 87 (2), 166-175 (2015).
- Moussaieff, A., et al. High-resolution metabolic mapping of cell types in plant roots. Proc. Natl. Acad. Sci. U. S. A. 110 (13), E1232-E1241 (2013).
- Johnson, C., et al. A metabolic signature of colon cancer initiating cells. Cancer Metab. 2, 32 (2014).
- Roci, I., et al. Metabolite Profiling and Stable Isotope Tracing in Sorted Subpopulations of Mammalian Cells. Anal. Chem. 88 (5), 2707-2713 (2016).
- Shapiro, H. . Practical flow cytometry. , (2003).
- Sakaue-Sawano, A., et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell. 132 (3), 487-498 (2008).
- Noack, S., Wiechert, W. Quantitative metabolomics: A phantom. Trends Biotechnol. 32 (5), 238-244 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유