A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
اختبار لتحديد كمية البروتين-RNA الربط في البكتيريا
In This Article
Summary
في هذه الطريقة، نقوم بتحديد التقارب الملزم للبروتينات الملزمة الحمض النووي الريبي (RBPs) إلى مواقع ربط الكُونات وغير الكونيات باستخدام اختبار مراسل بسيط ومباشر في الخلايا البكتيرية. ويستند هذا القول على قمع جين مراسل.
Abstract
في خطوة بدء ترجمة البروتين، يرتبط الريبوسم بمنطقة بدء mRNA. يمكن حظر بدء الترجمة عن طريق ربط بروتين ملزم الحمض النووي الريبي (RBP) إلى منطقة بدء mRNA، الذي يتداخل مع ربط ريبوسم. في الطريقة المعروضة، نستخدم هذه الظاهرة حجب لتحديد مدى التقارب ملزمة من RBPs إلى مواقعها ملزمة cognate وغير cognate. للقيام بذلك، نقوم بإدراج موقع ربط اختبار في منطقة بدء mRNA مراسل والحث على التعبير عن RBP الاختبار. وفي حالة ربط RBP-RNA، لاحظنا قمعاً السينياً للتعبير الصحفي كدالة لتركيز الممارسات التجارية التقييدية. وفي حالة عدم وجود تقارب أو تقارب منخفض جداً بين الموقع الملزم والممارسات التجارية التقييدية، لم يلاحظ أي قمع كبير. يتم تنفيذ هذه الطريقة في الخلايا البكتيرية الحية، ولا تتطلب آلات مكلفة أو متطورة. ومن المفيد تحديد الخصائص والمقارنة بين أوجه التقارب الملزمة لمختلف الممارسات التجارية التقييدية التي تعمل في البكتيريا إلى مجموعة من مواقع الربط المصممة. قد يكون هذا الأسلوب غير مناسب للمواقع الملزمة ذات التعقيد الهيكلي العالي. ويرجع ذلك إلى إمكانية قمع بدء ريبوسومال من قبل هيكل MRNA معقدة في غياب RBP، مما يؤدي إلى التعبير الجيني مراسل القاعدية أقل، وبالتالي قمع مراسل أقل ملاحظة على ربط RBP.
Introduction
وقد تمت دراسة اللوائح القائمة على البروتين الملزم للRNA (RBP) بعد النسخ، وعلى وجه التحديد توصيف التفاعل بين الممارسات التجارية التقييدية والحمض النووي الريبي، على نطاق واسع في العقود الأخيرة. هناك أمثلة متعددة من التنظيم أسفل الترجمة في البكتيريا الناشئة من RBPs تثبيط،أو تتنافس مباشرة مع، الريبوسم ملزمة 1،2،3. في مجال البيولوجيا التركيبية، تظهر تفاعلات RBP-RNA كأداة هامة لتصميم الدوائر الوراثية المستندة إلى النسخ4و5. ولذلك، هناك زيادة في الطلب على توصيف هذه التفاعلات بين الممارسات التجارية التقييدية والحمض النووي الريبي في سياق خلوي.
الطرق الأكثر شيوعا لدراسة التفاعلات البروتين الحمض النووي الريبي هي تحويل التنقل الكهربائي (EMSA)6، والذي يقتصر على إعدادات المختبر ، ومختلف الاختبارات المنسدلة7، بما في ذلك طريقة CLIP8،9 . في حين أن هذه الأساليب تمكن من اكتشاف مواقع الربط الحمض النووي الريبي دي نوفو, أنها تعاني من عيوب مثل بروتوكولات كثيفة العمالة وردود الفعل التسلسل العميق مكلفة وقد تتطلب جسم اضدي معين لRBP المنسدلة. بسبب الطبيعة القابلة للتأثر من الحمض النووي الريبي لبيئتها، يمكن أن تؤثر عوامل كثيرة على تفاعلات RBP-RNA، مع التأكيد على أهمية استجواب RBP-RNA ملزمة في السياق الخلوي. على سبيل المثال، لقد أظهرنا نحن وآخرون اختلافات كبيرة بين هياكل الحمض النووي الريبي في الجسم الحي وفي المختبر10،11.
استنادا ً إلى نهج دراسة سابقة12، أظهرنا مؤخرا10 أنه عند وضع مواقع ملزمة مصممة مسبقا للRBPs كابسيد من البكتيريا GA13، MS214، PP715، وQβ16 في ترجمة بداية منطقة من مراسلة [مرنا], مراسلة قمعت تعبير بقوّة. نحن نقدم طريقة بسيطة وكمية نسبيا، استنادا إلى هذه الظاهرة القمع، لقياس التقارب بين RBPs وما يقابلها RNA موقع ملزمs في الجسم الحي.
Protocol
1. إعداد النظام
- تصميم بلازميدات الموقع الملزم
- تصميم شريط كاسيت موقع الربط كما هو موضح في الشكل 1. كل المينيجين يحتوي على الأجزاء التالية (5' إلى 3'): Eagl موقع تقييد، و40 قواعد من 5 ' نهاية كانامايسين (كان) الجينات المقاومة، pLac-Ara المروج، موقع ربط ريبوسم (RBS)، AUG من الجين mCherry، فاصل (δ)، موقع ربط RBP، 80 قواعد من نهاية 5 ' من الجين mCherry، وموقع تقييد ApaLI.
ملاحظة: لزيادة معدل نجاح الاختبار، قم بتصميم ثلاثة أشرطة فيديو موقع ربط لكل موقع ربط، مع فواصل تتكون من قاعدة واحدة واثنين وثلاث قواعد على الأقل. راجع قسم النتائج التمثيلية للحصول على مزيد من المبادئ التوجيهية.
- تصميم شريط كاسيت موقع الربط كما هو موضح في الشكل 1. كل المينيجين يحتوي على الأجزاء التالية (5' إلى 3'): Eagl موقع تقييد، و40 قواعد من 5 ' نهاية كانامايسين (كان) الجينات المقاومة، pLac-Ara المروج، موقع ربط ريبوسم (RBS)، AUG من الجين mCherry، فاصل (δ)، موقع ربط RBP، 80 قواعد من نهاية 5 ' من الجين mCherry، وموقع تقييد ApaLI.
- استنساخ بلازميدات الموقع الملزمة
- اطلب أشرطة الكاسيت الملزمة كجينات صغيرة مزدوجة التقطعت بالحمض النووي (dsDNA). كل الجين المصغر هو 500 نقطة أساس طويلة ويحتوي على موقع تقييد Eagl وموقع تقييد ApaLI في 5' و 3' ينتهي، على التوالي (انظر الخطوة 1.1.1).
ملاحظة: في هذه التجربة، أمرت الجينات الصغيرة مع نصف جين كانامايسين لتسهيل الكشف عن المستعمرات الإيجابية. مهما, [جبسن] اجتماع17 أيضا مناسبة هنا, [إين وهيش يس] ال [كنفيك ستب] يستطيع كنت أمرت كاثنان قصيرة تكميليّة [سنغل-تّف] [دنا] [أوليغوس]. - مزدوجة هضم كل من الجينات المصغرة وناقلات الهدف مع Eagl-HF وApaLI من قبل بروتوكول تقييد18، وتنقية العمود19.
- يُحَمِّل الجينات الصغيرة المُهضمة إلى العمود الفقري في الموقع المُلزم الذي يحتوي على بقية جين مراسل mCherry، المنهي، وجين مقاومة كانامايسين20.
- تحويل محلول الربط إلى خلايا TOP10 القولونية الإشريكية 21.
- تحديد التحولات الإيجابية عن طريق تسلسل Sanger.
- تصميم التمهيدي 100 قواعد المنبع إلى المنطقة ذات الأهمية (انظر الجدول 1 للاطلاع على تسلسلات التمهيدي).
- Miniprep عدد قليل من المستعمرات البكتيرية22.
- إعداد 5 ميكرولتر من محلول 5 mM من التمهيدي و 10 ميكرولتر من الحمض النووي في تركيز 80 نانوغرام /ميكرولتر.
- إرسال الحل الثاني إلى منشأة مريحة لتسلسل Sanger23.
- تخزين بلازميدات تنقية في -20 درجة مئوية، وسلالات البكتيرية كما أسهم الجلسرين24،وكلاهما في شكل 96 جيدا. ثم سيتم استخدام الحمض النووي للتحول إلى خلايا E. coli TOP10 التي تحتوي على واحدة من أربعة بلازميدات الانصهار RBP (انظر الخطوة 1.3.5).
- اطلب أشرطة الكاسيت الملزمة كجينات صغيرة مزدوجة التقطعت بالحمض النووي (dsDNA). كل الجين المصغر هو 500 نقطة أساس طويلة ويحتوي على موقع تقييد Eagl وموقع تقييد ApaLI في 5' و 3' ينتهي، على التوالي (انظر الخطوة 1.1.1).
- تصميم وبناء بلازميد RBP
ملاحظة: الأحماض الأمينية وتسلسل النيوكليوتيد اتّباعاً لبروتينات المعطف المستخدمة في هذه الدراسة مدرجة في الجدول2.- طلب تسلسل RBP المطلوبة تفتقر إلى codon وقف كجين مصغر dsDNA أمر مخصص تفتقر إلى codon وقف مع مواقع تقييد في نهايات (الشكل 1).
- استنساخ RBP اختبار تفتقر إلى codon وقف المصب على الفور من المروج اللاإحتياج والمنبع من بروتين الفلورسنت تفتقر إلى codon بداية (الشكل1)،على غرار الخطوات 1.2.2-1.2.4. تأكد من أن بلازميد RBP يحتوي على جين مقاومة المضادات الحيوية مختلفة من بلازميد الموقع ملزمة.
- تحديد التحولات الإيجابية عن طريق تسلسل Sanger، على غرار الخطوة 1-2-5 (انظر الجدول 1 للاطلاع على التسلسلات التمهيدية).
- اختيار واحد تحويل إيجابي وجعلها كيميائيا المختصة25. تخزين كما بلازميدات نقية الجلسرين في -20 درجة مئوية ومخزونات الجلسرين من سلالات البكتيرية24 في -80 درجة مئوية في لوحات 96 جيدا.
- تحويل بلازميدات الموقع الملزم (من الخطوة 1.2.6) المخزنة في لوحات 96-well إلى خلايا بكتيرية مختصة كيميائيا تحتوي بالفعل على بلازميد RBP-mCerulean21. لتوفير الوقت، بدلا من طلاء الخلايا على أطباق بيتري، لوحة لهم باستخدام pipettor 8 قناة على لوحات 8 حارة تحتوي على لوريا بيرتاني (LB)26 أجار مع المضادات الحيوية ذات الصلة (كان وأمبير). المستعمرات يجب أن تظهر في 16 ح.
- حدد مستعمرة واحدة لكل تحويل مزدوج وتنمو بين عشية وضحاها في وسط LB مع المضادات الحيوية ذات الصلة (كان وأمبير) وتخزينها كما أسهم الجلسرين24 في -80 درجة مئوية في لوحات 96 جيدا.
2. إعداد التجربة
ملاحظة: تم تنفيذ البروتوكول المعروض هنا باستخدام نظام روبوتي للتعامل مع السوائل مع حاضنة وقارئ لوحة. وقد أُجري كل قياس لـ 24 تركيزات محفزة، مع نسختين مكررتين لكل سلالة + تركيبة محفزة. وباستخدام هذا النظام الروبوتي، جُمعت بيانات عن 16 سلالات في اليوم مع 24 تركيزات محفزة. ومع ذلك، إذا كان مثل هذا الجهاز غير متوفر، أو إذا كان عدد أقل من التجارب اللازمة، يمكن أن يتم ذلك بسهولة باليد باستخدام 8 قنوات متعددة الماصة وتكييف البروتوكول وفقا لذلك. فعلى سبيل المثال، تم الحصول على نتائج أولية لأربع سلالات في اليوم مع 12 تركيز محفز وأربع نقاط زمنية بهذه الطريقة.
- إعداد، مقدما، 1 لتر من المخزن المؤقت اختبار حيوي (BA) عن طريق خلط 0.5 غرام من التربتون، 0.3 مل من الجلسرين، 5.8 غرام من حمض الهيدروكلوريك، 50 مل من 1 م غ سو4،1 مل من 10X الفوسفات المخزنة المالحة (PBS) العازلة pH 7.4، و 950 مل من الماء المقطر المزدوج (DDW). الأوتوكلاف أو معقمة تصفية المخزن المؤقت مكتبة الإسكندرية.
- تنمو سلالات مزدوجة التحويل في 37 درجة مئوية و 250 دورة في الدقيقة تهتز في 1.5 مل رطل مع المضادات الحيوية المناسبة (كانامايسين في تركيز نهائي من 25 ميكروغرام / مل وampicillin في تركيز نهائي من 100 ميكروغرام / مل)، في لوحات 48 جيدا، على مدى فترة 18 ساعة (بين عشية وضحاها).
-
في الصباح، قم بالاستعدادات التالية.
- لوحة محفز. في لوحة نظيفة 96 جيدا، وإعداد الآبار مع المتوسطة شبه الفقيرة (SPM) تتكون من 95٪ BA و 5٪ LB26 في الحاضنة في 37 درجة مئوية. عدد الآبار يتوافق مع العدد المطلوب من تركيزات الحث. إضافة C4-HSL إلى الآبار في لوحة الحث التي سوف تحتوي على أعلى تركيز محفز (218 nM).
- برنامج الروبوت لتخفيف متوسط تسلسليا من كل من الآبار أعلى تركيز في 23 تركيزات أقل تتراوح بين 0 إلى 218 nM. يجب أن يكون حجم كل تخفيف محفز كافية لجميع السلالات (بما في ذلك التكرارات).
- في حين يجري إعداد تخفيف الحث، دافئ 180 درجة مئوية من SPM في الحاضنة في 37 درجة مئوية، في لوحات 96 جيدا.
- تخفيف سلالات بين عشية وضحاها من الخطوة 2.2 بعامل من 100 بواسطة التخفيف التسلسلي: تخفيف أولا بعامل من 10 عن طريق خلط 100 ميكرولتر من البكتيريا مع 900 ميكرولتر من SPM في لوحات 48 جيدا، ومن ثم تخفيف مرة أخرى بعامل 10 عن طريق أخذ 20 ميكرولتر من الحل المخفف في 180 ميكرولتر من SPM قبل تسخينها، في لوحات 96 جيدا مناسبة لقياسات الفلورسنت.
- إضافة الحث المخفف من لوحة الحث إلى لوحات 96 جيدا مع سلالات مخففة وفقا لتركيزات النهائي.
- هز لوحات 96 جيدا في 37 درجة مئوية لمدة 6 ساعة، في حين أخذ قياسات الكثافة البصرية في 595 نانومتر (OD595)،mCherry (560 نانومتر/ 612 نانومتر) ومسيرولانس (460 نانومتر/ 510 نانومتر) الفلورة عن طريق قارئ لوحة كل 30 دقيقة. لأغراض التطبيع، قياس نمو SMP مع عدم إضافة الخلايا.
3- التحليل الأولي للنتائج
- لكل يوم من أيام التجربة، اختر فاصل زمني للنمو اللوغاريتمي وفقا لمنحنيات النمو المقاسة، بين مرحلة النمو الخطي والثابتة (T0،Tfinal). خذ حوالي 6-8 نقاط زمنية، مع تجاهل القياسات الأولى والأخيرة لتجنب الخطأ الناتج عن عدم دقة الكشف عن النمو الأسي (انظر الشكل 2A،اللوحة العليا).
ملاحظة: تجاهل السلالات التي تظهر منحنيات النمو غير طبيعية أو سلالات حيث لا يمكن الكشف عن مرحلة النمو اللوغاريتمي وتكرار التجربة. -
حساب متوسط الفلورة تطبيع من mCerulean ومعدل إنتاج mCherry، من البيانات الخام لكل من mCerulean وmCherry الفلورة لكل تركيز محفز (الشكل2A).
- حساب mCerulean التسوية كما يلي:
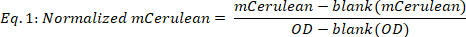
حيث فارغة (mCerulean) هو مستوى mCerulean [a.u.] للمتوسط فقط، فارغة (OD) هو الكثافة البصرية للمتوسط فقط، وmCerulean وOD هي الفلورة mCerulean وقيم الكثافة البصرية، على التوالي. - متوسط mCerulean على نقاط زمنية مختلفة (الشكل2B،أعلى اثنين من لوحات) على النحو التالي:
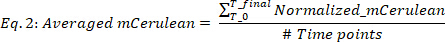
حيث #Time نقطة هو عدد نقاط البيانات الزمنية التي تؤخذ في الاعتبار، T0 هو الوقت الذي تبدأ فيه مرحلة النمو الأسي، وTالنهائي هو الوقت الذي تنتهي فيه مرحلة النمو الأسي. - حساب معدل mCherry من الإنتاج (الشكل2B،أسفل اثنين من لوحات) على النحو التالي:
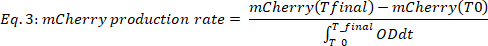
حيث mCherry(t) هو مستوى mCherry [a.u.] في الوقت t، OD هو قيمة الكثافة البصرية، T0 هو الوقت الذي تبدأ فيه مرحلة النمو الأسي، وTالنهائي هو الوقت الذي تنتهي فيه مرحلة النمو الأسي.
- حساب mCerulean التسوية كما يلي:
- وأخيرا، رسم معدل mCherry من الإنتاج كدالة mCerulean، وخلق منحنيات استجابة الجرعة كدالة للفلورة الانصهار RBP-mCerulean (الشكل2C). مثل هذه المؤامرات تمثل إنتاج الجين مراسل كدالة لوجود RBP في الخلية.
4. جرعة استجابة وظيفة تركيب الروتينية وKRBP استخراج
- تحت افتراض أن معدل الريبوسم للترجمة مع ربط RBP ثابت، نموذج معدل إنتاج mCherry على النحو التالي (انظر الشكل 2D،الخط الأخضر):

حيث [x] هو متوسط الفلورة mCerulean المعدلة حسب مكافئ 2، معدل إنتاج mCherry هو القيمة المحسوبة وفقا لEq. 3، KRBP هو التقارب ملزمة نسبيا [a.u.]، Kغير منضم هو معدل الريبوسوسوم من الترجمة مع RBP غير منضم، ن هو عامل التعاون، وC هو الفلورة الأساسية [a.u.]. تم العثور على C, n, Kunbound,و KRBP عن طريق تركيب بيانات معدل إنتاج mCherry إلى النموذج (Eq. 4). - باستخدام برنامج تحليل البيانات، إجراء إجراء مناسب على قطع الأراضي التي تصور معدل إنتاج mCherry كدالة لمتوسط mCerulean (الخطوة 3.3)، واستخراج المعلمات صالح وفقا للصيغة في Eq. 4.
ملاحظة: يتم أخذ النتائج المناسبة فقط مع R2 > 0.6 في الاعتبار. بالنسبة لتلك النوبات، يكون الخطأ في KRBP في الغالب في نطاق 0.5% إلى 20% من قيمK RBP، لفترة ثقة 0.67، في حين يمكن أيضًا التحقق من أولئك الذين يعانون من خطأRBP K أعلى عن طريق العين. - تطبيع قيمK RBP حسب القيمة القصوى لكل من متوسط mCerulean لكل وظيفة الجرعة استجابة.
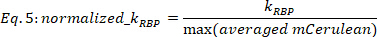
حيث KRBP في [a.u.] هي القيمة المستخرجة من إجراء التركيب في Eq. 4، والحد الأقصى (متوسط mCerulean) هو متوسط أقصى إشارة mCerulean [a.u] لوحظ للسلالة الحالية.
ملاحظة: وييسر التطبيع المقارنة الصحيحة بين الأثر التنظيمي عبر السلالات عن طريق القضاء على الاعتماد على مستويات التعبير القصوى الخاصة بالممارسات التجارية التقييدية.
النتائج
تستخدم الطريقة المعروضة المنافسة بين RBP والريبوسوم لربط جزيءmRNA (الشكل 1). وتنعكس هذه المنافسة من خلال انخفاض مستويات mCherry كدالة لزيادة إنتاج RBP-mCerulean، وذلك بسبب زيادة تركيزات المحفز. في حالة زيادة الفلورة mCerulean، مع عدم وجود تغييرات كبيرة في mCherry، يتم استنتاج...
Discussion
الطريقة الموصوفة في هذه المقالة يسهل القياس الكمي في الجسم الحي من تقارب ربط RBP-RNA في خلايا القولونية E. والبروتوكول سهل نسبيا ويمكن إجراؤه دون استخدام آلية متطورة، وتحليل البيانات واضح. وعلاوة على ذلك، تُنتج النتائج على الفور، دون وقت الانتظار الطويل نسبيا المرتبط بنتائج الجيل التال?...
Disclosures
وليس لدى أصحاب البلاغ ما يكشفون عنه.
Acknowledgements
وقد تلقى هذا المشروع تمويلا ً من برنامج I-CORE التابع للجنة التخطيط والميزنة ومؤسسة العلوم الإسرائيلية (المنحة رقم 152/11)، والمنحة رقم ماري كوري لإعادة الإدماج. PCIG11-GA- 2012-321675، ومن برنامج أبحاث وابتكار أفق 2020 التابع للاتحاد الأوروبي بموجب اتفاقية المنحة رقم 664918 - MRG-Grammar.
Materials
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
References
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved