需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
定量细菌中蛋白质-RNA结合的测定
摘要
在这种方法中,我们使用在细菌细胞中的简单、实时的、报告学测定来量化RNA结合蛋白(RBPs)与共和结合位点的结合亲和力。测定基于对报告者基因的抑制。
摘要
在蛋白质转化的起始步骤中,核糖体与mRNA的起始区域结合。通过将RNA结合蛋白(RBP)与mRNA的起始区域结合,可以阻断翻译启动,从而干扰核糖体结合。在所提出的方法中,我们利用这种阻塞现象来量化限制性商业惯例与其共性和非共性绑定位点的绑定亲和力。为此,我们在报告器 mRNA 的起始区域插入一个测试绑定位点,并诱导测试 RBP 的表达。在RBP-RNA结合的情况下,我们观察到作为RBP浓度函数的对报告者表达的抑制。在绑定站点和 RBP 之间没有亲和力或非常低的亲和力的情况下,没有观察到明显的抑制。该方法在活细菌细胞中进行,不需要昂贵或精密的机械。它可用于量化和比较细菌中功能的不同限制性商业惯例与一组设计结合位点之间的结合亲和力。此方法可能不适合具有高结构复杂性的绑定站点。这是因为在没有RBP的情况下,复杂的mRNA结构可能抑制核糖体启动,这将导致基底报告者基因表达降低,因此对RBP结合的观察量较低。
引言
近几十年来,基于RNA结合蛋白(RBP)的转录后调节,特别是RPS和RNA之间相互作用的表征,得到了广泛的研究。在来自限制性商业惯例的细菌中,有许多转化降调节的例子,它们抑制或直接与核糖体结合1、2、3竞争。在合成生物学领域,RBP-RNA相互作用正在成为设计基于转录基因电路4,5的重要工具。因此,在细胞环境中对这种RBP-RNA相互作用的表征需求增加。
研究蛋白质-RNA相互作用的最常见方法是电泳移动移位测定(EMSA)6,仅限于体外设置,以及各种下拉测定7,包括CLIP方法8,9.虽然这些方法能够发现脱新RNA结合位点,但它们存在诸如劳动密集型协议和昂贵的深度测序反应等缺点,并且可能需要一种特定的抗体来进行RBP下拉。由于RNA对其环境的易感性,许多因素会影响RBP-RNA相互作用,强调在细胞环境中对RBP-RNA结合进行探究的重要性。例如,我们和其他人已经证明体内RNA结构与体外RNA结构有显著差异10,11。
根据先前研究12的方法,我们最近演示了10,当为来自噬菌体GA 13、MS2 14、PP715和Q+16的细胞内大黄蜂RBPs放置预先设计的结合位点时,翻译启动区一位记者mRNA,记者的表情被强烈压制。基于这种抑制现象,我们提出了一种相对简单和定量的方法,以测量RBPs与其在体内的相应RNA结合位点之间的亲和力。
研究方案
1. 系统准备
- 结合位点质粒的设计
- 如图1所示,设计装订位盒。每个微基因包含以下部分(5'至3'):Eagl限制位点,[40基端的kanamycin(Kan)抗性基因,pLac-Ara启动子,核糖体结合位点(RBS),MCherry基因的AUG,一个间隔(+),一个RBP结合位点,5'端的80个碱基mCherry基因和ApaLI限制位。
注:为了提高测定的成功率,为每个装订位点设计三个结合位盒,垫块至少由一个、两个和三个碱基组成。有关进一步指南,请参阅代表性结果部分。
- 如图1所示,设计装订位盒。每个微基因包含以下部分(5'至3'):Eagl限制位点,[40基端的kanamycin(Kan)抗性基因,pLac-Ara启动子,核糖体结合位点(RBS),MCherry基因的AUG,一个间隔(+),一个RBP结合位点,5'端的80个碱基mCherry基因和ApaLI限制位。
- 结合位点质粒的克隆
- 将结合位点盒订购为双链DNA(dsDNA)小基因。每个微型基因长 ±500 bp,并分别包含 Eagl 限制站点和 ApaLI 限制站点,分别位于 5' 和 3' 端(请参阅步骤 1.1.1)。
注:在这个实验中,对一半卡那霉素基因的微型基因进行了排序,以方便对阳性菌落进行筛查。然而,吉布森组装17也适合在这里,在这种情况下,结合位点可以订购为两个较短的互补单链DNA寡聚物。 - 通过限制协议18,双消化微基因和目标载体,用Eagl-HF和ApaLI,柱纯化19。
- 利盖特消化的微型基因到结合位位主干,包含其余的mCherry报告基因,终结者,和卡纳霉素抗性基因20。
- 将结扎溶液转化为大肠杆菌TOP10细胞21。
- 通过桑格测序识别阳性转化剂。
- 在感兴趣的区域上游设计引基 100 基(有关引信序列,请参阅表1)。
- 迷你准备几个细菌菌落22。
- 在80纳克/μL浓度下制备5 mM的引液溶液和10μL的DNA。
- 将两个解决方案发送到一个方便的设施,为桑格测序23。
- 储存纯化质粒在-20°C,细菌菌株作为甘油库存24,均以96井格式。然后,DNA将用于转化为含有四个融合-RBP质粒之一的大肠杆菌TOP10细胞(参见步骤1.3.5)。
- 将结合位点盒订购为双链DNA(dsDNA)小基因。每个微型基因长 ±500 bp,并分别包含 Eagl 限制站点和 ApaLI 限制站点,分别位于 5' 和 3' 端(请参阅步骤 1.1.1)。
- RBP质粒的设计与建造
注:本研究中使用的涂层蛋白的氨基酸和核苷酸序列列在表2中。- 订购所需的RBP序列,缺少停止科顿作为一个自定义的dsDNA微型基因缺乏一个停止科顿与限制位点在末端(图1)。
- 克隆测试的RBP缺乏一个停止的康顿,立即下游的诱导启动子和荧光蛋白的上游缺乏启动科顿(图1),类似于步骤1.2.2-1.2.4。确保RBP质粒含有不同于结合位点质粒的抗生素耐药性基因。
- 通过 Sanger 测序识别正转化剂,类似于步骤 1.2.5(有关引物序列,请参见表1)。
- 选择一个正转化剂,使其具有化学能力25。在-20°C储存为甘油纯化质粒,在96孔板中储存细菌菌株24的甘油库存在-80°C。
- 将储存在96孔板中的结合位位质粒(从步骤1.2.6)转化为具有化学能力的细菌细胞,这些细胞已经含有RBP-mCerulean质粒21。为了节省时间,而不是在培养皿上电镀细胞,在含有Luria-Bertani(LB)26琼脂(Kan和Amp)的8通道板上用8通道移管器进行板。殖民地应在16小时出现。
- 为每个双转化剂选择一个菌群,并在LB介质中使用相关抗生素(Kan和Amp)一夜生长,并在96孔板中储存为甘油库存24,温度为-80°C。
2. 实验设置
注:这里提出的协议是使用液体处理机器人系统与孵化器和板读取器相结合。每次测量对24个诱导剂浓度进行,每个菌株和诱导剂组合有两个重复。利用这个机器人系统,收集了每天16个菌株的数据,其中24个诱导剂浓度。但是,如果此类设备不可用,或者需要较少的实验,则可以轻松地使用 8 通道多移液器手动完成这些实验,并相应地调整协议。例如,以这种方法获得每天4个菌株的初步结果,其中12个诱导剂浓度和4个时间点。
- 通过混合 0.5 克试金石、0.3 mL、5.8 g NaCl、50 mL 1 M MgSO 4、1m L 10x 磷酸盐缓冲盐 (PBS) 缓冲液 pH 7.4 和 950 mL 双蒸馏水 (DDW), 提前制备 1 L 生物测定缓冲液 (BA)。高压灭菌或无菌过滤BA缓冲液。
- 在48孔板中,在48孔板中,在18孔板(过夜)内,用适当的抗生素(最终浓度为25微克/mL的卡那霉素和最终浓度为100微克/mL的甘霉素),在37°C和250rpm下在1.5 mL LB下生长双转化菌株。
-
早上,做以下准备。
- 诱导板。在干净的 96 孔板中,在 37°C 的培养箱中制备半差介质 (SPM) 的井,在培养箱中包括 95% BA 和 5% LB26。井数对应于所需的诱导剂浓度数。将 C4-HSL 添加到诱导板中的孔中,该孔将含有最高的诱导剂浓度 (218 nM)。
- 将机器人从每个浓度最高的井中连续稀释到 23 个浓度从 0 到 218 nM 的低浓度。每个诱导剂稀释的体积应足以满足所有菌株(包括重复菌株)。
- 在制备诱导稀释剂时,在37°C的培养箱中,在96孔板中加热180μL的SPM。
- 通过连续稀释将步骤 2.2 的隔夜菌株稀释 100 倍:首先将 100 μL 的细菌与 900 μL 的 SPM 混合在 48 孔板中,再稀释 10 倍,从稀释溶液中加入 20 μL180 μL 的预加热SPM,适用于96孔板,适用于荧光测量。
- 将稀释的诱导剂从诱导板添加到96孔板中,并根据最终浓度将稀释菌株加入。
- 在 37°C 下摇动 96 孔板 6 小时,同时每 30 分钟通过板读取器测量 595 nm (OD 595)、mCherry (560 nm/612 nm) 和 mCerulean (460 nm/510 nm) 荧光。出于规范化目的,测量 SMP 的生长,不添加任何单元格。
3. 初步结果分析
- 对于每天的实验,根据测量的生长曲线,在线性生长阶段和静止(T 0,T最终)之间选择对数增长的时间间隔。以大约 6⁄8 个时间点为例,同时放弃第一个和最后一个测量值,以避免因指数增长检测不准确而产生的错误(参见图 2A,顶部面板)。
注:丢弃显示异常生长曲线或菌株的菌株,其中无法检测到对数生长阶段,并重复实验。 -
根据每种诱导剂浓度的mCerulean和mCherry荧光的原始数据计算mCerulean的平均正定荧光和mCherry的产生率(图2A)。
- 计算规范化的 mCerulean,如下所示:
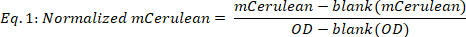
其中空白(mCerulean)是仅介质的mCerulean水平[a.u.],空白(OD)是介质的光学密度,mCerulean和OD分别是mCerulean荧光和光密度值。 - 不同时间点的平均 mCerulean(图 2B,前两个面板),如下所示:
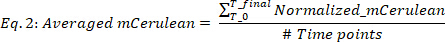
其中#Time点是考虑的数据时间点数,T0是指数增长阶段开始的时间,T最终点是指数增长阶段结束的时间。 - 计算 mCherry 的生产率(图 2B,底部两个面板),如下所示:
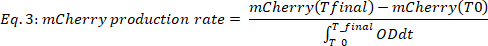
其中 mCherry(t) 是时间 t 的 mCherry 级别 [a.u.],OD 是光学密度值,T0是指数增长阶段开始的时间,T最终是指数增长阶段结束的时间。
- 计算规范化的 mCerulean,如下所示:
- 最后,将mCherry的产生速率绘制为mCerulean的函数,创建剂量反应曲线作为RBP-mCerulean融合荧光的函数(图2C)。这些图表示报告基因的产生,作为细胞中RBP存在的函数。
4. 剂量响应功能拟合例程和 KRBP提取
- 假设具有 RBP 绑定的核糖体转换速率是恒定的,则对 mCherry 生产速率进行如下建模(参见图 2D,绿线):

其中[x]是根据Eq.2计算的标准化平均mCerulean荧光,mCherry生产速率是根据Eq.3计算的值,KRBP是相对结合的亲和力[a.u.],K未绑定是核糖体速率与RBP的转换未绑定,n是协同系数,C是基荧光[a.u.]。通过将 mCherry 生产速率数据拟合到模型中(Eq. 4),可以找到 C、n、K未绑定和 KRBP。 - 使用数据分析软件,对将 mCherry 生产率描述为平均 mCerulean 函数的绘图执行拟合过程(步骤 3.3),并根据 Eq. 4 中的公式提取拟合参数。
注:只考虑具有 R2 > 0.6 的拟合结果。对于这些拟合,KRBP误差主要在 KRBP值的 0.5% 到 20% 的范围内,为 0.67 置信区间,而 KRBP误差较高的误差也可以通过眼睛进行验证。 - 按每个剂量反应函数的平均值 mCerulean 的相应最大值对 KRBP值进行标准化。
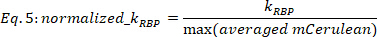
其中 KRBP在 [a.u.] 是从 Eq. 4 中的拟合过程中提取的值,最大值(平均 mCeran)是为当前应变观测到的最大平均 mCeran 信号 [a.u]。
注:规范化通过消除对特定最大 RBP 表达水平的依赖,促进不同菌株调节效果的正确比较。
结果
所提出的方法利用RBP和核糖体之间的竞争,与mRNA分子结合(图1)。这种竞争反映在由于诱导剂浓度增加而降低mCherry水平,作为RBP-mCeran产量增加的函数。在增加mCerulean荧光的情况下,mCherry没有显著变化,可以推断出缺乏RBP结合。图 2描述了正应变和负应变的代表性结果。在图 2A中,OD、mCherry 和 mCerulean 通道在四个?...
讨论
本文描述的方法有助于对大肠杆菌细胞中的RBP-RNA结合亲和力进行定量的体内测量。该协议相对简单,无需使用精密机械即可进行,数据分析也非常简单。此外,结果会立即产生,没有与下一代测序 (NGS) 结果相关的相对长的等待时间。
这种方法的一个限制是,它只在细菌细胞中工作。然而,先前的研究12已经证明在哺乳动物细胞中使用类似的方法对L7AE RBP具有?...
披露声明
作者没有什么可透露的。
致谢
该项目得到了规划和预算委员会和以色列科学基金会(第152/11号赠款)的I-CORE方案的资助,玛丽·居里重返社会赠款号为第152/11号赠款。PCIG11-GA- 2012-321675,以及欧盟地平线2020年研究与创新计划,根据授权协议664918 - MRG-Grammar。
材料
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8- lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| glycerol | BIO LAB | 071205 | |
| incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| ligase | NEB | B0202S | |
| liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| SODIUM CHLORIDE (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
参考文献
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。