É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Um ensaio para quantificar a ligação de RNA de proteína em bactérias
Neste Artigo
Resumo
Neste método, nós quantificamos a afinidade obrigatória de proteínas de ligação do RNA (rbps) aos locais de ligação cognato e non-cognato usando um simples, vivo, ensaio do repórter em pilhas bacterianas. O ensaio é baseado na repressão de um gene repórter.
Resumo
No passo de iniciação da tradução da proteína, o Ribossome liga-se à região de iniciação do mRNA. A iniciação da tradução pode ser obstruída pela ligação de uma proteína obrigatória do RNA (RBP) à região de iniciação do mRNA, que interfere com o emperramento ribossoma. No método apresentado, utilizamos este fenômeno de bloqueio para quantificar a afinidade vinculativa de RBPs aos seus locais de ligação cognato e não cognato. Para fazer isso, inserimos um site de vinculação de teste na região de iniciação de um mRNA de repórter e induzem a expressão do teste RBP. No caso da Associação RBP-RNA, observou-se uma repressão sigmoidal da expressão repórter em função da concentração de RBP. No caso de não afinidade ou de afinidade muito baixa entre o local de ligação e a RBP, não foi observada nenhuma repressão significativa. O método é realizado em células bacterianas ao vivo, e não requer maquinaria dispendiosa ou sofisticada. É útil para quantificar e comparar entre as afinidades de ligação de RBPs diferentes que são funcionais nas bactérias a um jogo de locais de ligação projetados. Este método pode ser inadequado para sites de ligação com alta complexidade estrutural. Isto é devido à possibilidade da repressão da iniciação ribosomal pela estrutura complexa do mRNA na ausência de RBP, que conduziria à expressão de gene mais baixa do repórter basal, e assim à repressão menos-Observable do repórter em cima da ligação de RBP.
Introdução
A regulação pós-transcricional de proteína de ligação de RNA (RBP), especificamente a caracterização da interação entre RBPs e RNA, tem sido extensivamente estudada nas últimas décadas. Existem vários exemplos de baixo-regulação translacional em bactérias originárias da inibição do rbps, ou diretamente competindo com, ligação Ribossome1,2,3. No campo da biologia sintética, as interações RBP-RNA estão surgindo como uma ferramenta significativa para o desenho de circuitos genéticos baseados em transcrição4,5. Portanto, há um aumento na demanda para caracterização dessas interações RBP-RNA em um contexto celular.
Os métodos mais comuns para o estudo das interações proteína-RNA são o ensaio eletroforético de deslocamento de mobilidade (EMSA)6, que é limitado a configurações in vitro, e vários ensaios pull-down7, incluindo o método de clipe8,9 . Quando tais métodos permiterem a descoberta de locais de ligação do RNA de de novo, sofrem dos inconvenientes tais como protocolos labor-intensivos e de reações de seqüenciamento profundas caras e podem exigir um anticorpo específico para a tração-para baixo de RBP. Devido à natureza suscetível do RNA ao seu ambiente, muitos fatores podem afetar as interações RBP-RNA, enfatizando a importância de interrogar a ligação RBP-RNA no contexto celular. Por exemplo, nós e outros temos demonstrado diferenças significativas entre as estruturas de RNA in vivo e in vitro10,11.
Baseado na aproximação de um estudo precedente12, Nós demonstramos recentemente10 que ao coloc locais obrigatórios pre-projetados para o capsídeo rbps dos bacteriófagos GA13, MS214, PP715, e qβ16 no região de iniciação de tradução de um repórter mRNA, a expressão do repórter é fortemente reprimido. Nós apresentamos um método relativamente simples e quantitativo, baseado neste fenômeno da repressão, para medir a afinidade entre RBPs e seu local de ligação RNA correspondentes in vivo.
Protocolo
1. preparação do sistema
- Projeto de plasmídeos do ligação-local
- Projete a gaveta do local de ligação como representado na Figura 1. Cada que contém as seguintes peças (5 ' a 3 '): Eagl local de restrição, ~ 40 bases do 5 ' fim do canamicina (Kan) gene de resistência, pLac-ara promotor, Ribossoma Binding site (RBS), Aug do gene mcherry, um espaçador (δ), um local de ligação RBP, 80 bases do 5 ' final do gene mCherry, e um local de restrição ApaLI.
Nota: Para aumentar a taxa de sucesso do ensaio, projetar três gavetas de encadernação para cada local de ligação, com espaçadores consistindo de pelo menos um, dois e três bases. Consulte a seção resultados representativos para obter mais diretrizes.
- Projete a gaveta do local de ligação como representado na Figura 1. Cada que contém as seguintes peças (5 ' a 3 '): Eagl local de restrição, ~ 40 bases do 5 ' fim do canamicina (Kan) gene de resistência, pLac-ara promotor, Ribossoma Binding site (RBS), Aug do gene mcherry, um espaçador (δ), um local de ligação RBP, 80 bases do 5 ' final do gene mCherry, e um local de restrição ApaLI.
- Clonagem de plasmídeos do local obrigatório
- Encomende as fitas do site de encadernação como minigenos de DNA de duas cordas (dsDNA). Cada que é ~ 500 BP longo e contem um local da limitação de Eagl e um local da limitação de apali nos 5 ' e 3 ' extremidades, respectivamente (veja a etapa 1.1.1).
Nota: Neste experimento, minigenes com metade do gene da kanamicina foram ordenados para facilitar a triagem para colônias positivas. Entretanto, a montagem17 de Gibson é igualmente apropriada aqui, neste caso o local obrigatório pode ser requisitado como dois oligos single-stranded complementares mais curtos do ADN. - Duplo-digerir ambos os mini-genes e o vetor alvo com Eagl-HF e apali pelo protocolo de restrição18, e coluna purificar19.
- Ligate os minigenes digeridos à espinha dorsal do emperramento-local que contem o descanso do gene do repórter de mcherry, do terminador, e de um gene20da resistência do canamicina.
- Transforme a solução de ligadura em células de Escherichia coli -21.
- Identifique os transformantes positivos através do sequenciamento de Sanger.
- Projetar uma cartilha 100 bases upstream para a região de interesse (ver tabela 1 para sequências de primer).
- Miniprep algumas colônias bacterianas22.
- Prepare 5 μL de uma solução de 5 mM da primeira demão e 10 μL de ADN a 80 ng/μL de concentração.
- Envie as duas soluções para uma instalação conveniente para o sequenciamento de Sanger23.
- Armazene plasmíos purificados a-20 ° c e cepas bacterianas como estoques de glicerol24, ambos no formato 96-well. O DNA será então usado para a transformação em células de E. coli ------------------------(ver passo 1.3.5)
- Encomende as fitas do site de encadernação como minigenos de DNA de duas cordas (dsDNA). Cada que é ~ 500 BP longo e contem um local da limitação de Eagl e um local da limitação de apali nos 5 ' e 3 ' extremidades, respectivamente (veja a etapa 1.1.1).
- Concepção e construção do plasmídeo RBP
Nota: As sequências de aminoácido e nucleotídeo das proteínas do revestimento utilizadas neste estudo estão listadas na tabela 2.- Encomende a sequência de RBP necessária sem um Codon de parada como um minigeno dsDNA ordenado encomenda, faltando um Codon de parada com locais de restrição nas extremidades (Figura 1).
- Clonar o RBP testado faltando um Codon de parada imediatamente a jusante de um promotor induzível e a montante de uma proteína fluorescente que falta um Codon do começo (Figura 1), similar às etapas 1.2.2-1.2.4. Certifique-se de que o plasmídeo RBP contenha um gene diferente da resistência antibiótica do que o plasmídeo do ligação-local.
- Identifique os transformantes positivos através do sequenciamento de Sanger, semelhante ao passo 1.2.5 (ver tabela 1 para sequências de primer).
- Escolha um transformante positivo e torne-o quimicamente-competente25. Armazene como plasmídos purificados de glicerol a-20 ° c e estoques de glicerol de cepas bacterianas24 at-80 ° c em placas 96-well.
- Transforme os plasmídeo do local de ligação (da etapa 1.2.6) armazenados em placas 96-well em pilhas bacterianas quimicamente-competentes que contêm já um plasmídeo RBP-mcerulean21. Para economizar tempo, em vez de galvanizar as células em placas de Petri, placa-los usando um pipetador de 8 canais em 8-pistas de pista contendo Luria-Bertani (LB)26 Agar com antibióticos relevantes (Kan e amp). As colônias devem aparecer em 16 h.
- Selecione uma única colônia para cada transformante duplo e cresça durante a noite no meio LB com os antibióticos relevantes (Kan e amp) e armazene como estoques de glicerol24 at-80 ° c em placas 96-well.
2. configuração do experimento
Nota: O protocolo aqui apresentado foi realizado por meio de um sistema robótico de manuseio de líquidos em combinação com uma incubadora e um leitor de placas. Cada medida foi realizada para 24 concentrações de indutor, com duas duplicatas para cada combinação de estirpe + indutor. Utilizando este sistema robótico, foram coletados dados para 16 cepas por dia com 24 concentrações de indutor. No entanto, se tal dispositivo não estiver disponível, ou se forem necessários menos experimentos, estes podem ser facilmente feitos manualmente usando uma multipipeta de 8 canais e adaptando o protocolo de acordo. Por exemplo, resultados preliminares para quatro cepas por dia com 12 concentrações de indutor e quatro tempos-pontos foram adquiridos dessa maneira.
- Prepare, com antecedência, 1 L de tampão de bioensaio (BA) misturando 0,5 g de triptona, 0,3 mL de glicerol, 5,8 g de NaCl, 50 mL de 1 M MgSO4, 1 ml de tampão fisiológico de fosfato (PBS) de 10x tamponado e 7,4 ml de água destilada dupla (DDW). Autoclave ou filtro estéril o tampão BA.
- Cresça as estirpes do duplo-Transformant em 37 ° c e 250 rpm que agitam em 1,5 mL LB com os antibióticos apropriados (kanamycin em uma concentração final de 25 μg/mL e Ampicillin em uma concentração final de 100 μg/mL), em placas de 48-well, durante um período de 18 h (durante a noite).
-
De manhã, faça os seguintes preparativos.
- Placa do indutor. Em uma placa limpa 96-well, prepare poços com meio semipobre (SPM) que consiste em 95% BA e em 5% LB26 na incubadora em 37 ° c. O número de poços corresponde ao número desejado de concentrações de indutor. Adicione C4-HSL aos poços na placa do indutor que conterá a concentração a mais elevada do indutor (218 nanômetro).
- Programe o robô para diluir serialmente o meio de cada um dos poços de maior concentração em 23 concentrações inferiores variando de 0 a 218 nM. O volume de cada diluição do indutor deve ser suficiente para todas as estirpes (incluindo duplicatas).
- Enquanto as diluições do indutor estão sendo preparadas, aqueça 180 μL de SPM na incubadora em 37 ° c, em placas de 96 poços.
- Diluir as cepas da noite da etapa 2,2 por um fator de 100 por diluições seriais: primeiro diluir por um fator de 10, misturando 100 μL de bactérias com 900 μL de SPM em placas de 48 poços, e depois diluir novamente por um fator de 10, tomando 20 μL da solução diluída em 180 μL de SPM pré-aquecido, em placas de 96 poços adequadas para medições fluorescentes.
- Adicione o indutor diluído da placa do indutor às placas 96-well com as tensões diluídas de acordo com as concentrações finais.
- Agite as placas 96-well em 37 ° c por 6 h, ao tomar medidas da densidade ótica em 595 nanômetro (OD595), em mcherry (560 nm/612 nanômetro) e em fluorescência de mCerulean (460 nm/510 nanômetro) através de um leitor da placa cada 30 minutos. Para fins de normalização, medir o crescimento de SMP sem células adicionadas.
3. análise preliminar dos resultados
- Para cada dia de experimento, escolha um intervalo de tempo de crescimento logarítmico de acordo com as curvas de crescimento medido, entre a fase de crescimento linear e a estacionária (T0, tfinal). Tome aproximadamente 6 − 8 pontos de tempo, ao descartar a primeira e a última medida para evitar o erro derivado da inexatidão da deteção exponencial do crescimento (veja Figura 2a, painel superior).
Nota: Descarte as cepas que mostram curvas de crescimento anormais ou cepas em que a fase de crescimento logarítmico não pôde ser detectada e repetia o experimento. -
Calcule a fluorescência normalizada média de mCerulean e a taxa de produção de mCherry, a partir dos dados brutos de fluorescência mCerulean e mCherry para cada concentração de indutor (Figura 2a).
- Calcule mCerulean normalizado como segue:
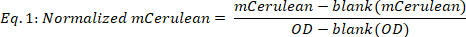
onde em branco (mCerulean) é o nível de mCerulean [u.a.] para o meio somente, o espaço em branco (OD) é a densidade ótica para o meio somente, e o mCerulean e o OD são os valores da fluorescência e da densidade ótica de mCerulean, respectivamente. - MCerulean médio sobre os pontos diferentes do tempo (Figura 2b, os dois painéis superiores) como segue:
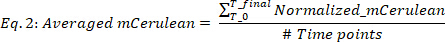
onde #Time pontos é o número de temporais dados tidos em conta, t0 é o tempo em que começa a fase de crescimento exponencial, e tfinal é o tempo em que termina a fase de crescimento exponencial. - Calcule a taxa de produção de mCherry (Figura 2b, dois painéis inferiores) da seguinte forma:
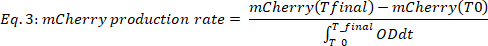
onde mCherry (t) é o nível de mCherry [u.a.] no tempo t, OD é o valor de densidade óptica, T0 é o tempo em que a fase de crescimento exponencial começa, e tfinal é o tempo em que a fase de crescimento exponencial termina.
- Calcule mCerulean normalizado como segue:
- Por fim, traçar a taxa de produção de mCherry em função de mCerulean, criando curvas de resposta de dose em função da fluorescência de fusão RBP-mCerulean (Figura 2C). Tais parcelas representam a produção do gene repórter em função da presença de RBP na célula.
4. função de resposta da dose rotina de encaixe e extração de KRBP
- a suposição que a taxa Ribossoma de tradução com o limite de RBP é constante, modela a taxa de produção de mcherry como segue (veja Figura 2D, linha verde):

onde [x] é a fluorescência mCerulean média normalizada calculada de acordo com EQ. 2, taxa de produção de mCherry é o valor calculado de acordo com EQ. 3, KRBP é a afinidade de ligação relativa [u.a.], knão acoplado é a taxa Ribossome de Tradução com a RBP não acoplada, n é o fator de cooperatividade, e C é a fluorescência de base [u.a.]. C, n, Knão acoplado, e kRBP são encontrados ajustando os dados da taxa de produção de mcherry ao modelo (EQ. 4). - Usando o software da análise de dados, conduza um procedimento apropriado nos lotes que descrevem a taxa de produção de mCherry em função da média mCerulean (etapa 3,3), e extraia os parâmetros do ajuste de acordo com a fórmula em EQ. 4.
Nota: Somente os resultados de encaixe com R2 > 0,6 são tomados em consideração. Para esses ajustes, o erro de KRBP é principalmente na faixa de 0,5% a 20% dos valores de kRBP , para um intervalo de confiança de 0,67, enquanto aqueles com maior erro de kRBP também podem ser verificados por olho. - Normalize os valores de KRBP pelo respectivo valor máximo de mCerulean média para cada função dose-resposta.
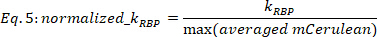
onde KRBP em [u.a.] é o valor extraído do procedimento de encaixe em EQ. 4, e Max (mcerulean média) é o sinal mcerulean de média máxima [a. u] observado para a tensão atual.
Nota: A normalização facilita a comparação correta do efeito regulatório entre as cepas, eliminando a dependência dos níveis máximos de expressão de RBP específicos.
Resultados
O método apresentado utiliza a competição entre uma RBP e o Ribossome para ligação à molécula de mRNA (Figura 1). Esta competição é refletida pela diminuição dos níveis de mCherry em função do aumento da produção de RBP-mCerulean, devido ao aumento das concentrações de indutor. No caso de aumento da fluorescência de mCerulean, sem alterações significativas em mCherry, a falta de ligação à RBP é deduzada. Os resultados representativos...
Discussão
O método descrito neste artigo facilita a medida in vivo quantitativa da afinidade obrigatória da RBP-RNA em pilhas de E. coli . O protocolo é relativamente fácil e pode ser conduzido sem o uso de maquinaria sofisticada, e a análise de dados é direta. Além disso, os resultados são produzidos imediatamente, sem o tempo de espera relativamente longo associado com os resultados de sequenciamento da próxima geração (NGS).
Uma limitação a este método é que trabalha somente e...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este projeto recebeu financiamento do programa I-CORE do Comitê de planejamento e orçamento e da Fundação de ciência de Israel (Grant no. 152/11), Marie Curie reintegration Grant no. PCIG11-GA-2012-321675, e do programa Horizonte 2020 de investigação e inovação da União Europeia, a Convenção de subvenção n. º 664918-MRG-gramática.
Materiais
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
Referências
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados