Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Un ensayo para cuantificar la unión de proteínas y ARN en bacterias
En este artículo
Resumen
En este método, cuantificamos la afinidad de unión de proteínas de unión al ARN (RBP) a sitios de unión cognados y no cognados utilizando un ensayo de reportero simple, vivo y en células bacterianas. El ensayo se basa en la represión de un gen reportero.
Resumen
En el paso de iniciación de la traducción proteica, el ribosoma se une a la región de iniciación del ARNm. La iniciación de la traducción se puede bloquear mediante la unión de una proteína de unión al ARN (RBP) a la región de iniciación del ARNm, lo que interfiere con la unión ribosoma. En el método presentado, utilizamos este fenómeno de bloqueo para cuantificar la afinidad de unión de los BBP con sus sitios de enlace cognados y no cognados. Para ello, insertamos un sitio de enlace de prueba en la región de iniciación de un ARNm de reportero e inducemos la expresión de la RBP de prueba. En el caso de la unión RBP-RNA, observamos una represión sigmoidal de la expresión del reportero en función de la concentración de RBP. En el caso de la no afinidad o la afinidad muy baja entre el sitio de enlace y la RBP, no se observó una represión significativa. El método se lleva a cabo en células bacterianas vivas, y no requiere maquinaria costosa o sofisticada. Es útil para cuantificar y comparar entre las afinidades de unión de diferentes PBP que son funcionales en bacterias a un conjunto de sitios de unión diseñados. Este método puede ser inapropiado para sitios de enlace con alta complejidad estructural. Esto se debe a la posibilidad de represión de la iniciación ribosomal por compleja estructura de ARNm en ausencia de RBP, lo que resultaría en una expresión genética de reportero basal más baja, y por lo tanto una represión de reportero menos observable sobre la vinculación de la RBP.
Introducción
La regulación post-transcripción basada en proteína de unión al ARN (RBP), caracterizada específicamente de la interacción entre los PBP y el ARN, se ha estudiado extensamente en las últimas décadas. Hay múltiples ejemplos de regulación descendente traslacional en bacterias originadas por PBP que inhiben, o compiten directamente con, la unión ribosoma1,2,3. En el campo de la biología sintética, las interacciones RBP-ARN están emergiendo como una herramienta significativa para el diseño de circuitos genéticos basados en transcripción4,5. Por lo tanto, hay un aumento en la demanda de caracterización de tales interacciones RBP-ARN en un contexto celular.
Los métodos más comunes para estudiar las interacciones proteína-ARN son el ensayo de cambio de movilidad electroforético (EMSA)6, que se limita a entornos in vitro, y varios ensayos desplegables7,incluido el método CLIP8,9 . Si bien estos métodos permiten el descubrimiento de sitios de enlace de ARN novo, sufren de inconvenientes tales como protocolos intensivos en mano de obra y costosas reacciones de secuenciación profunda y pueden requerir un anticuerpo específico para la extracción de RBP. Debido a la naturaleza susceptible del ARN a su entorno, muchos factores pueden afectar las interacciones RBP-ARN, haciendo hincapié en la importancia de interrogar la unión RBP-ARN en el contexto celular. Por ejemplo, nosotros y otros hemos demostrado diferencias significativas entre las estructuras de ARN in vivo e in vitro10,11.
Basándonos en el enfoque de un estudio anterior12, recientemente demostramos10 que al colocar sitios de unión prediseñados para los PBP cápside de los bacteriófagos GA13, MS214, PP715y Q-16 en el región de iniciación de traducción de un ARNm reportero, la expresión del reportero está fuertemente reprimida. Presentamos un método relativamente simple y cuantitativo, basado en este fenómeno de represión, para medir la afinidad entre los PBP y su correspondiente sitio de unión de ARN in vivo.
Protocolo
1. Preparación del sistema
- Diseño de plásmidos de sitio de encuadernación
- Diseñe el cassette del sitio de enlace como se muestra en la Figura1. Cada minigen contiene las siguientes partes (5' a 3'): sitio de restricción de Eagl, 40 bases del extremo de 5' del gen de resistencia de kanamicina (Kan), promotor pLac-Ara, sitio de unión ribosoma (RBS), AUG del gen mCherry, un espaciador ( ) , un sitio de unión RBP, 80 bases del extremo 5' del gen mCherry, y un sitio de restricción de ApaLI.
NOTA: Para aumentar la tasa de éxito del ensayo, diseñe tres casetes de sitio de enlace para cada sitio de enlace, con espaciadores que consisten en al menos una, dos y tres bases. Consulte la sección Resultados representativos para obtener más directrices.
- Diseñe el cassette del sitio de enlace como se muestra en la Figura1. Cada minigen contiene las siguientes partes (5' a 3'): sitio de restricción de Eagl, 40 bases del extremo de 5' del gen de resistencia de kanamicina (Kan), promotor pLac-Ara, sitio de unión ribosoma (RBS), AUG del gen mCherry, un espaciador ( ) , un sitio de unión RBP, 80 bases del extremo 5' del gen mCherry, y un sitio de restricción de ApaLI.
- Clonación de plásmidos de sitios de encuadernación
- Ordene los casetes del sitio de unión como minigenes de ADN de doble cadena (dsDNA). Cada minigene tiene 500 bp de largo y contiene un sitio de restricción de Eagl y un sitio de restricción ApaLI en los extremos de 5' y 3', respectivamente (ver paso 1.1.1).
NOTA: En este experimento, se ordenaron minigenes con la mitad del gen kanamicina para facilitar la detección de colonias positivas. Sin embargo, Gibson ensamblaje17 también es adecuado aquí, en cuyo caso el sitio de unión se puede pedir como dos oligos de ADN complementarios más cortos de una sola cadena. - Digerir dos veces tanto los minigenes como el vector de destino con Eagl-HF y ApaLI mediante el protocolo de restricción18,y purificar la columna19.
- Ligar los minigenes digeridos a la columna vertebral del sitio de unión que contiene el resto del gen reportero mCherry, terminador, y un gen de resistencia a la kanamicina20.
- Transformar la solución de ligación en Escherichia coli TOP10 células21.
- Identifique transformadores positivos a través de la secuenciación de Sanger.
- Diseñe una imprimación de 100 bases aguas arriba de la región de interés (véase el Cuadro 1 para las secuencias de imprimación).
- Miniprep algunas colonias bacterianas22.
- Preparar 5 ml de una solución de 5 mM de la imprimación y 10 ml del ADN a una concentración de 80 ng/L.
- Envíe las dos soluciones a una instalación conveniente para la secuenciación de Sanger23.
- Almacene los plásmidos purificados a -20 oC, y las cepas bacterianas como las existencias de glicerol24,ambas en el formato de 96 oC. El ADN se utilizará para la transformación en células De. coli TOP10 que contienen uno de los cuatro plásmidos de fusión-RBP (ver paso 1.3.5).
- Ordene los casetes del sitio de unión como minigenes de ADN de doble cadena (dsDNA). Cada minigene tiene 500 bp de largo y contiene un sitio de restricción de Eagl y un sitio de restricción ApaLI en los extremos de 5' y 3', respectivamente (ver paso 1.1.1).
- Diseño y construcción del plásmido RBP
NOTA: Las secuencias de aminoácidos y nucleótidos de las proteínas de capa utilizadas en este estudio se enumeran en la Tabla2.- Ordene la secuencia RBP requerida que carezca de un codón de parada como un minigénmino dsDNA ordenado a medida que carece de un codón de parada con sitios de restricción en los extremos (Figura1).
- Clonar la RBP probada que carece de un codón de parada inmediatamente aguas abajo de un promotor inducible y aguas arriba de una proteína fluorescente que carece de un codón de inicio (Figura1), similar a los pasos 1.2.2-1.2.4. Asegúrese de que el plásmido RBP contiene un gen de resistencia a los antibióticos diferente del plásmido del sitio de unión.
- Identifique transformadores positivos mediante la secuenciación de Sanger, similar al paso 1.2.5 (consulte la Tabla 1 para las secuencias de imprimación).
- Elija un transformador positivo y hacerlo químicamente competente25. Conservar como plásmidos purificados de glicerol a -20 oC y reservas de glicerol de cepas bacterianas24 a -80 oC en placas de 96 pocillos.
- Transformar los plásmidos del sitio de unión (del paso 1.2.6) almacenados en placas de 96 pocillos en células bacterianas químicamente competentes que ya contienen un plásmido RBP-mCerulean21. Para ahorrar tiempo, en lugar de enchapar las células en los platos de Petri, placarlos usando un pipeteador de 8 canales en placas de 8 carriles que contienen Luria-Bertani (LB)26 agar con antibióticos relevantes (Kan y Amp). Las colonias deben aparecer en 16 h.
- Seleccione una sola colonia para cada doble transformador y crezca durante la noche en medio LB con los antibióticos relevantes (Kan y Amp) y almacene como reservas de glicerol24 a -80 oC en placas de 96 pocillos.
2. Configuración del experimento
NOTA: El protocolo presentado aquí se realizó utilizando un sistema robótico de manipulación de líquidos en combinación con una incubadora y un lector de placas. Cada medición se llevó a cabo para 24 concentraciones de inductores, con dos duplicados para cada cepa + combinación de inductor. Usando este sistema robótico, se recogieron datos de 16 cepas por día con 24 concentraciones de inductores. Sin embargo, si un dispositivo de este tipo no está disponible, o si se realizan menos experimentos, estos se pueden hacer fácilmente a mano utilizando una multipipe de 8 canales y adaptando el protocolo en consecuencia. Por ejemplo, se adquirieron resultados preliminares para cuatro cepas al día con 12 concentraciones de inductores y cuatro puntos de tiempo de esta manera.
- Preparar, de antemano, 1 L de tampón de bioensayo (BA) mezclando 0,5 g de tripatona, 0,3 ml de glicerol, 5,8 g de NaCl, 50 ml de 1 M MgSO4, 1 ml de 10x de tampón de solución salina con fosfato (PBS) pH 7,4 y 950 ml de agua destilada doble (DDW). Autoclave o filtro estéril del búfer BA.
- Cultivar las cepas de doble transformación a 37 oC y 250 rpm agitando en 1,5 ml de LB con antibióticos adecuados (kanamicina a una concentración final de 25 g/ml y ampicilina a una concentración final de 100 ég/ml), en placas de 48 pocillos, durante un período de 18 h (durante la noche).
-
Por la mañana, haga los siguientes preparativos.
- Placa de inductor. En una placa limpia de 96 pocillos, prepare los pozos con un medio semi-pobre (SPM) que consta de 95% BA y 5% LB26 en la incubadora a 37oC. El número de pozos corresponde al número deseado de concentraciones de inductores. Añadir C4-HSL a los pocillos de la placa inductora que contendrá la mayor concentración de inductor (218 nM).
- Programe el robot para diluir en serie el medio de cada uno de los pozos de mayor concentración en 23 concentraciones más bajas que van de 0 a 218 nM. El volumen de cada dilución del inductor debe ser suficiente para todas las cepas (incluidos los duplicados).
- Mientras se preparan las diluciones del inductor, caliente 180 s de SPM en la incubadora a 37 oC, en placas de 96 pocillos.
- Diluir las cepas durante la noche del paso 2.2 por un factor de 100 por diluciones en serie: primero diluir por un factor de 10 mezclando 100 s de bacterias con 900 sL de SPM en placas de 48 pocillos, y luego diluir de nuevo por un factor de 10 tomando 20 s de la solución diluida en 180 l de SPM precalentado, en placas de 96 pocillos adecuadas para mediciones fluorescentes.
- Agregue el inductor diluido de la placa del inductor a las placas de 96 pocillos con las cepas diluidas de acuerdo con las concentraciones finales.
- Agitar las placas de 96 pocillos a 37oC durante 6 h, mientras se toman medidas de densidad óptica a 595 nm (OD595),mCherry (560 nm/612 nm) y fluorescencia mCerulean (460 nm/510 nm) a través de un lector de placas cada 30 minutos. Para fines de normalización, mida el crecimiento de SMP sin necesidad de agregar células.
3. Análisis preliminar de resultados
- Para cada día de experimento, elija un intervalo de tiempo de crecimiento logarítmico de acuerdocon las curvas de crecimiento medidas, entre la fase de crecimiento lineal y la estacionaria (T 0, Tfinal). Tome aproximadamente 6 x 8 puntos de tiempo, mientras descarta la primera y la última mediciones para evitar errores derivados de la inexactitud de la detección exponencial del crecimiento (véase la figura 2A,panel superior).
NOTA: Deseche las cepas que muestran curvas de crecimiento anormales o cepas en las que no se pudo detectar la fase de crecimiento logarítmico y repita el experimento. -
Calcular la fluorescencia normalizada media de mCerulean y la tasa de producción de mCherry, a partir de los datos brutos de fluorescencia mcerulean y mCherry para cada concentración de inductor (Figura2A).
- Calcule mCerulean normalizado de la siguiente manera:
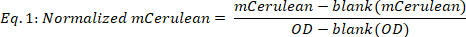
donde blank(mCerulean) es el nivel mCerulean [a.u.] para medio solamente, blank(OD) es la densidad óptica para medios solamente, y mCerulean y OD son los valores de fluorescencia mceruleana y densidad óptica, respectivamente. - MCerulean promedio sobre los diferentes puntos de tiempo (Figura2B,dos paneles superiores) de la siguiente manera:
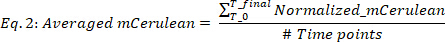
donde #Time puntos es el número de puntos de tiempo de datos que se tienen en cuenta, T0 es el momento en el que comienza la fase de crecimiento exponencial, y Tfinal es el momento en el que finaliza la fase de crecimiento exponencial. - Calcule la tasa de producción de mCherry (Figura2B,dos paneles inferiores) de la siguiente manera:
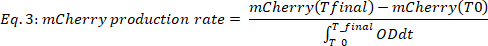
donde mCherry(t) es el nivel de mCherry [a.u.] en el momento t, OD es el valor de densidad óptica, T0 es el momento en el que comienza la fase de crecimiento exponencial, y Tfinal es el momento en el que finaliza la fase de crecimiento exponencial.
- Calcule mCerulean normalizado de la siguiente manera:
- Por último, trazar la tasa de producción de mCherry en función de mCerulean, creando curvas de respuesta a la dosis en función de la fluorescencia de fusión RBP-mCerulean (Figura2C). Tales tramas representan la producción del gen reportero en función de la presencia de la RBP en la célula.
4. Rutina de ajuste de la función de respuesta de la dosis y extracción de KRBP
- Bajo el supuesto de que la tasa de traducción ribosoma con el límite RBP es constante, modele la tasa de producción de mCherry de la siguiente manera (véase la figura 2D,línea verde):

donde [x] es la fluorescencia media normalizada de mCerulean calculada de acuerdo con Eq. 2, la tasa de producción de mCherry es el valor calculado de acuerdo con Eq. 3, KRBP es la afinidad de unión relativa [a.u.], Ksin consolidar es la tasa ribosoma de traducción con la RBP sin consolidar, n es el factor de cooperatividad, y C es la fluorescencia base [a.u.]. C, n, Ksin consolidary KRBP se encuentran ajustando los datos de la velocidad de producción de mCherry al modelo (Eq. 4). - Utilizando el software de análisis de datos, lleve a cabo un procedimiento de ajuste en parcelas que representen la tasa de producción de mCherry en función de mCerulean promedio (paso 3.3) y extraiga los parámetros de ajuste de acuerdo con la fórmula en Eq. 4.
NOTA: Solo se tienen en cuenta los resultados de ajuste con R2 > 0,6. Para esos ajustes, error KRBP está principalmente en el rango de 0.5% a 20% de los valores de KRBP, para un intervalo de confianza 0.67, mientras que aquellos con mayor error KRBP también pueden ser verificados por ojo. - Normalizar los valores deK RBP por el valor máximo respectivo de mCerulean promediado para cada función dosis-respuesta.
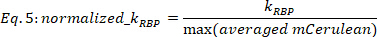
donde KRBP en [a.u.] es el valor extraído del procedimiento de ajuste en Eq. 4, y max (mCerulean promediado) es la señal mCerulean media máxima [a.u] observada para la cepa actual.
NOTA: La normalización facilita la comparación correcta del efecto regulador entre las cepas al eliminar la dependencia de los niveles máximos de expresión de RBP particulares.
Resultados
El método presentado utiliza la competencia entre una RBP y el ribosoma para la unión a la molécula de ARNm (Figura1). Esta competencia se refleja en la disminución de los niveles de mCherry en función del aumento de la producción de RBP-mCerulean, debido al aumento de las concentraciones de inductor. En el caso del aumento de la fluorescencia de la macurrulenta, sin cambios significativos en el mCherry, se deduce la falta de unión RBP. Los resultados ...
Discusión
El método descrito en este artículo facilita la medición cuantitativa in vivo de la afinidad de unión RBP-ARN en células de E. coli. El protocolo es relativamente fácil y se puede llevar a cabo sin el uso de maquinaria sofisticada, y el análisis de datos es sencillo. Además, los resultados se producen inmediatamente, sin el tiempo de espera relativamente largo asociado con los resultados de la secuenciación de próxima generación (NGS).
Una limitación a este método es que ...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este proyecto recibió financiación del Programa I-CORE del Comité de Planificación y Presupuesto y de la Fundación Científica de Israel (Grant No 152/11), Marie Curie Reintegration Grant No. PCIG11-GA- 2012-321675, y del Programa de Investigación e Innovación Horizonte 2020 de la Unión Europea en virtud del acuerdo de subvención no 664918 - MRG-Grammar.
Materiales
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
Referencias
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados