È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un saggio per quantificare il legame proteino-RNA nei batteri
In questo articolo
Riepilogo
In questo metodo, quantifichiamo l'affinità legante delle proteine leganti l'RNA (RBP) per cognare e siti di legame non-cognati utilizzando un semplice, dal vivo, saggio reporter nelle cellule batteriche. Il saggio si basa sulla repressione di un gene reporter.
Abstract
Nella fase di avvio della traduzione delle proteine, il ribosoma si lega alla regione di avvio dell'mRNA. L'avvio della traduzione può essere bloccato legando una proteina legante RNA (RBP) alla regione di avvio dell'mRNA, che interferisce con il legame ribosomico. Nel metodo presentato, utilizziamo questo fenomeno di blocco per quantificare l'affinità vincolante degli RBP ai loro siti di legame cognati e non cognati. Per fare questo, inseriamo un sito di rilegatura di test nell'area di avvio di un mRNA reporter e induciamo l'espressione del test RBP. Nel caso dell'associazione RBP-RNA, abbiamo osservato una repressione sigmoidale dell'espressione dei reporter in funzione della concentrazione di RBP. Nel caso di nessuna affinità o di affinità molto bassa tra sito vincolante e RBP, non è stata osservata alcuna repressione significativa. Il metodo viene eseguito in cellule batteriche vive e non richiede macchinari costosi o sofisticati. È utile per quantificare e confrontare tra le affinità di legame di diversi RBP che sono funzionali nei batteri con una serie di siti di legame progettati. Questo metodo può essere inappropriato per i siti di rilegatura con elevata complessità strutturale. Ciò è dovuto alla possibilità di repressione dell'avvio ribosomico da parte di una complessa struttura di mRNA in assenza di RBP, che si tradurrà in una minore espressione genica del reporter basale, e quindi meno conlacuabile repressione dei reporter sul legame RBP.
Introduzione
Negli ultimi decenni la regolazione post-trascrizione basata sulla proteina legante dell'RNA (RBP), in particolare la caratterizzazione dell'interazione tra RBP e RNA, è stata ampiamente studiata negli ultimi decenni. Ci sono diversi esempi di down-regolazione traslazionale nei batteri originari di RBP che inibiscono, o direttamente in competizione con, ribosoma legame1,2,3. Nel campo della biologia sintetica, le interazioni RBP-RNA stanno emergendo come uno strumento significativo per la progettazione di circuiti genetici basati sulla trascrizione4,5. Pertanto, c'è un aumento della domanda di caratterizzazione di tali interazioni RBP-RNA in un contesto cellulare.
I metodi più comuni per studiare le interazioni proteina-RNA sono l'analisi elettroforica del cambio di mobilità (EMSA)6, che è limitato alle impostazioni in vitro, e vari saggi pull-down7, tra cui il metodo CLIP8,9 . Mentre tali metodi consentono la scoperta di siti di legame de novo RNA, soffrono di inconvenienti come protocolli ad alta intensità di lavoro e costose reazioni di sequenziamento profondo e possono richiedere un anticorpo specifico per il pull-down RBP. A causa della natura suscettibile dell'RNA al suo ambiente, molti fattori possono influenzare le interazioni RBP-RNA, sottolineando l'importanza di interrogare il legame RBP-RNA nel contesto cellulare. Ad esempio, noi e altri abbiamo dimostrato differenze significative tra le strutture di RNA in vivo e in vitro10,11.
Sulla base dell'approccio di uno studio precedente 12, abbiamo recentemente dimostrato10 che quando si posizionano siti di legame pre-progettati per gli RBP capsidi dei batteriofages GA13, MS214, PP715e Q regione di avvio della traduzione di un reporter mRNA, espressione reporter è fortemente repressa. Vi presentiamo un metodo relativamente semplice e quantitativo, basato su questo fenomeno di repressione, per misurare l'affinità tra gli RBP e il loro corrispondente sito di legame dell'RNAin vivo.
Protocollo
1. Preparazione del sistema
- Progettazione di plasmidi di sito di rilegatura
- Progettare la cassetta del sito di rilegatura come illustrato nella Figura 1. Ogni minigene contiene le seguenti parti (da 5 a 3'): sito di restrizione Eagl, 40 basi dell'estremità 5' del gene di resistenza kanamycin (Kan), promotore di pLac-Ara, sito di rilegatura ribosomi (RBS), AUG del gene mCherry, un distanziale (che si allarga un sito di legame RBP, 80 basi dell'estremità 5'), AUG del gene mCherry, un distanziale (che si allarga un sito di legame RBP, 80 basi dell'estremità 5'), un aUG del gene mCherry, un distanziatore (che si snoda in un sito di legame RBP" del gene mCherry, e un sito di restrizione ApaLI.
NOT: Per aumentare il tasso di successo del saggio, progettare tre cassette di rilegatura per ogni sito di rilegatura, con distanziali costituiti da almeno una, due e tre basi. Vedere la sezione Risultati rappresentativi per ulteriori linee guida.
- Progettare la cassetta del sito di rilegatura come illustrato nella Figura 1. Ogni minigene contiene le seguenti parti (da 5 a 3'): sito di restrizione Eagl, 40 basi dell'estremità 5' del gene di resistenza kanamycin (Kan), promotore di pLac-Ara, sito di rilegatura ribosomi (RBS), AUG del gene mCherry, un distanziale (che si allarga un sito di legame RBP, 80 basi dell'estremità 5'), AUG del gene mCherry, un distanziale (che si allarga un sito di legame RBP, 80 basi dell'estremità 5'), un aUG del gene mCherry, un distanziatore (che si snoda in un sito di legame RBP" del gene mCherry, e un sito di restrizione ApaLI.
- Clonazione di plotoni di rilegatura del sito
- Ordinare le cassette del sito di rilegatura come minigeni di DNA a doppio filamento (dsDNA). Ogni minigene è lungo 500 bp e contiene un sito di restrizione Eagl e un sito di restrizione ApaLI alle estremità 5' e 3' rispettivamente (vedere il passaggio 1.1.1).
NOT: In questo esperimento, ai minigeni con metà del gene della kanamicicina è stato ordinato di facilitare lo screening delle colonie positive. Tuttavia, l'assemblaggio Gibson17 è adatto anche qui, nel qual caso il sito di rilegatura può essere ordinato come due oligodi DNA a filamento singolo complementari più brevi. - Raddoppiare sia i minigeni che il vettore bersaglio con Eagl-HF e ApaLI conil protocollo di restrizione18e la colonna purifica 19 .
- Ligate i minigeni digeriti alla backbone legante-sito contenente il resto del gene reporter mCherry, terminatore, e un gene di resistenza alla kanamicina20.
- Trasformare la soluzione di legatura in Escherichia coli TOP10 cellule21.
- Identificare i trasformativi positivi tramite il sequenziamento di Sanger.
- Progettare un primer 100 basi a monte della regione di interesse (vedere la tabella 1 per le sequenze di primer).
- Miniprep un paio di colonie batteriche22.
- Preparare 5 l di una soluzione di 5 mM dell'primer e 10 l del DNA a una concentrazione di 80 ng/L.
- Inviare le due soluzioni a una comoda struttura per Sanger sequenziamento23.
- Conservare plasmidi purificati a -20 gradi centigradi, e ceppi batterici come scorte glicerolo24, entrambi nel formato 96-well. Il DNA sarà quindi utilizzato per la trasformazione in cellule E. coli TOP10 contenenti uno dei quattro plasmidi fusione RBP (vedi fase 1.3.5).
- Ordinare le cassette del sito di rilegatura come minigeni di DNA a doppio filamento (dsDNA). Ogni minigene è lungo 500 bp e contiene un sito di restrizione Eagl e un sito di restrizione ApaLI alle estremità 5' e 3' rispettivamente (vedere il passaggio 1.1.1).
- Progettazione e costruzione del plasmide RBP
NOT: Le sequenze di amminoacidi e nucleotidi delle proteine del mantello utilizzate in questo studio sono elencate nella tabella 2.- Ordinare la sequenza RBP richiesta priva di un codon di arresto come minigene dsDNA personalizzato privo di un codon di arresto con siti di restrizione alle estremità (Figura 1).
- Clonare l'RBP testato privo di un codone di arresto immediatamente a valle di un promotore inducibile e a monte di una proteina fluorescente priva di un codon di inizio (Figura 1), simile ai passi 1.2.2-1.2.4. Assicurarsi che il plasmide RBP contenga un gene di resistenza agli antibiotici diverso rispetto al plasmide del sito di legame.
- Identificare i trasformazioni positivi tramite il sequenziamento di Sanger, in modo simile al passaggio 1.2.5 (vedere la tabella 1 per le sequenze di primer).
- Scegli un trasformatire positivo e rendilo chimicamente-competente25. Conservare come plasmidi purificati di glicerolo a -20 gradi centigradi e scorte di glicerolo di ceppi batterici24 a -80 gradi centigradi in piastre di 96 pozze.
- Trasformare i plasmidi del sito di rilegatura (dal punto 1.2.6) conservati in lastre di 96 pozzi in cellule batteriche chimicamente competenti che contengono già un plasmide RBP-mCerulean21. Per risparmiare tempo, invece di placcare le cellule sui piatti Petri, placcarle usando un pipettor a 8 canali su piatti a 8 corsie contenenti Luria-Bertani (LB)26 agar con antibiotici pertinenti (Kan e Amp). Le colonie dovrebbero comparire in 16 h.
- Selezionare una singola colonia per ogni doppio trasformazione e crescere durante la notte in supporto LB con gli antibiotici pertinenti (Kan e Amp) e memorizzare come scorte di glicerolo24 a -80 gradi centigradi in piatti 96-bene.
2. Configurazione dell'esperimento
NOT: Il protocollo qui presentato è stato eseguito utilizzando un sistema robotico a gestione dei liquidi in combinazione con un incubatore e un lettore di lastre. Ogni misurazione è stata effettuata per 24 concentrazioni di induttori, con due duplicati per ogni ceppo: combinazione di induttore. Utilizzando questo sistema robotico, sono stati raccolti dati per 16 ceppi al giorno con 24 concentrazioni di induttori. Tuttavia, se un tale dispositivo non è disponibile, o se sono necessari meno esperimenti, questi possono essere facilmente fatti a mano utilizzando una multi-pipetta a 8 canali e adattando il protocollo di conseguenza. Ad esempio, i risultati preliminari per quattro ceppi al giorno con 12 concentrazioni di induttori e quattro punti temporali sono stati acquisiti in questo modo.
- Preparare, in anticipo, 1 L di buffer di bioassenza (BA) mescolando 0,5 g di tryptone, 0,3 mL di glicerolo, 5,8 g di NaCl, 50 mL di 1 M MgSO4, 1 mL di 10x tampina di svafatto (PBS) tampone da tampone 7.4 e 950 mL di acqua doppia distillata (DDW). Filtro automatico o sterile del buffer BA.
- Far crescere i ceppi a doppia trasformazione a 37 e 250 giri/m l in 1,5 mL di LB con antibiotici appropriati (kanamycin ad una concentrazione finale di 25 g/mL e ampicillina ad una concentrazione finale di 100 g/mL), in piastre di 48 pozzetti, per un periodo di 18 h (pernotte).
-
Al mattino, fare i seguenti preparativi.
- Piastra di induttrice. In una piastra pulita di 96 pozzetti, preparare i pozzi con mezzi semi-poveri (SPM) composti da 95% BA e 5% LB26 nell'incubatrice a 37 gradi centigradi. Il numero di pozzi corrisponde al numero desiderato di concentrazioni di induttori. Aggiungere C4-HSL ai pozzi nella piastra dell'induttore che conterranno la più alta concentrazione di induttori (218 nM).
- Programmare il robot per diluire in serie il mezzo da ciascuno dei pozzi ad alta concentrazione in 23 concentrazioni più basse che vanno da 0 a 218 nM. Il volume di ogni diluizione dell'induttore dovrebbe essere sufficiente per tutti i ceppi (compresi i duplicati).
- Mentre le diluizioni dell'induttore sono in fase di preparazione, calde 180 gradi di SPM nell'incubatrice a 37 gradi centigradi, in piastre di 96 pozze.
- Diluire le sollecitazioni notturne da passo 2.2 con un fattore di 100 per diluizioni seriali: prima diluire con un fattore 10 mescolando 100 L di batteri con 900 SPM in lastre di 48 pozze, e poi diluire di nuovo di un fattore di 10 prendendo 20 180 l di SPM preriscaldato, in 96 pozze adatte per misurazioni fluorescenti.
- Aggiungere l'induttore diluito dalla piastra dell'induttore alle piastre del pozzo 96 con le varietà diluite in base alle concentrazioni finali.
- Agitare le lastre di 96 pozze a 37 gradi centigradi per 6 h, misurando la densità ottica a 595 nm (OD595),mCherry (560 nm/612 nm) e mCerulean (460 nm/510 nm) fluorescenza tramite un lettore di lastre ogni 30 min. Ai fini della normalizzazione, misurare la crescita di SMP senza celle aggiunte.
3. Analisi preliminare dei risultati
- Per ogni giorno dell'esperimento, scegliere un intervallo di tempo di crescita logaritmica in base alle curve di crescita misurate, tra la fase di crescita lineare e la stagente (T0, Tfinale). Prendere circa 6-8 punti di tempo, mentre scartare la prima e l'ultima misurazione per evitare errori derivati dall'imprecisione del rilevamento della crescita esponenziale (vedere Figura 2A, pannello superiore).
NOT: Eliminare i ceppi che mostrano curve o ceppi di crescita anomali in cui non è stato possibile rilevare la fase di crescita logaritmica e ripetere l'esperimento. -
Calcolare la fluorescenza media normalizzata di mCerulean e il tasso di produzione di mCherry, dai dati grezzi della fluorescenza mCerulea e mCherry per ogni concentrazione di induttore (Figura 2A).
- Calcolare mCeruleano normalizzato come segue:
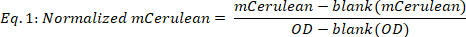
dove blank(mCerulean) è il livello mCeruleano [a.u.] solo per medie, blank(OD) è la densità ottica solo per il medio, e mCerulean e OD sono rispettivamente i valori di fluorescenza mCerulean e densità ottica. - Media mCerulean sui diversi punti temporali (Figura 2B, primi due pannelli) come segue:
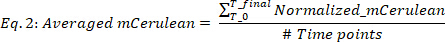
dove #Time punti è il numero di punti di tempo dei dati presi in considerazione, T0 è il momento in cui inizia la fase di crescita esponenziale e Tfinale è il momento in cui termina la fase di crescita esponenziale. - Calcolare il tasso di produzione di mCherry (Figura 2B, due pannelli inferiori) come segue:
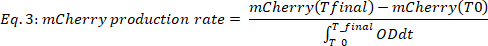
dove mCherry(t) è il livello mCherry [a.u.] al momento t, OD è il valore di densità ottica, T0 è il momento in cui inizia la fase di crescita esponenziale e Tfinale è il momento in cui termina la fase di crescita esponenziale.
- Calcolare mCeruleano normalizzato come segue:
- Infine, tracciare il tasso di produzione mCherry in funzione di mCerulean, creando curve di risposta alla dose in funzione della fluorescenza di fusione RBP-mCeruleana (Figura 2C). Tali trame rappresentano la produzione del gene reporter in funzione della presenza RBP nella cellula.
4. Dose Response Funzione di adattamento Routine e KRBP Estrazione
- Partendo dal presupposto che il tasso di traslazione ribososo con il limite RBP è costante, modellare il tasso di produzione di mCherry come segue (vedere la figura 2D,linea verde):

dove [x] è la fluorescenza mCerulea media normalizzata calcolata in base a Eq. 2, il tasso di produzione di mCherry è il valore calcolato in base a Eq. 3, KRBP è l'affinità di legame relativa [a.u.], Knon vincolata è il tasso di ribosoma di traduzione con il RBP non associato, n è il fattore coty, e C è la fluorescenza di base [a.u.]. C, n,K , e KRBP si trovano adattando i dati del tasso di produzione di mCherry al modello (Eq. 4). - Utilizzando il software di analisi dei dati, condurre una procedura di adattamento su grafici che raffigurano il tasso di produzione di mCherry in funzione della media mCerulean (passaggio 3.3), ed estrarre i parametri di adattamento in base alla formula in Eq. 4.
NOT: Vengono presi in considerazione solo i risultati di montaggio con R2 > 0.6. Per quelle si adatta, KRBP errore è per lo più nell'intervallo di 0.5% A 20% dei valoriRBP K, per un intervallo di confidenza 0.67, mentre quelli con più alto errore KRBP possono anche essere verificati ad occhio. - Normalizzare i valori KRBP in base al rispettivo valore massimo di mCerulean mediato per ogni funzione dose-risposta.
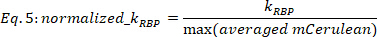
dove KRBP in [a.u.] è il valore estratto dalla procedura di adattamento in Eq. 4 e max (media mCerulean) è il segnale mCerulean medio massimo [a.u] osservato per la deformazione corrente.
NOT: La normalizzazione facilita il corretto confronto dell'effetto normativo tra i ceppi eliminando la dipendenza dai livelli massimi di espressione RBP.
Risultati
Il metodo presentato utilizza la competizione tra un RBP e il ribosoma per il legame con la molecola mRNA (Figura 1). Questa concorrenza si riflette nella diminuzione dei livelli di mCherry in funzione dell'aumento della produzione di RBP-mCerulean, a causa delle crescenti concentrazioni di induttore. Nel caso di aumento della fluorescenza mCerulea, senza cambiamenti significativi in mCherry, viene dedotta una mancanza di legame RBP. I risultati rappresentati...
Discussione
Il metodo descritto in questo articolo facilita la misurazione quantitativa in vivo dell'affinità di legame RBP-RNA nelle cellule di E. coli. Il protocollo è relativamente semplice e può essere condotto senza l'uso di macchinari sofisticati e l'analisi dei dati è semplice. Inoltre, i risultati vengono prodotti immediatamente, senza il tempo di attesa relativamente lungo associato ai risultati di sequenziamento (NGS) di prossima generazione.
Una limitazione a questo metodo è che f...
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo progetto ha ricevuto finanziamenti dal programma I-CORE del Comitato di pianificazione e budgeting e dalla Israel Science Foundation (Grant no. 152/11), Marie Curie Reintegration Grant No. PCIG11-GA- 2012-321675 e dal programma di ricerca e innovazione Orizzonte 2020 dell'Unione europea nell'ambito dell'accordo di sovvenzione n. 664918 - MRG-Grammar.
Materiali
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
Riferimenti
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon