Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un jeu d'enfant pour quantifier la liaison protéine-ARN chez les bactéries
Dans cet article
Résumé
Dans cette méthode, nous quantifions l'affinité de liaison des protéines de liaison d'ARN (RBPs) aux emplacements de liaison de cognate et non-cognate utilisant un simple, vivant, test de journaliste dans les cellules bactériennes. L'exemple est basé sur la répression d'un gène journaliste.
Résumé
Dans l'étape d'initiation de la traduction de protéine, le ribosome se lie à la région d'initiation de l'ARNm. L'initiation à la traduction peut être bloquée par la liaison d'une protéine de liaison de l'ARN (RBP) à la région d'initiation de l'ARNm, qui interfère avec la liaison ribosome. Dans la méthode présentée, nous utilisons ce phénomène de blocage pour quantifier l'affinité contraignante des RRP à leurs sites de liaison cognate et non cognate. Pour ce faire, nous insérons un site de liaison de test dans la région d'initiation d'un ARNm journaliste et induisons l'expression du test RBP. Dans le cas de la liaison RBP-ARN, nous avons observé une répression sigmoïdale de l'expression du journaliste en fonction de la concentration du RBP. Dans le cas de l'absence d'affinité ou d'une très faible affinité entre le site de liaison et le RBP, aucune répression significative n'a été observée. La méthode est réalisée dans des cellules bactériennes vivantes, et ne nécessite pas de machinerie coûteuse ou sophistiquée. Il est utile pour quantifier et comparer entre les affinités de liaison de différents RBP qui sont fonctionnels dans les bactéries à un ensemble de sites de liaison conçus. Cette méthode peut être inappropriée pour lier les sites à forte complexité structurelle. Cela est dû à la possibilité de répression de l'initiation ribosomale par la structure complexe de l'ARNm en l'absence de RBP, ce qui se traduirait par une expression plus faible du gène journaliste basique, et donc moins observable journaliste répression sur RBP liaison.
Introduction
La régulation post-transcriptionnelle basée sur la protéine de liaison d'ARN (RBP), spécifiquement la caractérisation de l'interaction entre RBPs et ARN, a été étudiée intensivement au cours des dernières décennies. Il existe de multiples exemples de régulation translationnelle des bactéries provenant de RRP inhibant, ou en concurrence directe avec, la liaison ribosome1,2,3. Dans le domaine de la biologie synthétique, les interactions RBP-ARN apparaissent comme un outil important pour la conception de circuits génétiques basés sur la transcription4,5. Par conséquent, il y a une augmentation de la demande pour la caractérisation de telles interactions RBP-ARN dans un contexte cellulaire.
Les méthodes les plus courantes pour étudier les interactions protéine-ARN sont l'analyse de changement de mobilité électrophorétique (EMSA)6, qui est limitée aux paramètres in vitro, et divers essais de tractionvers lebas 7 , y compris la méthode CLIP8,9 . Bien que ces méthodes permettent la découverte de sites de liaison de l'ARN de novo, elles souffrent d'inconvénients tels que des protocoles à forte intensité de main-d'œuvre et des réactions coûteuses de séquençage profond et peuvent nécessiter un anticorps spécifique pour le retrait du RBP. En raison de la nature sensible de l'ARN à son environnement, de nombreux facteurs peuvent affecter les interactions RBP-ARN, soulignant l'importance d'interroger la liaison RBP-ARN dans le contexte cellulaire. Par exemple, nous et d'autres avons démontré des différences significatives entre les structures de l'ARN in vivo et in vitro10,11.
Basé sur l'approche d'une étude précédente12, nous avons récemment démontré10 que lors du placement de sites de liaison pré-conçus pour les RRP capsides des bactériophages GA13, MS214, PP715, et Q16 dans le région d'initiation de traduction d'un ARNm de journaliste, l'expression de journaliste est fortement réprimée. Nous présentons une méthode relativement simple et quantitative, basée sur ce phénomène de répression, pour mesurer l'affinité entre les RBP et leur site de liaison d'ARN correspondantin vivo.
Protocole
1. Préparation du système
- Conception de plasmides de site de liaison
- Concevoir la cassette du site de liaison telle qu'elle est représentée dans la figure 1. Chaque minigène contient les parties suivantes (5' à 3') : site de restriction d'Eagl, 40 bases de l'extrémité 5' du gène de résistance de kanamycine (Kan), promoteur de pLac-Ara, site de liaison de ribosome (RBS), AUG du gène mCherry, un espaceur () un site de liaison DeBP, 80 bases de la fin de 5' du gène mCherry, et un site de restriction ApaLI.
REMARQUE: Pour augmenter le taux de réussite de l'assiduité, concevez trois cassettes de site de liaison pour chaque site de liaison, avec des espaceurs composés d'au moins une, deux et trois bases. Voir la section Résultats représentatifs pour d'autres lignes directrices.
- Concevoir la cassette du site de liaison telle qu'elle est représentée dans la figure 1. Chaque minigène contient les parties suivantes (5' à 3') : site de restriction d'Eagl, 40 bases de l'extrémité 5' du gène de résistance de kanamycine (Kan), promoteur de pLac-Ara, site de liaison de ribosome (RBS), AUG du gène mCherry, un espaceur () un site de liaison DeBP, 80 bases de la fin de 5' du gène mCherry, et un site de restriction ApaLI.
- Clonage des plasmides de site de liaison
- Commandez les cassettes de site de liaison en minigènes à double brin d'ADN (ADN d'ADN). Chaque minigène mesure 500 pb de long et contient un site de restriction Eagl et un site de restriction ApaLI aux extrémités de 5' et 3' respectivement (voir l'étape 1.1.1).
REMARQUE: Dans cette expérience, des mini-gènes avec la moitié du gène de la kanamycine ont été ordonnés pour faciliter le dépistage des colonies positives. Cependant, l'assemblage Gibson17 est également approprié ici, auquel cas le site de liaison peut être commandé comme deux oligos complémentaires plus courts d'ADN simple brin. - Double-digérer à la fois les mini-gènes et le vecteur cible avec Eagl-HF et ApaLI par le protocole de restriction18, et la colonne purifier19.
- Ligate les minigènes digérés à l'épine dorsale du site de liaison contenant le reste du gène journaliste mCherry, terminator, et un gène de résistance à la kanamycine20.
- Transformez la solution de ligation en cellules EScherichia coli TOP1021.
- Identifier les transformateurs positifs par séquençage Sanger.
- Concevoir une amorce 100 bases en amont de la région d'intérêt (voir tableau 1 pour les séquences d'amorce).
- Miniprep quelques colonies bactériennes22.
- Préparer 5 ll d'une solution de 5 mM de l'amorce et 10 l de l'ADN à une concentration de 80 ng/L.
- Envoyer les deux solutions à une installation pratique pour le séquençage Sanger23.
- Entreposez les plasmides purifiés à -20 oC, et les souches bactériennes sous forme de stocks de glycérol24, tous deux dans le format 96 puits. L'ADN sera ensuite utilisé pour la transformation en cellules E. coli TOP10 contenant l'un des quatre plasmides fusion-RBP (voir l'étape 1.3.5).
- Commandez les cassettes de site de liaison en minigènes à double brin d'ADN (ADN d'ADN). Chaque minigène mesure 500 pb de long et contient un site de restriction Eagl et un site de restriction ApaLI aux extrémités de 5' et 3' respectivement (voir l'étape 1.1.1).
- Conception et construction du plasmide RBP
REMARQUE: Les séquences d'acide aminé et de nucléotide des protéines de couche utilisées dans cette étude sont énumérées dans le tableau 2.- Commandez la séquence RBP requise sans codon d'arrêt en tant que minigène d'ADN personnalisé dépourvu d'un codon d'arrêt avec des sites de restriction aux extrémités (Figure 1).
- Cloner le RBP testé sans codon d'arrêt immédiatement en aval d'un promoteur inductible et en amont d'une protéine fluorescente dépourvue d'un codon de départ (figure 1), semblable aux étapes 1.2.2-1.2.4. Assurez-vous que le plasmide RBP contient un gène de résistance aux antibiotiques différent du plasmide du site de liaison.
- Identifier les transformants positifs par séquençage Sanger, semblable à l'étape 1.2.5 (voir le tableau 1 pour les séquences d'apprêt).
- Choisissez un transformateur positif et le rendre chimiquement compétent25. Conservez sous forme de plasmides purifiés au glycérol à -20 oC et en stocks de glycérol de souches bactériennes24 à -80 oC dans des assiettes de 96 puits.
- Transformez les plasmides du site de liaison (à partir de l'étape 1.2.6) stockés dans des plaques de 96 puits en cellules bactériennes chimiquement compétentes contenant déjà un plasmide RBP-mCerulean21. Pour gagner du temps, au lieu de placage les cellules sur les plats Petri, les plaquer à l'aide d'un pipettor à 8 canaux sur des plaques à 8 voies contenant Luria-Bertani (LB)26 agar avec des antibiotiques pertinents (Kan et Amp). Les colonies devraient apparaître en 16 h.
- Sélectionnez une seule colonie pour chaque double transformateur et poussez du jour au lendemain dans le milieu LB avec les antibiotiques pertinents (Kan et Amp) et entreposez les stocks de glycérol24 à -80 oC dans des assiettes de 96 puits.
2. Configuration d'expérience
REMARQUE: Le protocole présenté ici a été exécuté à l'aide d'un système robotique de manipulation liquide en combinaison avec un incubateur et un lecteur de plaque. Chaque mesure a été effectuée pour 24 concentrations d'inducteurs, avec deux doublons pour chaque combinaison souche et inducteur. À l'aide de ce système robotique, des données sur 16 souches par jour avec 24 concentrations d'inducteurs ont été recueillies. Toutefois, si un tel dispositif n'est pas disponible, ou si moins d'expériences sont nécessaires, ceux-ci peuvent facilement être faites à la main en utilisant un multi-pipette à 8 canaux et en adaptant le protocole en conséquence. Par exemple, les résultats préliminaires de quatre souches par jour avec 12 concentrations d'inducteurs et quatre points de temps ont été acquis de cette façon.
- Préparer, à l'avance, 1 L de tampon de bio-analyse (BA) en mélangeant 0,5 g de tryptone, 0,3 ml de glycérol, 5,8 g de NaCl, 50 ml de 1 M MgSO4, 1 mL de 10x tampon salin tamponné par phosphate (PBS) pH 7,4, et 950 mL d'eau distillée double (DDW). Filtre automatique ou stérile de la mémoire tampon BA.
- Cultivez les souches à double régime à 37 oC et 250 tr/min en secouant dans 1,5 mL lb avec des antibiotiques appropriés (kanamycine à une concentration finale de 25 g/mL et ampicilline à une concentration finale de 100 g/mL), dans des plaques de 48 puits, sur une période de 18 h (nuit).
-
Le matin, faites les préparatifs suivants.
- Plaque inducteur. Dans une assiette propre de 96 puits, préparer les puits avec un milieu semi-pauvre (SPM) composé de 95 % de BA et deLB 26 à 5 % dans l'incubateur à 37 oC. Le nombre de puits correspond au nombre souhaité de concentrations inducteurs. Ajouter c4-HSL aux puits de la plaque inducteur qui contiendra la plus forte concentration d'inducteurs (218 nM).
- Programmez le robot pour diluer en série le milieu de chacun des puits les plus à concentration élevée en 23 concentrations inférieures allant de 0 à 218 nM. Le volume de chaque dilution inducteur doit être suffisant pour toutes les souches (y compris les doublons).
- Pendant que les dilutions inducteurs sont en cours de préparation, réchauffez 180 L de SPM dans l'incubateur à 37 oC, dans des assiettes de 96 puits.
- Diluer les souches de nuit de l'étape 2.2 par un facteur de 100 par dilutions en série: diluer d'abord par un facteur de 10 en mélangeant 100 L de bactéries avec 900 L de SPM dans 48-puits plaques, puis diluer à nouveau par un facteur de 10 en prenant 20 L de la solution diluée dans 180 l de SPM pré-chauffé, dans des plaques de 96 puits adaptées aux mesures fluorescentes.
- Ajouter l'inducteur dilué de la plaque inducteur aux plaques de 96 puits avec les souches diluées selon les concentrations finales.
- Secouez les plaques de 96 puits à 37 oC pendant 6 h, tout en prenant des mesures de densité optique à 595 nm (OD595), mCherry (560 nm/612 nm) et mCerulean (460 nm/510 nm) fluorescence via un lecteur de plaque toutes les 30 min. Aux fins de normalisation, mesurez la croissance de SMP sans ajout de cellules.
3. Analyse préliminaire des résultats
- Pour chaque jour d'expérience, choisissez un intervalle de temps de croissance logarithmique en fonction des courbes de croissance mesurées, entre la phase de croissance linéaire et l'stationnaire (T0,T final). Prenez environ 6 à 8 points de temps, tout en rejetant les premières et dernières mesures pour éviter les erreurs découlant de l'inexactitude de la détection exponentielle de la croissance (voir la figure 2A, panneau supérieur).
REMARQUE: Jetez les souches qui présentent des courbes de croissance anormales ou des souches où la phase de croissance logarithmique n'a pas pu être détectée et répéter l'expérience. -
Calculer la fluorescence normalisée moyenne du mCerulean et le taux de production de mCherry, à partir des données brutes de fluorescence mCerulean et mCherry pour chaque concentration inducteure (Figure 2A).
- Calculer mCerulean normalisé comme suit:
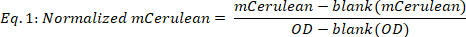
où le blanc (mCerulean) est le niveau mCerulean [a.u.] pour le milieu seulement, blanc (OD) est la densité optique pour le milieu seulement, et le mCerulean et oD sont la fluorescence mCerulean et les valeurs de densité optique, respectivement. - Moyenne mCerulean sur les différents points de temps (Figure 2B, les deux premiers panneaux) comme suit:
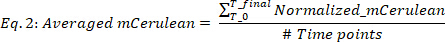
lorsque #Time points est le nombre de points de temps de données pris en compte, T0 est le moment où la phase de croissance exponentielle commence, et Tfinal est le moment où la phase de croissance exponentielle se termine. - Calculer le taux de production de mCherry (Figure 2B, les deux panneaux inférieurs) comme suit :
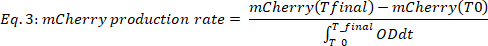
où mCherry(t) est le niveau mCherry [a.u.] au moment t, OD est la valeur de densité optique, T0 est le moment où la phase de croissance exponentielle commence, et Tfinale est le moment où la phase de croissance exponentielle se termine.
- Calculer mCerulean normalisé comme suit:
- Enfin, tracez le taux de production de mCherry en fonction du mCerulean, en créant des courbes de réponse de dose en fonction de la fluorescence de fusion RBP-mCerulean (figure 2C). Ces parcelles représentent la production du gène reporter en fonction de la présence de RBP dans la cellule.
4. Dose Response Function Fitting Routine et KRBP Extraction
- En supposant que le taux de traduction de ribosome avec la limite RBP est constant, modélisez le taux de production de mCherry comme suit (voir figure 2D, ligne verte) :

où [x] est la fluorescence moyenne normalisée de mCerulean calculée selon Eq. 2, taux de production de mCherry est la valeur calculée selon Eq. 3, KRBP est l'affinité relative de liaison [a.u.], Kunbound est le taux de ribosome de traduction avec le RBP non lié, n est le facteur de coopérativité, et C est la fluorescence de base [a.u.]. C, n, Kunbound, et KRBP sont trouvés en adaptant les données de taux de production mCherry au modèle (Eq. 4). - À l'aide d'un logiciel d'analyse de données, effectuez une procédure d'ajustement sur les parcelles représentant le taux de production de mCherry en fonction de la moyenne mCerulean (étape 3.3), et extrayez les paramètres d'ajustement selon la formule de l'Eq. 4.
REMARQUE: Seuls les résultats d'ajustement avec R2 'gt; 0.6 sont pris en compte. Pour ces ajustements, l'erreur KRBP est principalement dans la gamme de 0,5% à 20% des valeurs KRBP, pour un intervalle de confiance de 0,67, tandis que ceux avec une erreur Plus élevée KRBP peut également être vérifié par l'œil. - Normalisez les valeurs KRBP par la valeur maximale respective du mCerulean moyen pour chaque fonction dose-réponse.
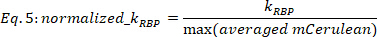
où KRBP dans [a.u.] est la valeur extraite de la procédure d'ajustement dans Eq. 4, et max (mCerulean moyen) est le signal mCerulean moyen maximal [a.u] observé pour la souche actuelle.
REMARQUE: La normalisation facilite la comparaison correcte de l'effet réglementaire entre les souches en éliminant la dépendance à l'égard des niveaux d'expression maximaux particuliers du RBP.
Résultats
La méthode présentée utilise la concurrence entre un RBP et le ribosome pour se lier à la molécule d'ARNm (Figure 1). Cette concurrence se reflète par la diminution des niveaux de mCherry en fonction de l'augmentation de la production de RBP-mCerulean, en raison de l'augmentation des concentrations d'inducteur. Dans le cas de l'augmentation de la fluorescence mCérulean, sans changements significatifs dans mCherry, un manque de liaison DeRBP est déduit...
Discussion
La méthode décrite dans cet article facilite la mesure in vivo quantitative de l'affinité de liaison RBP-ARN dans les cellules de E. coli. Le protocole est relativement facile et peut être effectué sans l'utilisation de machines sophistiquées, et l'analyse des données est simple. En outre, les résultats sont produits immédiatement, sans le temps d'attente relativement long associé aux résultats du séquençage de la prochaine génération (NGS).
Une limitation à cette mét...
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce projet a reçu un financement du programme I-CORE du Comité de planification et de budget et de la Fondation des sciences israéliennes (Grant no 152/11), Marie Curie Reintegration Grant No. PCIG11-GA- 2012-321675, et du programme de recherche et d'innovation Horizon 2020 de l'Union européenne dans le cadre de l'accord de subvention no 664918 - MRG-Grammar.
matériels
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
Références
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon