A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
שיטת בדיקת מעטפת חלבון-RNA כריכת חיידקים
In This Article
Summary
בשיטה זו, אנו מכמת את הזיקה המחייב של מאגד כריכת RNA (rbps) כדי קנצוני ואתרי כריכה שאינם קנצוני באמצעות שיטה פשוטה, לחיות, העיתונאי בתאי חיידקי. השיטת היסוד מבוססת על דיכוי של גן עיתונאי.
Abstract
בשלב החניכה של תרגום חלבונים, הריבוחלק נקשר לאזור החניכה של mRNA. אתחול תרגום ניתן לחסום על ידי איגוד של חלבון כריכת RNA (RBP) לאזור החניכה של mRNA, אשר מפריעה עם ריבוכמה הכריכה. בשיטה המוצגת, אנו מנצלים את תופעת החסימה הזאת לכמת את הזיקה המחייב של rbps לאתרים שאינם מחייבים קנצוני שלהם. כדי לעשות זאת, אנו מכניסים אתר מחייב בדיקה באזור החניכה של העיתונאי mRNA ולגרום לביטוי RBP הבדיקה. במקרה של RBP-RNA כריכה, הבחנו דיכוי החתימה של ביטוי הכתב כפונקציה של ריכוז RBP. במקרה של חוסר אהדה או זיקה נמוכה מאוד בין אתר האיגוד ל-RBP, לא נצפתה דיכוי משמעותי. השיטה מבוצעת בתאי חיידקי חיים, ואינה דורשת מכונות יקרות או מתוחכמות. היא שימושית לכימות והשוואה בין הקשרים המחייבים של האיגוד של RBPs שונים הפונקציונליים בחיידקים לקבוצה של אתרי איגוד מעוצבים. שיטה זו עשויה להיות לא מתאימה לאתרי איגוד בעלי מורכבות מבנית גבוהה. זאת בשל האפשרות של דיכוי של חניכה הריבוזומלית על ידי מבנה mRNA מורכב בהעדר RBP, אשר יגרום הביטוי הנמוך ביותר גנים כתבת בסיס, ולכן הדיכוי כתב פחות הנצפה על הכריכה RBP.
Introduction
ה-RNA כריכת חלבון (RBP) מבוסס לאחר ההמרה רגולציה, אפיון במיוחד של האינטראקציה בין Rbp ו-RNA, נחקרו בהרחבה בעשורים האחרונים. ישנן מספר דוגמאות של התקנה למטה הטרנסלtional בחיידקים שמקורם rbps עיכוב, או מתחרה ישירות עם, ריבוכמה קשירה1,2,3. בתחום הביולוגיה הסינתטית, האינטראקציות של rbp-RNA מתפתחות ככלי משמעותי לעיצוב המעגלים הגנטיים המבוססים על תמלול4,5. לכן, קיימת עלייה בביקוש לאפיון של אינטראקציות RBP-RNA כגון בהקשר הסלולר.
השיטות הנפוצות ביותר ללימוד אינטראקציות של חלבון-RNA הן שיטת השינוי בשיטת הניידות האלקטרופיניטית (emsa)6, המוגבלת להגדרות חוץ-גופית, ושונות משיכה מספר7, כולל שיטת הקליפ8,9 . בעוד שיטות כאלה לאפשר גילוי של אתרי האיגוד של דה נובו RNA, הם סובלים מחסרונות כגון פרוטוקולים עתירי עבודה ותגובות ברצף יקר והוא עשוי לדרוש נוגדן ספציפי עבור RBP למשוך למטה. בשל האופי הפגיע של RNA לסביבתו, גורמים רבים יכולים להשפיע על אינטראקציות RBP-RNA, תוך שימת דגש על החשיבות של חקירת מחייב RBP-RNA בהקשר הסלולר. לדוגמא, אנחנו ואחרים הדגמנו הבדלים משמעותיים בין מבני RNA בvivo ובתוך מבחנה10,11.
בהתבסס על הגישה של המחקר הקודם12, לאחרונה הפגינו10 כי כאשר ממקמים מעוצב מראש אתרי קשירה עבור capsid rbps מתוך בקטביאז GA13, MS214, PP715, ו Qβ16 ב התרגום האזור של כתבת mRNA, ביטוי העיתונאי הוא מודחק מאוד. אנו מציגים שיטה פשוטה יחסית וכמותית, המבוססת על תופעה זו של דיכוי, כדי למדוד את הזיקה בין RBPs לאתר המקבילשל ה -RNA שלהם ב-vivo.
Protocol
1. הכנת המערכת
- תכנון הפלמידים באתר
- עצב את קלטת אתר האיגוד כמתואר באיור 1. כל minigene מכיל את החלקים הבאים (5 ' עד 3 '): הגבלת האתר, ∼ 40 בסיסים של 5 ' סוף של kanamycin ' גן ההתנגדות, פנקייק-Ara מקדם, ריבוחלק באתר מחייב (RBS), אוג של הגן mCherry, מרווח (δ), אתר כריכת RBP, 80 בסיסים של 5 ' סוף של הגן של מג. ואתר הגבלת אפאלי
הערה: כדי להגדיל את שיעור ההצלחה של המנה, עצב שלוש קלטות של האתר המחייב עבור כל אתר מחייב, עם מרווחים הכוללים לפחות אחד, שניים ושלושה בסיסים. לקבלת הנחיות נוספות, ראה סעיף תוצאות מייצגות.
- עצב את קלטת אתר האיגוד כמתואר באיור 1. כל minigene מכיל את החלקים הבאים (5 ' עד 3 '): הגבלת האתר, ∼ 40 בסיסים של 5 ' סוף של kanamycin ' גן ההתנגדות, פנקייק-Ara מקדם, ריבוחלק באתר מחייב (RBS), אוג של הגן mCherry, מרווח (δ), אתר כריכת RBP, 80 בסיסים של 5 ' סוף של הגן של מג. ואתר הגבלת אפאלי
- שיבוט של משטח כריכה
- הזמן את הקלטות של אתר האיגוד כמו DNA כפול תקוע (dsDNA) minigenes. כל minigene הוא ∼ 500 bp ארוך ומכיל אתר הגבלה של איקל ואתר הגבלה ApaLI ב 5 ' ו 3 ' מסתיים, בהתאמה (ראה שלב 1.1.1).
הערה: בניסוי זה, מיני גנים עם חצי של הגן kanamycin הורו להקל על ההקרנה עבור מושבות חיוביות. עם זאת, מכלול גיבסון17 מתאים גם כאן, ובמקרה זה את האתר הכריכה ניתן להזמין כמו שני קצר יותר משלימים בודדים-DNA oligos. - יש לעכל את שני הגנים הקטנים ואת וקטור היעד באמצעות הפרוטוקול, והעמודה מטהראת התדר19.
- ליגייט המיניגנים מתעכל לעמוד השדרה של האיגוד המכיל את שאר הגן של העיתונאי mCherry, שליחות קטלנית, ואת התנגדות kanamycin '20.
- הפוך את הפתרון הTOP10 לתוך es, coli קולי תאים21.
- זהה transformants חיוביות באמצעות רצף שםשל Sanger.
- עיצוב פריימר 100 בסיסים במעלה הזרם לאזור העניין (ראה לוח 1 עבור רצפים פריימר).
- . מיניכין כמה מושבות בקטריאלי22
- להכין 5 μL של פתרון 5 מ"מ של פריימר ו 10 μL של ה-DNA ב 80 ng/μL ריכוז.
- שילחו את שני הפתרונות למתקן נוח.
- חנות הפלסטיק מטוהרים ב-20 ° c, וזנים חיידקיים כמו גליצרול מניות24, הן בפורמט 96-גם. ה-DNA לאחר מכן ישמש לטרנספורמציה לתוך E. coli TOP10 תאים המכילים אחד ארבעה היתוך-rbp פלמידים (ראה שלב 1.3.5).
- הזמן את הקלטות של אתר האיגוד כמו DNA כפול תקוע (dsDNA) minigenes. כל minigene הוא ∼ 500 bp ארוך ומכיל אתר הגבלה של איקל ואתר הגבלה ApaLI ב 5 ' ו 3 ' מסתיים, בהתאמה (ראה שלב 1.1.1).
- תכנון ובניית פלסמיד RBP
הערה: חומצת אמינו רצפי נוקלאוטיד של חלבונים מעיל המשמשים במחקר זה מפורטים בטבלה 2.- הזמן את רצף rbp הנדרש חסר קודון לעצור כמו מותאם אישית dsdna minigene חסר הפסקת קודון עם אתרי הגבלה בקצוות (איור 1).
- שיבוט rbp נבדק חסר לעצור קודון מיד במורד של יזם inducible ובמעלה של חלבון פלורסנט חסר קודון להתחיל (איור 1), בדומה לשלבים 1.2.2-1.2.4. ודא כי במרכז RBP מכיל גנים שונים עמידות לאנטיביוטיקה מאשר האיגוד באתר הקשירה.
- זהה transformants חיובי באמצעות רצף שלבים, בדומה לשלב 1.2.5 (ראה לוח 1 עבור רצפים פריימר).
- לבחור טרנספורנט חיובי אחד ולהפוך אותו כימית מוסמך25. חנות כמו גליצנול מטוהרים פלמידים ב-20 ° צ' וגליצרול מלאי של זנים חיידקיים24 ב-80 ° c ב 96-טוב צלחות.
- הפוך את הפלמידים באתר האיגוד (משלב 1.2.6) המאוחסנים 96-היטב צלחות לתוך תאים חיידקיים כימית מוסמך כבר מכיל RBP-mCerulean פלמיד21. כדי לחסוך זמן, במקום להתאים את התאים על מנות פטרי, לוחית אותם באמצעות שמונה ערוצים של מצנאו על לוחות 8-ליין המכילים לוריא-ברטני (LB)26 אגר עם אנטיביוטיקה רלוונטיים (Kan ו Amp). . המושבות אמורות להופיע ב -16 שעות
- בחר מושבה אחת עבור כל טרנספורנט כפול ולצמוח לילה במדיום LB עם אנטיביוטיקה הרלוונטיים (קאן ו Amp) ולאחסן כמו גליצרול מניות24 ב-80 ° c ב 96-טוב צלחות.
2. התקנת ניסוי
הערה: הפרוטוקול המוצג כאן בוצע באמצעות מערכת רובוטית עם טיפול נוזלי בשילוב עם אינקובטור וקורא צלחת. כל מדידה בוצעה עבור 24 ריכוזי סליל, עם שני כפילויות עבור כל מאמץ + הסליל שילוב. באמצעות מערכת רובוטית זו, נתונים עבור 16 זנים ליום עם 24 ריכוזי סליל נאסף. עם זאת, אם התקן כזה אינו זמין, או אם יש פחות ניסויים נחוצים, ניתן לעשות זאת בקלות באמצעות מולטי-פיפטה של 8 ערוצים והתאמת הפרוטוקול בהתאם. לדוגמה, תוצאות ראשוניות של ארבעה זנים ליום עם 12 ריכוזי סליל וארבעה נקודות זמן נרכשו באופן זה.
- היכונו, מראש, 1 L של מאגר ביולוגי (BA) על ידי ערבוב 0.5 g של טריטונים, 0.3 מ ל של גליצרול, 5.8 g של נרוג, 50 mL של 1 M MgSO4, 1 מ ל של מלוחים 10x פוספט באגירה (PBS) מאגר pH 7.4, ו-950 mL של מים כפולים מזוקקים סינון באמצעות אוטוקלב או סטרילי מאגר ה-BA.
- לגדל את זנים הטרנספורקנטים כפולה ב 37 ° צ' ו 250 rpm לרעוד 1.5 mL ליברות עם אנטיביוטיקה המתאימה (kanamycin בריכוז הסופי של 25 μg/mL ו אמפיצילין בריכוז הסופי של ה100 μg/mL), בשנת 48 לוחות, במשך תקופה של 18 h (לילה).
-
בבוקר, בצע את ההכנות הבאות.
- . לוחית השראה בצלחת נקייה 96-באר, להכין בארות עם מדיום חצי עני (SPM) המורכב של 95% BA ו 5% LB26 בחממה ב 37 ° c. מספר הבארות מקביל למספר הרצוי של ריכוזי הסליל. הוסף C4-HSL לבארות בצלחת הסליל שיכיל את הריכוז הגבוה ביותר הסליל (218 nM).
- תכנת את הרובוט כדי לדלל בינונית באופן סדרתי מכל אחד הבארות ריכוז הגבוהה ביותר 23 ריכוזים נמוכים החל מ 0 כדי 218 ננומטר. נפח של דילול כל הסליל צריך להספיק עבור כל הזנים (כולל כפילויות).
- בעוד הדילול הסליל הם להיות מוכנים, חם 180 μL של SPM בחממה ב 37 ° c, ב 96-טוב צלחות.
- לדלל את הזנים לילה משלב 2.2 על ידי פקטור של 100 על ידי דילול סדרתי: הראשון לדלל על ידי גורם של 10 על ידי ערבוב 100 μL של חיידקים עם 900 μL של SPM ב 48-טוב צלחות, ולאחר מכן לדלל שוב על ידי גורם של 10 על ידי נקיטת 20 μL מהפתרון מדולל לתוך 180 μL של מקדם-התחממות-מחמם, ב 96-טוב צלחות מתאים למדידות פלורסנט.
- הוסף את הסליל מדולל מן הצלחת הסליל אל 96-היטב צלחות עם זנים מדולל בהתאם לריכוזים הסופי.
- לנער את 96-היטב צלחות ב 37 ° צ' עבור 6 h, תוך נטילת מדידות של צפיפות אופטית ב 595 nm (OD595), mcherry (560 nm/612 nm) ו mCerulean (460 nm/510 nm) הזריחה דרך קורא צלחת כל 30 דקות. למטרות נורמליזציה, מדידת הצמיחה של SMP ללא תאים שנוספו.
3. ניתוח תוצאות ראשוניות
- לכל יום של ניסוי, בחר מרווח זמן של גידול לוגריתמי לפי עקומות הגדילה הנמדדות, בין שלב הצמיחה הליניארי לבין הנייח (T0, tfinal). קח כ 6-8 נקודות זמן, תוך השמטת המידות הראשונות והאחרונות כדי למנוע שגיאה הנגזרת מחוסר דיוק בזיהוי הגדילה האקספוננציאלי (ראה איור 2A, הלוח העליון).
הערה: להשליך זנים הצגת עקומות גדילה נורמלי או זנים שבו לא ניתן לזהות את שלב הגדילה לוגריתמי ולחזור על הניסוי. -
לחשב את הזריחה המנורמלת הממוצעת של mCerulean ושיעור הייצור של mCherry, מהנתונים הגולמיים של mCerulean ו-הקרינה הפלואורסצנטית עבור כל ריכוז הסליל (איור 2A).
- חשב mCerulean מנורמל כדלקמן:
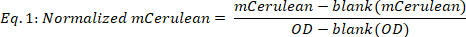
כאשר blank (mCerulean) הוא הרמה הmCerulean [a.u.] עבור בינונית בלבד, blank (OD) היא הדחיסות האופטית עבור בינונית בלבד, ו-mCerulean ו-OD הם ערכי הדחיסות האופטית והאופטים של mCerulean, בהתאמה. - ממוצע mCerulean על נקודות זמן שונות (איור 2B, שני לוחות העליון) כדלקמן:
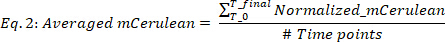
כאשרTime נקודות הוא מספר נקודות הזמן של הנתונים שנלקחו בחשבון, T0 הוא הזמן שבו מתחיל שלב הגידול האקספוננציאלי, ו-tfinal הוא הזמן בו מסתיים שלב הגידול האקספוננציאלי. - חשב את שיעור הייצור של mCherry (איור 2B, שני הפאנלים התחתונים) כדלקמן:
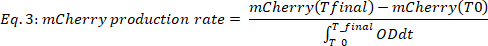
כאשר mCherry (t) היא רמת mCherry [a.u.] בזמן t, OD הוא ערך הצפיפות האופטית, T0 הוא הזמן שבו שלב הגידול האקספוננציאלי מתחיל, ו-t הסופי הוא הזמן שבו שלב הגידול האקספוננציאלי מסתיים.
- חשב mCerulean מנורמל כדלקמן:
- לבסוף, התווה את שיעור הייצור של mCherry כפונקציה של mCerulean, יצירת עקומות של תגובת מינון כפונקציה של היתוך RBP-mCerulean פיוז'ן (איור 2C). חלקות כאלה מייצגות ייצור של גן העיתונאי כפונקציה של נוכחות RBP בתא.
4. מינון פונקציה התאמה התאמת שגרתית KRbp חילוץ
- תחת ההנחה כי הריבוכמה שיעור התרגום עם מאוגד RBP הוא קבוע, לדגמן את שיעור הייצור mCherry כדלקמן (ראה איור 2D, הקו הירוק):

כאשר [x] הוא הmCerulean הממוצע המנורמל מחושב על פי Eq .2, שיעור ייצור mCherry הוא הערך המחושב על פי Eq .3, KRbp הוא הזיקה קשירה היחסי [a.u.], k לא מאוגד הוא הריבוכמה קצב של תרגום עם RBP לא מאוגד, n הוא הגורם הקופרקטיביות, ו-C הוא הזריחה הבסיסית [a.u.]. C, n, Kלא מאוגד, ו Krbp נמצאים על ידי התאמת נתוני שיעור הייצור Mcherry למודל (Eq. 4). - באמצעות תוכנה לניתוח נתונים, לנהל הליך הולם על מגרשים המתארים שיעור ייצור mCherry כפונקציה של הממוצע mCerulean (שלב 3.3), ולחלץ את הפרמטרים להתאים בהתאם לנוסחה Eq .4.
הערה: רק תוצאות מתאימים עם R2 > 0.6 נלקחים בחשבון. עבור אלה מתאימים, KRbp שגיאה היא בעיקר בטווח של 0.5% כדי 20% של ערכי Krbp , עבור מרווח ביטחון 0.67, בעוד אלה עם שגיאה krbp גבוהה יותר ניתן לאמת גם על ידי עין. - לנרמל ערכי KRbp לפי הערך המקסימלי המתאים של הממוצע mCerulean עבור כל פונקציה תגובה מינון.
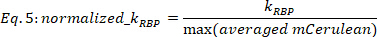
כאשר KRbp ב [a.u.] הוא הערך שחולצו מתוך הליך המדידה Eq. 4, ומקסימום (בממוצע mCerulean) הוא האות המקסימלי mCerulean הממוצע [a. u] שנצפו את המתח הנוכחי.
הערה: הנורמליזציה מאפשרת השוואה נכונה של ההשפעה התקינה על פני זנים על-ידי ביטול התלות ברמות הביטוי המקסימלי RBP בפרט.
תוצאות
השיטה המוצגת מנצלת את התחרות בין RBP לבין הריבוחלק לכריכה למולקולה mRNA (איור 1). תחרות זו משתקפת על ידי הפחתת רמות mCherry כפונקציה של ייצור מוגבר של RBP-mCerulean, בשל ריכוזי הגוברת של הסליל. במקרה של הגדלת mCerulean פלואורסצנטית, ללא שינויים משמעותיים ב-mCherry, חוסר של כריכת ...
Discussion
השיטה המתוארת במאמר זה מקלה כמותית במדידה vivo של RBP-RNA הזיקה הקשירה בתאים E. coli . הפרוטוקול קל יחסית וניתן להתנהל ללא שימוש במכונות מתוחכמות, וניתוח נתונים הוא פשוט. כמו-כן, התוצאות מיוצרות מיד, ללא הזמן ההמתנה הארוך המשויך לתוצאות רצף הדור הבא (NGS).
מגבלה אחת לשיטה זו היא שזה...
Disclosures
. למחברים אין מה לגלות
Acknowledgements
פרויקט זה קיבל מימון מתוכנית ה-I-CORE של ועדת התכנון והתקצוב והקרן הישראלית למדעים (גרנט No. 152/11), מארי קירי שילוב מחדש של המענק לא. PCIG11-GA-2012-321675, ומאופק האיחוד האירופי 2020 תוכנית מחקר וחדשנות תחת הסכם מענק מס ' 664918-MRG-דקדוק.
Materials
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
References
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved