Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein Assay zur Quantifizierung der Protein-RNA-Bindung in Bakterien
In diesem Artikel
Zusammenfassung
Bei dieser Methode quantifizieren wir die Bindungsaffinität von RNA-bindenden Proteinen (RBPs) zu kognazierenden und nicht-kogatierten Bindungsstellen mit einem einfachen, live, Reporter-Assay in Bakterienzellen. Der Test basiert auf der Unterdrückung eines Reportergens.
Zusammenfassung
Im Initiationsschritt der Proteintranslation bindet das Ribosom an den Initiationsbereich der mRNA. Die Translationsinitiation kann durch Bindung eines RNA-bindenden Proteins (RBP) an den Initiationsbereich der mRNA blockiert werden, der die Ribosome-Bindung stört. In der vorgestellten Methode nutzen wir dieses Blockierungsphänomen, um die Bindungsaffinität von RBPs zu ihren kognitierten und nicht kognierten Bindungsstellen zu quantifizieren. Dazu fügen wir eine Testbindungsstelle im Initiationsbereich einer Reporter-mRNA ein und induzieren die Expression des Test-RBP. Bei der RBP-RNA-Bindung beobachteten wir eine sigmoidale Unterdrückung des Reporterausdrucks als Funktion der RBP-Konzentration. Bei no-affinity oder sehr geringer Affinität zwischen Bindungsseite und RBP wurde keine signifikante Repression beobachtet. Die Methode wird in lebenden Bakterienzellen durchgeführt und erfordert keine teuren oder ausgeklügelten Maschinen. Es ist nützlich für die Quantifizierung und den Vergleich zwischen den Bindungsaffinitäten verschiedener RBPs, die in Bakterien funktionsfähig sind, zu einer Reihe von entworfenen Bindungsstellen. Diese Methode kann für die Bindung von Standorten mit hoher struktureller Komplexität ungeeignet sein. Dies ist auf die Möglichkeit der Unterdrückung der ribosomalen Initiation durch komplexe mRNA-Struktur in Abwesenheit von RBP zurückzuführen, was zu einer niedrigeren Basalreporter-Genexpression und damit zu einer weniger beobachtbaren Reporterrepression auf DIE RBP-Bindung führen würde.
Einleitung
Die RNA-bindende Protein-basierte posttranskriptionelle Regulation, speziell die Charakterisierung der Interaktion zwischen RBPs und RNA, wurde in den letzten Jahrzehnten ausgiebig untersucht. Es gibt mehrere Beispiele für translationale Down-Regulierung bei Bakterien, die von RBPs stammen, die Ribosombindung1,2,3hemmen oder direkt mit ihnen konkurrieren. Im Bereich der synthetischen Biologie entwickeln sich RBP-RNA-Wechselwirkungen zu einem bedeutenden Werkzeug für die Entwicklung von Transkriptionsbasierten genetischen Schaltkreisen4,5. Daher steigt die Nachfrage nach Charakterisierung solcher RBP-RNA-Wechselwirkungen im zellulären Kontext.
Die gebräuchlichsten Methoden zur Untersuchung von Protein-RNA-Wechselwirkungen sind der elektrophoretische Mobilitäts-Shift-Assay (EMSA)6, der auf In-vitro-Einstellungen beschränkt ist, und verschiedene Pull-Down-Assays7, einschließlich der CLIP-Methode8,9 . Während solche Methoden die Entdeckung von de novo RNA-Bindungsstellen ermöglichen, leiden sie unter Nachteilen wie arbeitsintensiven Protokollen und teuren tiefen Sequenzierungsreaktionen und erfordern möglicherweise einen spezifischen Antikörper für RBP-Pull-down. Aufgrund der anfälligen Natur der RNA für ihre Umgebung können viele Faktoren RBP-RNA-Wechselwirkungen beeinflussen, was die Bedeutung der Rekation der RBP-RNA-Bindung im zellulären Kontext unterstreicht. Zum Beispiel haben wir und andere signifikante Unterschiede zwischen RNA-Strukturen in vivo und in vitro10,11gezeigt.
Basierend auf dem Ansatz einer früheren Studie12, haben wir vor kurzem10 gezeigt, dass bei der Platzierung vorgefertigter Bindungsstellen für die Kapsid-RBPs aus den Bakteriophagen GA13, MS214, PP715und Q.16 in der ÜbersetzungInitiationsregion eines Reporters mRNA, Reporter Ausdruck wird stark unterdrückt. Wir präsentieren eine relativ einfache und quantitative Methode, basierend auf diesem Repressionsphänomen, um die Affinität zwischen RBPs und ihrer entsprechenden RNA-Bindungsstelle in vivo zu messen.
Protokoll
1. Systemvorbereitung
- Design von Bindungs-Standort-Plasmiden
- Entwerfen Sie die Bindungsplatzkassette wie in Abbildung 1dargestellt. Jedes Minigen enthält die folgenden Teile (5' bis 3'): Eagl-Restriktionsstelle, 40 Basen des 5'-Endes des Kanamycin-Resistenzgens, pLac-Ara-Promotor, Ribosomenbindungsstelle (RBS), AUG des mCherry-Gens, ein Abstandsabstand , eine RBP-Bindungsstelle, 80 Basen des 5'-Ens des mCherry-Gens und einer ApaLI-Einschränkungsseite.
HINWEIS: Um die Erfolgsrate des Assays zu erhöhen, entwerfen Sie drei Bindungs-Seitenkassetten für jede Bindungsstelle, mit Abstandshaltern, die aus mindestens einer, zwei und drei Basen bestehen. Weitere Richtlinien finden Sie im Abschnitt Repräsentative Ergebnisse.
- Entwerfen Sie die Bindungsplatzkassette wie in Abbildung 1dargestellt. Jedes Minigen enthält die folgenden Teile (5' bis 3'): Eagl-Restriktionsstelle, 40 Basen des 5'-Endes des Kanamycin-Resistenzgens, pLac-Ara-Promotor, Ribosomenbindungsstelle (RBS), AUG des mCherry-Gens, ein Abstandsabstand , eine RBP-Bindungsstelle, 80 Basen des 5'-Ens des mCherry-Gens und einer ApaLI-Einschränkungsseite.
- Klonen von Bindungsstellenplasmiden
- Bestellen Sie die Bindungsplatzkassetten als doppelsträngige DNA(dsDNA) Minigene. Jedes Minigen ist 500 bp lang und enthält eine Eagl-Einschränkungsseite und eine ApaLI-Einschränkungsstelle an den Enden 5' bzw. 3' (siehe Schritt 1.1.1).
HINWEIS: In diesem Experiment wurden Minigene mit der Hälfte des Kanamycin-Gens bestellt, um das Screening auf positive Kolonien zu erleichtern. Allerdings eignet sich hier auch die Gibson-Baugruppe17, in diesem Fall kann die Bindungsstelle als zwei kürzere komplementäre einsträngige DNA-Oligos bestellt werden. - Doppelverdauen Sie sowohl die Minigene als auch den Zielvektor mit Eagl-HF und ApaLI durch das Restriktionsprotokoll18und Spaltenreinigung19.
- Ligate die verdauten Minigene an das Bindungs-Standort-Rückgrat, das den Rest des mCherry-Reportergens, Terminator und ein Kanamycin-Resistenzgen20enthält.
- Verwandeln Sie die Ligationslösung in Escherichia coli TOP10 Zellen21.
- Identifizieren Sie positive Transformationsmittel über die Sanger-Sequenzierung.
- Entwerfen Sie eine Grundierung 100 Basen vor dem Interessengebiet (siehe Tabelle 1 für Primersequenzen).
- Miniprep ein paar Bakterienkolonien22.
- Bereiten Sie 5 l einer 5 mM-Lösung des Primers und 10 l der DNA bei 80 ng/l Konzentration vor.
- Senden Sie die beiden Lösungen an eine praktische Einrichtung für Sanger-Sequenzierung23.
- Speichern Sie gereinigte Plasmide bei -20 °C und Bakterienstämme als Glycerinbestände24, beide im 96-Well-Format. DIE DNA wird dann zur Umwandlung in E. coli TOP10-Zellen verwendet, die eines von vier Fusions-RBP-Plasmiden enthalten (siehe Schritt 1.3.5).
- Bestellen Sie die Bindungsplatzkassetten als doppelsträngige DNA(dsDNA) Minigene. Jedes Minigen ist 500 bp lang und enthält eine Eagl-Einschränkungsseite und eine ApaLI-Einschränkungsstelle an den Enden 5' bzw. 3' (siehe Schritt 1.1.1).
- Entwurf und Konstruktion des RBP-Plasmids
HINWEIS: Aminosäure- und Nukleotidsequenzen der in dieser Studie verwendeten Mantelproteine sind in Tabelle 2aufgeführt.- Bestellen Sie die erforderliche RBP-Sequenz ohne Stop-Codon als benutzerdefiniertes dsDNA-Minigen ohne Stop-Codon mit Restriktionsstellen an den Enden (Abbildung 1).
- Klonen Sie das getestete RBP ohne Stop-Codon unmittelbar nach einem induzierbaren Promotor und vor einem fluoreszierenden Protein ohne Startcodon (Abbildung 1), ähnlich den Schritten 1.2.2-1.2.4. Stellen Sie sicher, dass das RBP-Plasmid ein anderes Antibiotikaresistenzgen enthält als das Bindungs-Standort-Plasmid.
- Identifizieren Sie positive Transformationsmittel über sanger-Sequenzierung, ähnlich wie Schritt 1.2.5 (siehe Tabelle 1 für Primersequenzen).
- Wählen Sie ein positives Transformant und machen Sie es chemisch kompetent25. Als Glycerin-gereinigte Plasmide bei -20 °C und Glycerinbestände von Bakterienstämmen24 bei -80 °C in 96-Well-Platten lagern.
- Verwandeln Sie die in 96-Well-Platten gelagerten Bindungs-Standort-Plasmide (ab Schritt 1.2.6) in chemisch kompetente Bakterienzellen, die bereits ein RBP-mCerulean-Plasmid21enthalten. Um Zeit zu sparen, anstatt die Zellen auf Petrischalen zu beschichten, bedecken Sie sie mit einem 8-Kanal-Pipettor auf 8-spurigen Platten mit Luria-Bertani (LB)26 Agar mit relevanten Antibiotika (Kan und Amp). Kolonien sollten in 16 h erscheinen.
- Wählen Sie für jedes Doppel-Transformant eine einzelne Kolonie aus und wachsen Sie über Nacht im LB-Medium mit den entsprechenden Antibiotika (Kan und Amp) und lagern Sie als Glycerinbestände24 bei -80 °C in 96-Well-Platten.
2. Experiment-Setup
HINWEIS: Das hier vorgestellte Protokoll wurde mit einem Flüssigkeitshandling-Robotersystem in Kombination mit einem Inkubator und einem Plattenleser durchgeführt. Jede Messung wurde für 24 Induktorkonzentrationen mit zwei Duplikaten für jede Dehnung + Induktor-Kombination durchgeführt. Mit diesem Robotersystem wurden Daten für 16 Stämme pro Tag mit 24 Induktorkonzentrationen gesammelt. Wenn ein solches Gerät jedoch nicht verfügbar ist oder weniger Experimente erforderlich sind, können diese einfach von Hand mit einer 8-Kanal-Multipipetette durchgeführt werden und das Protokoll entsprechend anpassen. So wurden beispielsweise vorläufige Ergebnisse für vier Stämme pro Tag mit 12 Induktorenkonzentrationen und vier Zeitpunkten auf diese Weise ermittelt.
- 1 L Bioassaypuffer (BA) im Voraus durch Mischen von 0,5 g Trypton, 0,3 ml Glycerin, 5,8 g NaCl, 50 ml 1 M MgSO4,1 ml 10x phosphatgepufferter Kochsalzlösung (PBS) pH 7,4 und 950 ml Doppeldestilliertem Wasser (DDW) vorbereiten. Autoklav oder steriler Filter des BA-Puffers.
- Die doppeltransformierenden Stämme bei 37 °C und 250 Umdrehungen pro Minute in 1,5 ml LB mit geeigneten Antibiotika (Kanamycin in einer Endkonzentration von 25 g/ml und Ampicillin bei einer Endkonzentration von 100 g/ml) in 48-Well-Platten über einen Zeitraum von 18 h (Übernachtung) anbauen.
-
Am Morgen die folgenden Vorbereitungen treffen.
- Induktorplatte. In einer sauberen 96-Well-Platte Brunnen mit halbarmem Medium (SPM) zubereiten, bestehend aus 95% BA und 5% LB26 im Inkubator bei 37 °C. Die Anzahl der Brunnen entspricht der gewünschten Anzahl von Induktorkonzentrationen. Fügen Sie C4-HSL zu den Brunnen in der Induktorplatte hinzu, die die höchste Induktorkonzentration (218 nM) enthalten.
- Programmieren Sie den Roboter, um das Medium von jedem der hochkonzentrierten Brunnen in 23 niedrigere Konzentrationen von 0 bis 218 nM zu verdünnen. Das Volumen jeder Induktorverdünnung sollte für alle Stämme (einschließlich Duplikate) ausreichen.
- Während die Induktorenverdünnungen vorbereitet werden, 180 l SPM im Inkubator bei 37 °C in 96-Well-Platten warm.
- Verdünnen Sie die Übernachtstämme von Schritt 2.2 um den Faktor 100 durch serielle Verdünnungen: zuerst um den Faktor 10 verdünnen, indem Sie 100 l Bakterien mit 900 l SPM in 48-Well-Platten mischen und dann um den Faktor 10 erneut verdünnen, indem 20 l aus der verdünnten Lösung in 180 l vorgewärmte SPM, in 96-Well-Platten geeignet für fluoreszierende Messungen.
- Fügen Sie den verdünnten Induktor von der Induktorplatte zu den 96-Well-Platten mit den verdünnten Stämmen entsprechend den Endkonzentrationen hinzu.
- Schütteln Sie die 96-Well-Platten bei 37 °C für 6 h, während Sie Messungen der optischen Dichte bei 595 nm (OD595), mCherry (560 nm/612 nm) und mCerulean (460 nm/510 nm) Fluoreszenz über einen Plattenleser alle 30 min. Messen Sie für Normalisierungszwecke das Wachstum von SMP ohne Zellen.
3. Vorläufige Ergebnisanalyse
- Wählen Sie für jeden Experimenttag ein Zeitintervall des logarithmischen Wachstums entsprechend den gemessenen Wachstumskurven zwischen der linearen Wachstumsphase und der stationären (T0, Tfinal). Nehmen Sie etwa 6 bis 8 Zeitpunkte, während Sie die erste und letzte Messung verwerfen, um Fehler zu vermeiden, die durch Ungenauigkeiten der exponentiellen Wachstumserkennung abgeleitet werden (siehe Abbildung 2A, oberes Panel).
HINWEIS: Verwerfen Sie Stämme, die abnormale Wachstumskurven oder Dehnungen aufweisen, bei denen die logarithmische Wachstumsphase nicht erkannt werden konnte, und wiederholen Sie das Experiment. -
Berechnen Sie die durchschnittliche normalisierte Fluoreszenz von mCerulean und die Produktionsrate von mCherry aus den Rohdaten von mCerulean und mCherry Fluoreszenz für jede Induktorkonzentration (Abbildung 2A).
- Berechnen Sie normalisiertem mCerulean wie folgt:
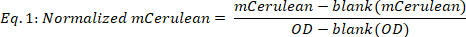
wobei blank(mCerulean) die mCerulean-Ebene [a.u.] nur für Medium ist, blank(OD) die optische Dichte nur für Medium und mCerulean und OD die mCerulean-Fluoreszenz- bzw. optischedichte Dichtewerte sind. - Durchschnittlicher mCerulean über die verschiedenen Zeitpunkte (Abbildung2B, zwei Top-Panels) wie folgt:
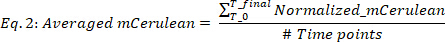
wobei #Time Punkte die Anzahl der berücksichtigten Datenzeitpunkte ist, T0 der Zeitpunkt, zu dem die exponentielle Wachstumsphase beginnt, und Tfinal der Zeitpunkt, zu dem die exponentielle Wachstumsphase endet. - Berechnen Sie die mCherry-Produktionsrate (Abbildung2B, untere zwei Panels) wie folgt:
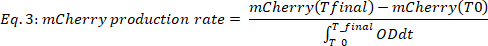
wobei mCherry(t) die mCherry-Ebene [a.u.] zum Zeitpunkt t, OD der optische Dichtewert, T0 der Zeitpunkt ist, zu dem die exponentielle Wachstumsphase beginnt, und Tfinal ist der Zeitpunkt, zu dem die exponentielle Wachstumsphase endet.
- Berechnen Sie normalisiertem mCerulean wie folgt:
- Schließlich zeichnen Sie die mCherry-Produktionsrate als Funktion von mCerulean, wodurch Dosis-Antwort-Kurven in Abhängigkeit von RBP-mCerulean-Fusionsfluoreszenz erstellt werden (Abbildung 2C). Solche Plots stellen die Produktion des Reportergens als Funktion der RBP-Präsenz in der Zelle dar.
4. Dosis-Response-Funktion Fitting Routine und K RBP-Extraktion
- Unter der Annahme, dass die Ribosomrate der Übersetzung mit der RBP-Gebunden konstant ist, modellieren Sie die mCherry-Produktionsrate wie folgt (siehe Abbildung 2D,grüne Linie):

wobei [x] die normalisierte durchschnittliche mCerulean-Fluoreszenz ist, berechnet nach Eq. 2, mCherry Produktionsrate ist der Wert, der nach Eq. 3 berechnet wird, KRBP ist die relative Bindungsaffinität [a.u.], Kungebunden ist die Ribosomrate von Übersetzung mit dem RBP ungebunden, n ist der Kooperatoritätsfaktor, und C ist die Basisfluoreszenz [a.u.]. C, n, Kunboundund KRBP werden gefunden, indem die mCherry Produktionsratendaten an das Modell anpasst (Eq. 4). - Mit Hilfe der Datenanalyse-Software führen Sie ein Anpassungsverfahren auf Diagrammen durch, die die mCherry-Produktionsrate als Funktion von gemittelten mCerulean (Schritt 3.3) darstellen, und extrahieren Sie die Anpassungsparameter gemäß der Formel in Eq. 4.
HINWEIS: Berücksichtigt werden nur passende Ergebnisse mit R2 > 0,6. Für diese Passte, KRBP Fehler ist meist im Bereich von 0,5% bis 20% der K RBP-Werte, für ein 0,67 Konfidenzintervall, während diejenigen mit höheren KRBP Fehler kann auch durch Auge überprüft werden. - Normalisieren Sie Die K-RBP-Werte um den jeweiligen Maximalwert von gemittelt emCerulean für jede Dosis-Wirkungs-Funktion.
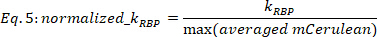
wobei KRBP in [a.u.] der Wert ist, der aus dem Fitting-Verfahren in Eq. 4 extrahiert wird, und max (gemittelt mCerulean) das maximal gemittelte mCerulean-Signal [a.u], das für den aktuellen Stamm beobachtet wird.
HINWEIS: Die Normalisierung erleichtert den korrekten Vergleich der regulatorischen Wirkung über Stämme hinweg, indem die Abhängigkeit von den jeweiligen maximalen RBP-Expressionsebenen beseitigt wird.
Ergebnisse
Die vorgestellte Methode nutzt den Wettbewerb zwischen einem RBP und dem Ribosom zur Bindung an das mRNA-Molekül (Abbildung 1). Dieser Wettbewerb spiegelt sich in sinkenden mCherry-Spiegeln als Funktion der erhöhten Produktion von RBP-mCerulean wider, aufgrund steigender Konzentrationen von Induktoren. Im Falle einer zunehmenden mCerulean-Fluoreszenz, ohne signifikante Änderungen in mCherry, wird ein Mangel an RBP-Bindung abgeleitet. Repräsentative Ergebn...
Diskussion
Die in diesem Artikel beschriebene Methode erleichtert die quantitative In-vivo-Messung der RBP-RNA-Bindungsaffinität in E. coli-Zellen. Das Protokoll ist relativ einfach und kann ohne den Einsatz ausgeklügelter Maschinen durchgeführt werden, und die Datenanalyse ist einfach. Darüber hinaus werden die Ergebnisse sofort erstellt, ohne die relativ lange Wartezeit, die mit den Ergebnissen der Next Generation Sequencing (NGS) verbunden ist.
Eine Einschränkung dieser Methode ist, dass...
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Dieses Projekt wurde aus dem I-CORE-Programm des Planungs- und Budgetierungsausschusses und der Israel Science Foundation (Grant No. 152/11), Marie Curie Reintegration Grant No. PCIG11-GA- 2012-321675 und aus dem Forschungs- und Innovationsprogramm Horizont 2020 der Europäischen Union im Rahmen der Finanzhilfevereinbarung Nr. 664918 - MRG-Grammar.
Materialien
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
Referenzen
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten