JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
박테리아에서 단백질 RNA 결합을 정량화하기위한 분석
요약
이 방법에서, 우리는 세균 세포에서 단순, 살아있는, 리포터 분석법을 사용하여 동축 및 비동종 결합 부위에 RNA 결합 단백질(RBPs)의 결합 친화도를 정량화한다. 분석은 기자 유전자의 억압에 근거를 둔다.
초록
단백질 번역의 개시 단계에서, 리보솜은 mRNA의 개시 부위에 결합한다. 번역 개시는 리보솜 결합을 방해하는 mRNA의 개시 지구에 RNA 결합 단백질 (RBP)의 결합에 의해 차단될 수 있습니다. 제시된 방법에서는, 우리는 그들의 동체 및 비 cognate 결합 사이트에 대한 RP의 결합 친화도를 정량화하기 위해 이 차단 현상을 이용한다. 이를 위해, 우리는 리포터 mRNA의 개시 부위에 시험 결합 부위를 삽입하고 시험 RBP의 발현을 유도한다. RBP-RNA 결합의 경우, 우리는 RBP 농도의 함수로서 리포터 발현의 시그노이드 억압을 관찰했다. 결합 부위와 RBP 사이의 친화성 또는 매우 낮은 친화력의 경우, 유의한 억압이 관찰되지 않았다. 이 방법은 살아있는 세균 세포에서 수행되며 비싸거나 정교한 기계가 필요하지 않습니다. 박테리아에서 기능하는 상이한 RP의 결합 친화도를 설계된 결합 부위세트에 정량화하고 비교하는 데 유용합니다. 이 방법은 구조적 복잡성이 높은 사이트를 바인딩하는 데 적합하지 않을 수 있습니다. 이는 RBP가 없는 복잡한 mRNA 구조에 의한 리보좀 개시의 억제 가능성, 이는 낮은 기저 리포터 유전자 발현을 초래할 것이고, 따라서 RBP 결합에 대한 관찰력이 떨어지는 리포터 억압에 기인한다.
서문
RNA 결합 단백질(RBP)-기반 전사 후 조절, 특히 RBPs와 RNA 사이의 상호작용의 특성화는 최근 수십 년 동안 광범위하게 연구되고 있다. RBPs 억제에서 유래 하는 박테리아에서 번역 다운 조절의 여러 예가 있다, 또는직접 경쟁, 리보솜 바인딩 1,2,3. 합성 생물학 분야에서, RBP-RNA 상호 작용은 전사 기반 유전 회로4,5의설계를위한 중요한 도구로 부상하고 있다. 따라서, 세포 문맥에서 이러한 RBP-RNA 상호작용의 특성화에 대한 수요가 증가하고 있다.
단백질 RNA 상호 작용을 연구하기 위한 가장 일반적인 방법은 시험관내 설정으로 제한되는 전기동이동성 시프트 분석법(EMSA)6,및 CLIP 방법8,9를 포함한 다양한 풀다운 분석7이다. . 이러한 방법은 de novo RNA 결합 부위의 발견을 가능하게 하는 동안, 그들은 노동 집약적인 프로토콜 및 비싼 깊은 시퀀싱 반응과 같은 결점 때문에 손해를 입고 RBP 풀다운을 위한 특정 항체를 요구할 수 있습니다. 그것의 환경에 RNA의 영향을 받기 쉬운 특성 때문에, 많은 요인은 세포 문맥에서 RBP-RNA 결합을 심문하는 의 중요성을 강조하는 RBP-RNA 상호 작용에 영향을 미칠 수 있습니다. 예를 들어, 우리 및 그 외는 생체내 RNA 구조와 체외10,11사이의 유의한 차이를 입증하였다.
이전 연구12의접근법에 기초하여, 우리는 최근에 박테리오파지 GA13,MS214,PP715및 Qβ16에서 캡시드 RBP에 대한 미리 설계된 결합 부위를 배치할 때 10을 입증했다. [mRNA] 기자의 번역 개시 부위, 리포터 표현이 강하게 억압된다. 우리는 이 억압 현상에 근거하여, RBP와 그들의 대응하는 RNA 결합부위의 생체 내 친화성을 측정하기 위해 비교적 간단하고 정량적인 방법을 제시한다.
프로토콜
1. 시스템 준비
- 바인딩 사이트 플라스미드 설계
- 그림1에 설명된 대로 바인딩 사이트 카세트를 디자인합니다. 각 미니유전자는 다음과 같은 부분(5' ~ 3')을 포함하며, Eagl 제한 부위, +40 기저의 카나마이신(Kan) 저항 유전자, pLac-Ara 프로모터, 리보솜 결합 부위(RBS), mCherry 유전자의 AUG, 스페이서(δ), RBP 결합 부위, 5' 말단의 80염기 mCherry 유전자, 및 ApaLI 제한 부위.
참고: 분석의 성공률을 높이기 위해 각 바인딩 부위에 대해 3개의 바인딩 사이트 카세트를 설계하고 스페이서가 적어도 1개, 2개 및 3개의 염기로 구성됩니다. 자세한 지침은 대표 결과 섹션을 참조하십시오.
- 그림1에 설명된 대로 바인딩 사이트 카세트를 디자인합니다. 각 미니유전자는 다음과 같은 부분(5' ~ 3')을 포함하며, Eagl 제한 부위, +40 기저의 카나마이신(Kan) 저항 유전자, pLac-Ara 프로모터, 리보솜 결합 부위(RBS), mCherry 유전자의 AUG, 스페이서(δ), RBP 결합 부위, 5' 말단의 80염기 mCherry 유전자, 및 ApaLI 제한 부위.
- 바인딩 사이트 플라스미드의 복제
- 결합 부위 카세트를 이중 가닥 DNA(dsDNA) 미니유전자로 주문합니다. 각 미니유전자는 ~500 bp 길이이며, 각각 5' 및 3' 말단에 Eagl 제한 사이트 및 ApaLI 제한 사이트를 함유하고 있다(1.1.1단계 참조).
참고: 본 실험에서, 카나마이신 유전자의 절반을 가진 미니 유전자는 양성 콜로니에 대한 스크리닝을 용이하게 하기 위해 주문하였다. 그러나, 깁슨 어셈블리(17)는 또한 여기에 적합하며, 이 경우 결합 부위는 2개의 짧은 상보적인 단일 가닥 DNA 올리고스로서 주문될 수 있다. - 제한 프로토콜18에의해 Eagl-HF 및 ApaLI를 가진 미니 유전자 및 표적 벡터 둘 다 이중 소화하고, 컬럼 은19를정제한다.
- mCherry 리포터 유전자, 터미네이터 및 카나마이신 내성 유전자(20)의 나머지를 포함하는 결합 부위백본에 소화된 미니유전자를 리게이트.
- 결찰 액을 대장균 TOP10세포(21)로변형시킨다.
- Sanger 시퀀싱을 통해 양성 변압자를 식별합니다.
- 관심 영역까지 상류에 프라이머(100) 염기를 설계합니다(프라이머 서열에 대한 표 1 참조).
- 미니 프렙 몇 가지 세균 식민지22.
- 프라이머의 5 mM 용액의 5 μL및 80 ng/μL 농도에서 DNA의 10 μL을 준비한다.
- Sanger 시퀀싱23을위한 편리한 시설로 두 가지 솔루션을 보냅니다.
- 정제된 플라스미드를 -20°C에서 저장하고, 글리세롤이24를스톡함에 따라 세균 균주를 96웰 형식으로 저장한다. DNA는 이어서 4개의 융합-RBP 플라스미드 중 하나를 함유하는 대장균 TOP10 세포로의 형질전환을 위해 사용될 것이다(단계 1.3.5 참조).
- 결합 부위 카세트를 이중 가닥 DNA(dsDNA) 미니유전자로 주문합니다. 각 미니유전자는 ~500 bp 길이이며, 각각 5' 및 3' 말단에 Eagl 제한 사이트 및 ApaLI 제한 사이트를 함유하고 있다(1.1.1단계 참조).
- RBP 플라스미드의 설계 및 시공
참고: 본 연구에 사용된 외투 단백질의 아미노산 및 뉴클레오티드 서열은 표2에 열거되어 있다.- 필요한 RBP 서열을 주문하여 정지 코돈이 결여된 정지 코돈이 단말에 제한 부위가있는 스톱 코돈이 결여된 맞춤형 dsDNA 미니유전자를 주문한다(도 1).
- 시험된 RBP를 복제하여 유도성 프로모터의 바로 하류및 시작 코돈이 결여된 형광 단백질의 상류하류에 정지 코돈이 결여된 것을 복제(도1),단계 1.2.2-1.2.4와 유사. RBP 플라스미드가 결합 부위 플라스미드와는 다른 항생 저항 유전자를 포함하고 있는지 확인한다.
- Sanger 시퀀싱을 통해 양성 변압자를 식별합니다(1.2.5단계와 유사).(프라이머 시퀀스의 경우 표 1 참조).
- 하나의 긍정적 인 변형을 선택하고 화학적으로 유능한25합니다. -20°C에서 글리세롤 정제 플라스미드로 보관하고, 96웰 플레이트에서 -80°C에서 세균 균주24의 글리세롤 스톡을 저장한다.
- 96웰 플레이트에 저장된 결합 부위 플라스미드(단계 1.2.6)를 이미 RBP-mCerulean 플라스미드(21)를함유하고 있는 화학적으로 유능한 세균 세포로 변형시킨다. 시간을 절약하기 위해 페트리 접시에 세포를 도금하는 대신 관련 항생제 (칸 및 Amp)가있는 루리아 베르타니 (LB)26 한천을 함유 한 8 채널 파이프터를 사용하여 접시에 담습니다. 식민지는 16 시간 안에 나타나야합니다.
- 각 이중 형질전환제에 대한 단일 콜로니를 선택하고 관련 항생제(Kan and Amp)로 LB 배지에서 하룻밤 동안 성장시키고 96웰 플레이트에서 -80°C에서 글리세롤 스톡24로 저장합니다.
2. 실험 설정
참고: 여기에 제시된 프로토콜은 인큐베이터 및 플레이트 리더와 함께 액체 처리 로봇 시스템을 사용하여 수행하였다. 각 측정은 24개의 유도자 농도에 대해 수행되었고, 각 스트레인 + 유도기 조합에 대해 2개의 중복이 있었다. 이 로봇 시스템을 사용하여 하루에 16 개의 균주에 대한 데이터를 24 유도체 농도로 수집했습니다. 그러나 이러한 장치를 사용할 수 없거나 실험이 더 적은 경우 8채널 멀티 파이펫을 사용하여 프로토콜을 적절하게 조정하여 수작업으로 쉽게 수행할 수 있습니다. 예를 들어, 12개의 유도자 농도와 4개의 시간 포인트를 가진 4개의 균주에 대한 예비 결과가 이런 식으로 획득되었다.
- 미리, 바이오분석완충제(BA) 1L의 트립톤 0.5 g, 글리세롤 0.3 mL, NaCl 5.8 g, 1 MMgSO4의50 mL, 10x 인산완충식염수(PBS) 완충액 pH 7.4, 및 950 mL의 이중 DW(diStillD)를 혼합하여 준비한다. BA 버퍼를 오토클레이브 또는 멸균 필터로 필터링합니다.
- 적절한 항생제 (100 μg / mL의 최종 농도에서 25 μg / mL및 ampicillin의 최종 농도에서 카나마이신)와 함께 1.5 mL LB에서 37 °C 및 250 rpm에서 흔들리는 이중 형질전환 균주를 48 웰 플레이트에서 18 시간 (하룻밤)의 기간 동안 성장시킵니다.
-
아침에 다음 을 준비하십시오.
- 유도판. 깨끗한 96웰 플레이트에서, 37°C에서 인큐베이터에서 95% BA 및 5% LB26으로 구성된 반불량 배지(SPM)로 우물을 준비한다. 웰의 수는 유도자 농도의 원하는 수에 해당합니다. 가장 높은 유도자 농도(218 nM)를 포함하는 유도판의 웰에 C4-HSL을 첨가한다.
- 로봇을 0에서 218 nM에 이르는 23개의 낮은 농도로 각각의 고농도 우물에서 매질을 연속적으로 희석하도록 프로그래밍합니다. 각 유도기 희석의 부피는 모든 균주(중복 포함)에 충분해야 합니다.
- 유도기 희석이 제조되는 동안, 96웰 플레이트에서 37°C에서 인큐베이터에서 SPM의 180 μL을 따뜻하게 한다.
- 2.2 단계에서 하룻밤 균주를 연속 희석에 의해 100 의 요인으로 희석 : 먼저 48 웰 플레이트에서 SPM의 900 μL과 박테리아 100 μL을 혼합하여 10 의 요인으로 희석 한 다음 희석 된 용액에서 20 μL을 복용하여 다시 10 배로 희석하십시오. 형광 측정에 적합한 96웰 플레이트에서 180 μL의 사전 따뜻화 된 SPM.
- 유도판으로부터 희석된 유도기를 최종 농도에 따라 희석된 균주를 가진 96웰 플레이트에 첨가한다.
- 37°C에서 37°C에서 6시간 동안 96웰 플레이트를 흔들면서 30분마다 플레이트 리더를 통해 595 nm(OD595),mCherry(560 nm/612 nm) 및 mCerulean(460 nm/510 nm)에서 광학 밀도를 측정합니다. 정규화를 위해 셀이 첨가되지 않은 SMP의 성장을 측정합니다.
3. 예비 결과 분석
- 실험의 각 일에 대해, 선형 성장 단계와 고정된(T 0, T최종)사이의 측정된성장 곡선에 따라 로그 성장의 시간 간격을 선택한다. 지수 성장 감지의 부정확성에서 파생된 오류를 방지하기 위해 첫 번째 및 마지막 측정값을 폐기하면서 약 6~8개의 시간 지점을 취합니다(그림 2A,상단 패널 참조).
참고: 비정상적인 성장 곡선 또는 로그 성장 단계를 감지할 수 없는 균주를 보여주는 균주를 버리고 실험을 반복합니다. -
mCerulean의 평균 정규화 형광 및 mCherry의 생산 속도, 각 유도체 농도에 대한 mCerulean 및 mCherry 형광의 원시 데이터로부터 계산 (도2A).
- 다음과 같이 정규화된 mCerulean을 계산합니다.
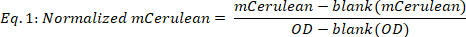
여기서 블랭크(mCerulean)는 mCerulean 수준[a.u.]이 중간 값에만, 블랭크(OD)는 중간 값에만 광학 밀도이고, mCerulean 및 OD는 각각 mCerulean 형광 및 광학 밀도 값입니다. - 서로 다른 시간 포인트(그림2B,상위 2개 패널)에 대한 평균 mCerulean은 다음과 같습니다.
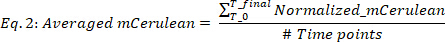
#Time 포인트가 고려되는 데이터 타임포인트의 수인 경우 T0은 지수 증가 단계가 시작되는 시간이며 Tfinal은 지수 증가 단계가 끝나는 시간입니다. - m체리 생산속도 계산(그림2B,하단 2개 패널) 다음과 같다.
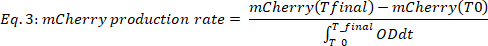
여기서 mCherry(t)는 시간 t에서 mCherry 수준[a.u.]이고, OD는 광학 밀도 값이고, T 0은 지수 성장 단계가 시작되는 시간이며, Tfinal은 지수 성장 단계가 끝나는 시간입니다.
- 다음과 같이 정규화된 mCerulean을 계산합니다.
- 마지막으로, mCerulean의 함수로서 생산의 mCherry 비율을 플롯하고, RBP-mCerulean 융합 형광의 함수로서투여량 응답 곡선을 생성한다(도 2C). 이러한 플롯은 세포내 RBP 존재의 함수로서 리포터 유전자의 생산을 나타낸다.
4. 복용량 응답 기능 피팅 루틴 및 KRBP 추출
- RBP 바운드를 가진 번역의 리보솜 비율이 일정하다는 가정 하에, mCherry 생산 속도를 다음과 같이 모델링합니다(그림 2D, 녹색 선 참조).

여기서 [x]는 Eq. 2에 따라 계산된 정규화된 평균 mCerulean 형광이, mCherry 생산률은 Eq. 3에 따라 계산된 값이며, KRBP는 상대적 결합 친화성 [a.u.], K언바운드는 리보솜 비율이다. RBP 언바운드로 변환, n은 협력 계수이고, C는 기본 형광 [a.u.]입니다. C, n, K언바운드및 KRBP는 mCherry 생산 속도 데이터를 모델(Eq. 4)에 피팅하여 발견됩니다. - 데이터 분석 소프트웨어를 사용하여 mCherry 생산 속도를 평균 mCerulean(단계 3.3)의 함수로 묘사하는 플롯에 피팅 절차를 수행하고 Eq. 4의 수식에 따라 맞춤 매개변수를 추출합니다.
참고: R2 > 0.6을 가진 피팅 결과만 고려됩니다. 이러한 적합의 경우, KRBP 오차는 주로 KRBP 값의 0.5%에서 20% 범위이며, 0.67 신뢰 구간의 경우 KRBP 오류가 높은 값도 눈으로 확인할 수 있습니다. - KRBP 값을 각 투여량-응답 함수에 대한 평균 mCerulean의 각각 최대 값으로 정규화합니다.
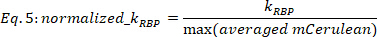
여기서 [a.u.]의 KRBP는 Eq. 4의 피팅 프로시저로부터 추출된 값이며, 최대(평균 mCerulean)는 현재 변형에 대해 관찰된 최대 평균 mCerulean 신호[a.u]이다.
참고: 정규화는 특정 최대 RBP 발현 수준에 대한 의존도를 제거하여 균주 전반에 걸친 조절 효과의 정확한 비교를 용이하게 합니다.
결과
제시된 방법은 mRNA 분자에 결합하기 위한 RBP와 리보솜사이의 경쟁을 이용한다(도 1). 이 경쟁은 유도제의 농도 증가로 인해 RBP-mCerulean의 증가 생산의 함수로 mCherry 수준을 감소시킴으로써 반영됩니다. mCherry에 큰 변화가 없는 mCerulean 형광이 증가하는 경우, RBP 결합의 부족은 추론된다. 양수 및 음의 변형모두에 대한 대표적인 결과는 그...
토론
본 항에 기술된 방법은 대장균 세포에서 RBP-RNA 결합 친화도의 생체 내 정량적 측정을 용이하게 한다. 이 프로토콜은 비교적 쉽고 정교한 기계를 사용하지 않고도 수행될 수 있으며 데이터 분석은 간단합니다. 더욱이, 결과는 차세대 염기서열 분석(NGS) 결과와 관련된 비교적 긴 대기 시간 없이 즉시 생성된다.
이 방법의 한 가지 제한은 세균 세포에서만 작동한다는 것입?...
공개
저자는 공개 할 것이 없다.
감사의 말
이 프로젝트는 기획 및 예산 위원회의 I-CORE 프로그램과 이스라엘 과학 재단 (그랜트 번호 152/11), 마리 퀴리 재통합 보조금 번호에서 자금을 받았다. PCIG11-GA- 2012-321675, 및 보조금 계약 번호 664918에 따라 유럽 연합 (EU)의 호라이즌 2020 연구 및 혁신 프로그램에서 - MRG-문법.
자료
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
참고문헌
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유