Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Анализ для количественной протеино-РНК Связывания в бактериях
В этой статье
Резюме
В этом методе мы количественно связываем связывающее сродство связывающих белков РНК (RBPs) к cognate и неcognate связывающих сайтов с помощью простой, живой, репортер анализ в бактериальных клетках. Ассем основан на подавлении гена репортера.
Аннотация
В шаге начала перевода протеина, ribosome связывает к зоне начала mRNA. Инициация перевода может быть заблокирована путем привязки связывающего белка РНК (RBP) к области инициации мРНК, которая мешает рибосомной связывания. В представленном методе мы используем это блокирующее явление для количественной оценки связывающей близости РБП к их cognate и не-cognate связывающих сайтов. Для этого мы вставляем тестовый узел связывания в область инициации репортера mRNA и индуцируем выражение теста RBP. В случае связывания RBP-РНК мы наблюдали сигмоидальные репрессии в отношении выражения репортера как функции концентрации РБП. В случае несродности или очень низкого сродства между связывающим сайтом и РБП не наблюдалось никаких существенных репрессий. Метод проводится в живых бактериальных клетках и не требует дорогостоящего или сложного оборудования. Это полезно для количественной оценки и сравнения связывающих сходств различных RBPs, которые функционируют в бактериях, чтобы набор разработанных мест связывания. Этот метод может быть неподходящим для связывающих сайтов с высокой структурной сложностью. Это должно к возможности репрессии ribosomal инициации сложной структурой mRNA в отсутствии RBP, которое привело бы к в более низком базальном экспрессии гена репортера, и таким образом более менее-наблюдаемых репрессиях репортера на связывании RBP.
Введение
В последние десятилетия широко изучалась регулировка после транскрипционного регулирования, основанного на РНК-связывающем белке (RBP), в частности, характеристика взаимодействия между РСП и РНК. Есть несколько примеров переводного вниз регулирования бактерий, происходящих из RBPs ингибирования, или непосредственно конкурировать с, рибосома связывания1,2,3. В области синтетической биологии, RBP-РНК взаимодействия становятся важным инструментом для разработки транскрипции на основе генетических схем4,5. Таким образом, растет спрос на характеристику таких взаимодействий RBP-RNA в клеточном контексте.
Наиболее распространенными методами для изучения белково-РНК взаимодействий являются электрофоретические передвижные переплет анализа (EMSA)6, который ограничивается в настройках пробирки, и различные выдвижные анализы7, в том числе метод CLIP8,9 . Хотя такие методы позволяют обнаружить de novo РНК связывания сайтов, они страдают от недостатков, таких как трудоемкие протоколы и дорогие глубокие реакции секвенирования и может потребовать конкретного антитела для вытягивания RBP. Из-за восприимчивого характера РНК к окружающей среде, многие факторы могут повлиять на взаимодействие RBP-РНК, подчеркивая важность допроса РБП-РНК связывания в клеточном контексте. Например, мы и другие продемонстрировали значительные различия между структурами РНК in vivo и in vitro10,11.
Основываясь на подходе предыдущего исследования12, мы недавно продемонстрировали10, что при размещении предварительно разработанных обязательных сайтов для капсида RBPs от бактериофагов GA13, MS214, PP7,иNo 16 в перевод инициации области репортера мРНК, репортер выражение сильно репрессированных. Мы представляем относительно простой и количественный метод, основанный на этом феномене репрессий, для измерения сродства между RBPs и их соответствующим ими связывающим сайтом РНКs in vivo.
протокол
1. Подготовка системы
- Дизайн обязательно-участок плазмиды
- Дизайн связывающей кассеты сайта, как показано на рисунке 1. Каждый миниген содержит следующие части (5' до 3'): Место ограничения Eagl, 40 оснований 5' конца гена сопротивления канамицина (Кан), промоутер pLac-Ara, место связывания рибосом (RBS), AUG гена mCherry, прокладка (я), место связывания RBP, 80 оснований 5' конца гена mCherry и места ограничения ApaLI.
ПРИМЕЧАНИЕ: Чтобы увеличить частоту асссея, разработай три связывающих кассеты для каждого связывающего участка, с помощью прокладок, состоящих по крайней мере из одной, двух и трех баз. Дополнительные рекомендации по результатам работы с представителями.
- Дизайн связывающей кассеты сайта, как показано на рисунке 1. Каждый миниген содержит следующие части (5' до 3'): Место ограничения Eagl, 40 оснований 5' конца гена сопротивления канамицина (Кан), промоутер pLac-Ara, место связывания рибосом (RBS), AUG гена mCherry, прокладка (я), место связывания RBP, 80 оснований 5' конца гена mCherry и места ограничения ApaLI.
- Клонирование связывающих площадок сайта
- Закажите связывающие кассеты в виде двухцепочечных минигенов ДНК (dsDNA). Каждый миниген имеет длину 500 б.п. и содержит место ограничения Eagl и место ограничения ApaLI на концах 5' и 3', соответственно (см. шаг 1.1.1).
ПРИМЕЧАНИЕ: В этом эксперименте, мини-гены с половиной гена канамицина было приказано облегчить скрининг для положительных колоний. Тем не менее, Гибсон сборки17 также подходит здесь, и в этом случае связывание сайт может быть заказан в качестве двух коротких дополнительных одноцепочечных олигоДНК ДНК. - Двойной переваривание как мини-генов и целевой вектор с Eagl-HF и ApaLI по протоколу ограничения18, и столбец очистить19.
- Ligate переваренных minigenes к связывающей-сайт позвоночника, содержащего остальную часть гена репортера mCherry, терминатор, и ген анамицин устойчивости20.
- Преобразуйте раствор перевязки в клетки Escherichia coli TOP1021.
- Определите положительные трансформанты с помощью секвенирования Sanger.
- Дизайн грунтовки 100 баз вверх по течению к региону интереса (см. таблицу 1 для грунтовых последовательностей).
- Miniprep несколько бактериальных колоний22.
- Приготовьте 5 мкм раствора грунтовки и 10 л ДНК при концентрации 80 нг/Л.
- Отправить два решения на удобный объект для Секне секвенирования23.
- Храните очищенные плазмиды при -20 градусах по Цельсию, а также бактериальные штаммы в виде глицерола24,как в 96-хорошем формате. ЗАТЕМ ДНК будет использоваться для преобразования в клетки E. coli TOP10, содержащие одну из четырех плазмид слияния-RBP (см. шаг 1.3.5).
- Закажите связывающие кассеты в виде двухцепочечных минигенов ДНК (dsDNA). Каждый миниген имеет длину 500 б.п. и содержит место ограничения Eagl и место ограничения ApaLI на концах 5' и 3', соответственно (см. шаг 1.1.1).
- Проектирование и строительство плазмида РБП
ПРИМЕЧАНИЕ: Аминокислоты и нуклеотидные последовательности белков пальто, используемых в этом исследовании, перечислены в таблице 2.- Заказать требуемую последовательность RBP не хватает остановки кодон как заказ dsDNA minigene не хватает остановки кодон с ограничения сайтов на концах (Рисунок 1).
- Клон испытания RBP не хватает остановки кодон сразу вниз по течению индуцируемых промоутер и вверх по течению флуоресцентного белка не хватает начала кодон (Рисунок 1), похож на шаги 1.2.2-1.2.4. Убедитесь, что плазмида RBP содержит другой ген устойчивости к антибиотикам, чем обязательная плазмида.
- Определите положительные трансформанты с помощью секвенирования Sanger, аналогичного шагу 1.2.5 (см. таблицу 1 для последовательностей грунтовки).
- Выберите один положительный трансформатор и сделать его химически компетентным25. Хранить в качестве глицерола очищенные плазмиды при -20 градусах по Цельсию и глицерол запасы бактериальных штаммов24 при -80 градусах По цельсию в 96-колодцах пластин.
- Преобразуйте пласмиды связывающего участка (от шага 1.2.6), хранящиеся в 96-колодцах, в химически компетентные бактериальные клетки, уже содержащие плазмид RBP-mCerulean21. Чтобы сэкономить время, вместо того, чтобы накрывать клетки на чашках Петри, наклеиваем их на 8-канальный пипетки на 8-полосные тарелки, содержащие Лурию-Бертани (LB)26 агар агар агар асоответствующих антибиотиков (Kan и Amp). Колонии должны появиться в 16 ч.
- Выберите одну колонию для каждого двойного трансформанта и расти на ночь в среде LB с соответствующими антибиотиками (Кан и Amp) и хранить как глицерол запасов24 при -80 градусов по Цельсию в 96-колодцепластины.
2. Эксперимент настройки
ПРИМЕЧАНИЕ: Представленный здесь протокол был выполнен с использованием робототехнической системы обработки жидкостей в сочетании с инкубатором и считывателем пластин. Каждое измерение проводилось для 24 концентраций индуктора, с двумя дубликатами для каждого штамма и индуктора комбинации. С помощью этой роботизированной системы были собраны данные по 16 штаммам в день с 24 концентрациями индуктора. Однако, если такое устройство недоступно, или если требуется меньше экспериментов, их можно легко сделать вручную с помощью 8-канальной мультипипетки и соответствующим образом адаптировать протокол. Например, таким образом были получены предварительные результаты по четырем штаммам в день с 12 концентрациями индуктора и четырьмя тайм-пойнтами.
- Приготовьте заранее 1 л биоасса-буфера (BA) путем смешивания 0,5 г триптона, 0,3 мл глицерола, 5,8 г NaCl, 50 мл 1 M MgSO4, 1 мл 10x фосфат-буферный солен (PBS) буфер рН 7,4 и 950 мл двойной дистиллированной воды (DD). Автоклав или стерильный фильтр буфер BA.
- Выращивайте двойные трансформанты при 37 градусах Цельсия и 250 об/мин, встряхивая в 1,5 мл ЛТ с соответствующими антибиотиками (канамицин при конечной концентрации 25 мкг/мл и ампициллин при конечной концентрации 100 мкг/мл), в 48-ну колодцев, в течение 18 л.с.
-
Утром сделайте следующие приготовления.
- Индуктора пластины. В чистой 96-хорошо пластины, подготовить скважины с полубедной среде (SPM), состоящей из 95% BA и 5% LB26 в инкубаторе при 37 градусов по Цельсию. Количество скважин соответствует желаемому количеству концентраций индуктора. Добавьте C4-HSL к скважинам в пластине индуктора, которая будет содержать самую высокую концентрацию индуктора (218 нм).
- Программа робота, чтобы последовательно разбавить среду от каждой из скважин с высокой концентрацией в 23 нижних концентраций, начиная от 0 до 218 нм. Объем разбавления каждого индуктора должен быть достаточным для всех штаммов (включая дубликаты).
- В то время как разбавления индуктора готовятся, тепло 180 л SPM в инкубаторе при 37 градусах Цельсия, в 96-колодцах пластин.
- Разбавить ночные штаммы со ступени 2,2 в 100 раз серийными разбавлениями: сначала разбавить в 10 раз, смешивая 100 л бактерий с 900 л SPM в 48-ну хорошо пластины, а затем разбавить снова в 10 раз, взяв 20 qL от разбавленного раствора в 180 л предварительно разогретого SPM, в 96-хорошо пластин, пригодных для флуоресцентных измерений.
- Добавьте разбавленный индуктор из индуктора в 96-колодцы пластины с разбавленными штаммами в соответствии с окончательными концентрациями.
- Встряхните 96-колодственные пластины при температуре 37 градусов по Цельсию на 6 ч, при измерении оптической плотности на уровне 595 нм (OD595),mCherry (560 нм/612 нм) и mCerulean (460 нм/510 нм) флуоресценции через плиту читателя каждые 30 минут. Для целей нормализации измеряйте рост СМП без добавления ячеек.
3. Предварительный анализ результатов
- Для каждого дня эксперимента выбирайте временной интервал логарифмического роста в соответствии с измеренными кривыми роста, между фазой линейного роста и стационарной (T0, Tfinal). Возьмите примерно 6-8 временных точек, отбрасывая первые и последние измерения, чтобы избежать погрешности, полученной из неточности экспоненциального обнаружения роста (см. рисунок 2A, верхняя панель).
ПРИМЕЧАНИЕ: Откажитесь от штаммов, которые показывают аномальные кривые роста или штаммы, где логарифмическая фаза роста не может быть обнаружена, и повторите эксперимент. -
Рассчитайте среднюю нормализованную флуоресценцию мцерулеана и скорость производства mCherry, из необработанных данных как mCerulean, так и mCherry флуоресценции для каждой концентрации индуктора(рисунок 2A).
- Рассчитайте нормализованный mCerulean следующим образом:
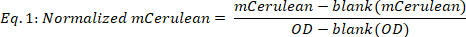
где пустой (mCerulean) является mCerulean уровнем (a.u.) для среднего только, пустой (OD) является оптическая плотность только для среднего только, и mCerulean и OD являются mCerulean флуоресценции и оптической плотности значения, соответственно. - Средний mCerulean над различными точками времени(рисунок 2B, верхние 2 панели) следующим образом:
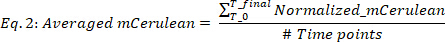
где #Time точками является количество данных тайм-точек, принятых во внимание, T0 это время, в котором начинается экспоненциальная фаза роста, и Tfinal это время, в котором экспоненциальная фаза роста заканчивается. - Рассчитайте скорость производства mCherry(рисунок 2B,две нижние панели) следующим образом:
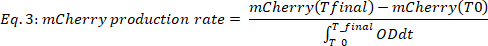
где mCherry(t) является уровнем mCherry в то время т, OD является значением оптической плотности, T0 это время, в котором начинается экспоненциальная фаза роста, а Tfinal – это время, в которое заканчивается экспоненциальная фаза роста.
- Рассчитайте нормализованный mCerulean следующим образом:
- Наконец, участок mCherry скорость производства в качестве функции mCerulean, создавая кривые реакции дозы в качестве функции RBP-mCerulean фьюжн(Рисунок 2C). Такие участки представляют собой производство гена репортера как функцию присутствия RBP в клетке.
4. Функция реагирования дозы Fitting Routine и KRBP Extraction
- Исходя из предположения, что рибосомная скорость перевода с привязанной РБП постоянна, моделируйте скорость производства mCherry следующим образом (см. рисунок 2D,зеленая линия):

где «x» является нормализованной средней мцерулеановой флуоресценции, рассчитанной в соответствии с Eq. 2, скорость производства mCherry является значением, рассчитанным в соответствии с Eq. 3, KRBP является относительной связывающей сродством ,a.u.), Kunbound является рибосомной скоростью перевод с RBP несвязанным, n является фактором куперативности, а C является базовой флуоресценцией .u. C, n, Kunboundи KRBP можно найти, приспосабливая данные о скорости производства mCherry к модели (Eq. 4). - С помощью программного обеспечения для анализа данных, провести установку процедуры на участках, изображающих скорость производства mCherry в качестве функции усредненного mCerulean (шаг 3.3), и извлечь соответствующие параметры в соответствии с формулой в Eq. 4.
ПРИМЕЧАНИЕ: Учитываются только соответствующие результаты с R2 и 0,6. Для тех, кто подходит, KRBP ошибка в основном в диапазоне от 0,5% до 20% от KRBP значения, для 0,67 доверительный интервал, в то время как те, с более высокой kRBP ошибка может быть также проверена глазом. - Нормализовать значения KRBP по соответствующей максимальной стоимости усреднения mCerulean для каждой функции реакции дозы.
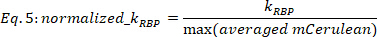
где KRBP в «a.u.» — это значение, извлеченное из процедуры установки в Eq. 4, а макс (средний mCerulean) — это максимальный усредненный mCerulean сигнал «a.u», наблюдаемый для текущего штамма.
ПРИМЕЧАНИЕ: Нормализация облегчает правильное сравнение регулирующего эффекта между штаммами, устраняя зависимость от конкретных максимальных уровней выражения RBP.
Результаты
Представленный метод использует конкуренцию между RBP и ribosome для связывать к молекуле мРНК (рисунок1). Эта конкуренция находит свое отражение в снижении уровня mCherry как функции увеличения производства RBP-mCerulean, в связи с увеличением концентрации индуктор...
Обсуждение
Метод, описанный в этой статье, облегчает количественное измерение связывания СВЯЗывающей РБП-РНК в клетках кишечной палочки. Протокол относительно прост и может быть проведен без использования сложного оборудования, а анализ данных прост. Кроме того, результаты получаются сразу...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Этот проект получил финансирование от Программы I-CORE Комитета по планированию и бюджетированию и Израильского научного фонда (Грант No 152/11), Гранта Марии Кюри по вопросам реинтеграции No. PCIG11-GA- 2012-321675, а также из программы исследований и инноваций Европейского союза Horizon 2020 по грантовому соглашению No 664918 - MRG-Grammar.
Материалы
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8-lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| Glycerol | BIO LAB | 071205 | |
| Incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| Ligase | NEB | B0202S | |
| Liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| Multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| Platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| Sodium Chloride (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
Ссылки
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены