このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
細菌におけるタンパク質-RNA結合を定量するためのアッセイ
要約
この方法では、細菌細胞における簡潔な生きたレポーターアッセイを用いて、RNA結合タンパク質(RBP)の結合親和性を、簡潔で生きたレポーターアッセイを用いて認知および非認知結合部位に定量する。アッセイはレポーター遺伝子の抑圧に基づいています。
要約
タンパク質翻訳の開始ステップでは、リボソームはmRNAの開始領域に結合する。翻訳開始は、リボソーム結合を妨げるmRNAの開始領域へのRNA結合タンパク質(RBP)の結合によって遮断されうる。提示された方法では、このブロッキング現象を利用して、その認知部位および非認知結合部位に対するRBPの結合親和性を定量化する。これを行うには、レポーターmRNAの開始領域に試験結合部位を挿入し、試験RBPの発現を誘導する。RBP-RNA結合の場合、RBP濃度の関数としてレポーター発現のシグモイド式の抑圧を観察した。結合部位とRBPとの間に親和性がないか、または非常に低い親和性の場合、有意な抑圧は認められなかった。この方法は、生きた細菌細胞で行われ、高価な機械や洗練された機械を必要としません。これは、細菌で機能する異なるBRBの結合親和性を、設計された結合部位のセットと定量化し、比較するのに有用である。この方法は、構造の複雑度が高い部位をバインドする場合には不適切な場合があります。これは、RBPが存在しない場合に複雑なmRNA構造によるリボソーム開始の抑圧の可能性に起因し、これは基底レポーター遺伝子発現を低下させ、したがってRBP結合時に観察可能なレポーターの抑圧をもたらす。
概要
RNA結合タンパク質(RBP)ベースの転写後調節、特にRBPとRNAとの相互作用の特徴付けは、ここ数十年で広範囲に研究されている。RbP阻害に起因する細菌における翻訳下調節の複数の例があり、または直接競合する、リボソーム結合1、2、3。合成生物学の分野では、RBP-RNA相互作用は転写ベースの遺伝回路4、5の設計のための重要なツールとして出現している。従って、このようなRBP-RNA相互作用の特性化に対する需要が細胞コンテキスト内で増加している。
タンパク質-RNA相互作用を研究するための最も一般的な方法は、インビトロ設定に限定される電気泳動シフトアッセイ(EMSA)6、およびCLIP法8、9を含む様々なプルダウンアッセイ7である。.このような方法は、デノボRNA結合部位の発見を可能にする一方で、労働集約的なプロトコルや高価な深いシーケンシング反応などの欠点に苦しんでおり、RBPプルダウンに対する特定の抗体を必要とする場合があります。RNAがその環境に対して感受性を持つため、多くの因子がRBP-RNA相互作用に影響を与える可能性があり、細胞コンテキストにおけるRBP-RNA結合を調知することの重要性を強調する。例えば、我々と他の人は、インビボとインビトロ10、11におけるRNA構造の間に有意な差を示した。
以前の研究12のアプローチに基づいて、我々は最近、バクテリオファージGA 13、MS214、PP715、およびQβ16からカプシドRBP用に事前に設計された結合部位を配置する際に10を実証した。レポーターmRNAの翻訳開始領域は、レポーター表現が強く抑圧されている。この抑圧現象に基づいて比較的単純で定量的な方法を提示し、生体内のRBPと対応するRNA結合部位との間の親和性を測定する。
プロトコル
1. システムの準備
- 結合部位プラスミドの設計
- 図 1に示すように、バインディング サイト カセットを設計します。各ミニ遺伝子は、以下の部分(5'〜3'):Eagl制限部位、カナマイシン(Kan)耐性遺伝子の5'末端の40塩基、pLac-Araプロモーター、リボソーム結合部位(RBS)、mCherry遺伝子のAUG、スペーサー(δ)、RBP結合部位、5'末端の80塩基を含むmCherry遺伝子、およびApaLI制限部位の。
注:アッセイの成功率を高めるために、各結合部位に対して3つの結合部位カセットを設計し、スペーサーは少なくとも1つ、2つ、および3つのベースで構成される。その他のガイドラインについては、「代表的な結果」セクションを参照してください。
- 図 1に示すように、バインディング サイト カセットを設計します。各ミニ遺伝子は、以下の部分(5'〜3'):Eagl制限部位、カナマイシン(Kan)耐性遺伝子の5'末端の40塩基、pLac-Araプロモーター、リボソーム結合部位(RBS)、mCherry遺伝子のAUG、スペーサー(δ)、RBP結合部位、5'末端の80塩基を含むmCherry遺伝子、およびApaLI制限部位の。
- 結合部位プラスミドのクローニング
- 結合部位カセットを二本鎖DNA(dsDNA)ミニ遺伝子として注文します。各ミニ遺伝子は500bpの長さで、それぞれ5'と3'の端にEagl制限部位とApaLI制限部位が含まれています(ステップ1.1.1を参照)。
注:本実験では、カナマイシン遺伝子の半分を含むミニ遺伝子を、陽性コロニーのスクリーニングを容易にするよう命じた。しかしながら、ギブソンアセンブリ17もまた、ここで適しており、その場合、結合部位は2つのより短い相補的な一本鎖DNAオリゴとして発注することができる。 - 制限プロトコル18によりEagl-HFおよびApaLIを用いてミニ遺伝子と標的ベクターの両方を二重消化し、カラム精製19.
- 消化されたミニ遺伝子を、残りのmCherryレポーター遺伝子、ターミネーター、およびカナマイシン耐性遺伝子20を含む結合部位バックボーンにライゲートする。
- ライゲーション溶液を大腸菌TOP10細胞21に変換する。
- サンガーシーケンスを使用して正の形質転換体を識別します。
- 対象地域に上流のプライマー 100 基を設計します (プライマー シーケンスについては、表 1を参照してください)。
- ミニプレップいくつかの細菌のコロニー22.
- プライマーの5 mM溶液の5 μLと80 ng/μL濃度でDNAの10 μLを調出します。
- 2 つのソリューションをサンガー シーケンシング23の便利な施設に送信します。
- 精製プラスミドを-20°Cで貯蔵し、グリセロールストック24として細菌株を、いずれも96ウェル形式で保存する。DNAは、4つの融合RBPプラスミドのいずれかを含む大腸菌TOP10細胞への形質転換に使用されます(ステップ1.3.5参照)。
- 結合部位カセットを二本鎖DNA(dsDNA)ミニ遺伝子として注文します。各ミニ遺伝子は500bpの長さで、それぞれ5'と3'の端にEagl制限部位とApaLI制限部位が含まれています(ステップ1.1.1を参照)。
- RBPプラスミドの設計と建設
注:本研究で用いられるコートタンパク質のアミノ酸およびヌクレオチド配列は表2に記載されている。- 制限部位を持つストップコドンを欠くカスタムオーダーdsDNAミニジーンとしてストップコドンを欠いている必要なRBP配列を注文します(図1)。
- 試験済みのRBPを、誘導性プロモーターの直下流に停止コドンを欠き、開始コドンを欠く蛍光タンパク質の上流(図1)を、ステップ1.2.2-1.2.4と同様にクローンする。RBPプラスミドに結合部位プラスミドとは異なる抗生物質耐性遺伝子が含まれていることを確認してください。
- ステップ 1.2.5 と同様に、Sanger シーケンスを使用して正の変換体を識別します (プライマー シーケンスについては表 1を参照)。
- 1つの正の形質転換剤を選択し、それを化学的に有能な25にする。グリセロール精製プラスミドを-20°C、96ウェルプレートで-80°Cで-80°Cの細菌株24のグリセロールストックとして保存する。
- 96ウェルプレートに保存された結合部位プラスミド(ステップ1.2.6から)を、RBP-mCeruleanプラスミド21を既に含む化学的に有能な細菌細胞に変換する。時間を節約するために、ペトリ皿に細胞をめっきする代わりに、ルリア・ベルタニ(LB)26寒天を含む8レーンプレートに8チャンネルのピペッターを使用して、関連する抗生物質(カンとアンプ)を使用してプレートを付けます。コロニーは16時間で現れるはずです。
- 各二重形質転換剤に対して単一のコロニーを選択し、関連する抗生物質(カンおよびアンプ)とLB培地で一晩成長し、96ウェルプレートで-80°Cでグリセロールストック24として保存します。
2. 実験設定
注:ここで提示されるプロトコルは、インキュベーターおよびプレートリーダーと組み合わせて液体処理ロボットシステムを用いて行った。各測定は、24のインデューサー濃度について行い、各株+インデューサーの組み合わせに対して2つの複製を用いて行った。このロボットシステムを用いて、24のインデューサー濃度を有する1日あたり16株のデータを収集した。しかし、このようなデバイスが利用できない場合、または必要な実験が少ない場合は、8チャンネルのマルチピペットを使用して、それに応じてプロトコルを適応させることが容易に行えます。例えば、12インデューサー濃度と4つのタイムポイントを有する1日あたり4株の予備結果は、このように取得された。
- 調製は、予め、0.5gのトリプトン、0.3mLのグリセロール、5.8gのナクル、1M MgSO4の50mL、10倍リン酸緩衝生理食生(PBS)バッファーpH7.4、および950mLの二重蒸し水(DL)を混合して、1Lのバイオアッセイバッファー(BA)を調製する。オートクレーブまたは無菌フィルタBAバッファ。
- 適切な抗生物質(25μg/mLの最終濃度でカナマイシン、最終濃度100μg/mLでアンピシリン)を1.5mL LBで振る37°Cおよび250rpmで二重形質菌株を48ウェルプレートで、18時間(一晩)に成長させます。
-
午前中は、以下の準備をしてください。
- インデューサープレート。きれいな96ウェルプレートで、37°Cのインキュベーターで95%BAおよび5%LB26からなる半不良培地(SPM)で井戸を準備する。ウェルの数は、所望のインデューサー濃度の数に対応する。最高インデューサ濃度(218 nM)を含むインデューサープレートのウェルにC4-HSLを追加します。
- ロボットをプログラムして、最も高濃度の井戸のそれぞれから0~218 nMまでの23の低濃度に連続的に媒体を希釈します。各インデューサー希釈の体積は、すべての株(重複を含む)に対して十分である必要があります。
- インデューサ希釈剤が調製されている間、96ウェルプレートで37°CのインキュベーターでSPMの180 μLを温める。
- ステップ2.2から一晩の菌株を連続希釈で100倍に希釈:まず、100μLの細菌と48ウェルプレートの900μLのSPMを混合して10倍に希釈し、希釈液から20μLを取り込んで再び10倍に希釈する。蛍光測定に適した96ウェルプレートで、予め温められたSPMの180 μL。
- 最終濃度に応じて希釈株を用いて96ウェルプレートに誘導板から希釈インデューサを加加える。
- 595nm(OD 595)、mCherry(560 nm/612 nm)、mCerulean(460 nm/510 nm)の蛍光を30分毎にプレートリーダーで測定しながら、37°Cで96ウェルプレートを6時間振ります。正規化の目的で、細胞を追加しない SMP の増殖を測定します。
3. 予備結果分析
- 実験の各日について、測定された成長曲線に従って対数成長の時間間隔を選択し、線形成長相と静止期(T0,T最終)の間にする。指数成長検出の不正確さから生じた誤差を避けるために、最初と最後の測定値を破棄しながら、約 6-8 の時点を取ります (図 2A、トップパネルを参照)。
注:対数成長相が検出できなかった異常な成長曲線または株を示す株を破棄し、実験を繰り返します。 -
mCeruleanの平均正規化蛍光およびmCherryの産生速度を、各インデューサー濃度に対するmCeruleanおよびmCherry蛍光の両方の生データから算出する(図2A)。
- 正規化された mCerulean を次のように計算します。
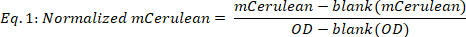
ブランク(mCerulean)は中程度のみのmCeruleanレベル[a.u.]で、ブランク(OD)は中程度のみの光学密度であり、mCeruleanおよびODはそれぞれmCerulean蛍光および光学密度値である。 - 異なるタイムポイント(図2B、上2パネル)の平均mCeruleanは次のとおりです。
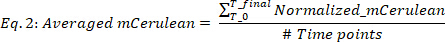
#Timeポイントは考慮されるデータタイムポイントの数であり、T0は指数成長フェーズが開始される時刻であり、T最終値は指数成長フェーズが終了する時刻です。 - 生産のmCherry率(図2B、下の2つのパネル)を次のように計算します。
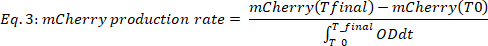
ここで mCherry(t) は時間 t の mCherry レベル [a.u.] で、OD は光学密度値、T0は指数成長フェーズが始まる時刻であり、Tファイナルは指数成長フェーズが終了する時刻です。
- 正規化された mCerulean を次のように計算します。
- 最後に、mCeruleanの関数として生産のmCherry率をプロットし、RBP-mCerulean融合蛍光の関数として用量応答曲線を作成する(図2C)。このようなプロットは、細胞内のRBP存在の関数としてのレポーター遺伝子の産生を表す。
4. 線量応答機能フィッティングルーチンとKRBP抽出
- RBP バインドを持つリボソーム変換率が一定であることを前提として、mCherry の生産率を次のようにモデル化します (図 2D、緑色の線を参照)。

ここで[x]は、Eq.2に従って計算された正規化平均mCerulean蛍光であり、mCherry産生率はEq.3に従って計算された値であり、KRBPは相対結合親和性[a.u.]であり、Kアンバウンドはリボソーム率である。RBP非結合での変換、nは協調性因子であり、Cはベース蛍光[a.u.]である。C、n、K非連結、および KRBPは、mCherry 生産率データをモデルに適合することによって見つかります (Eq. 4)。 - データ解析ソフトウェアを用いて、平均mCerulean(ステップ3.3)の関数としてmCherry生産率を描写するプロットにフィッティング手順を行い、Eq.4の式に従って適合パラメータを抽出する。
注:R2 > 0.6 の適合結果のみが考慮されます。これらの適合については、KRBP誤差は主にKRBP値の0.5%~20%の範囲にあり、0.67信頼区間では、KRBP誤差が高い場合も目で確認できます。 - 各用量応答関数の平均mCeruleanのそれぞれの最大値によってKRBP値を正規化する。
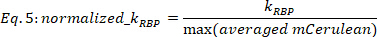
ここで[a.u.]のKRBPはEq.4のフィッティング手順から抽出された値であり、最大値(平均mCerulean)は現在の株に対して観察される最大平均mCeruleanシグナル[a.u]です。
注:正規化は、特定の最大RBP発現レベルへの依存性を排除することにより、株間の調節効果の正しい比較を容易にする。
結果
提示された方法は、mRNA分子に結合するためのRBPとリボソームとの間の競争を利用する(図1)。この競争は、インデューサーの濃度の増加に起因するRBP-mCeruleanの生産増加の関数としてmCherryレベルを減少させることによって反映されます。mCerulean蛍光を増加させる場合、mCherryに有意な変化はなく、RBP結合の欠如が推測される。正と負のひずみの両方...
ディスカッション
本稿に記載の方法は、大腸菌細胞におけるRBP-RNA結合親和性の生体内測定を容易にする。プロトコルは比較的簡単で、高度な機械を使用せずに行うことができ、データ分析は簡単です。さらに、結果は、次世代シーケンシング(NGS)結果に関連する比較的長い待ち時間なしで、すぐに生成されます。
この方法の 1 つの制限は、細菌細胞でのみ動作することです。.しか?...
開示事項
著者は何も開示していない。
謝辞
このプロジェクトは、計画予算委員会のI-COREプログラムとイスラエル科学財団(助成金第152/11)、マリー・キュリー再統合助成金第1号から資金を受け取りました。PCIG11-GA- 2012-321675、および補助金契約第664918号の下で欧州連合のホライズン2020研究革新プログラムから - MRG-文法。
資料
| Name | Company | Catalog Number | Comments |
| Ampicillin sodium salt | SIGMA | A9518 | |
| Magnesium sulfate (MgSO4) | ALFA AESAR | 33337 | |
| 48 plates | Axygen | P-5ML-48-C-S | |
| 8- lane plates | Axygen | RESMW8I | |
| 96-well plates | Axygen | P-DW-20-C | |
| 96-well plates for plate reader | Perkin Elmer | 6005029 | |
| ApaLI | NEB | R0507 | |
| Binding site sequences | Gen9 Inc. and Twist Bioscience | see Table 1 | |
| E. coli TOP10 cells | Invitrogen | C404006 | |
| Eagl-HF | NEB | R3505 | |
| glycerol | BIO LAB | 071205 | |
| incubator | TECAN | liconic incubator | |
| Kanamycin solfate | SIGMA | K4000 | |
| KpnI- HF | NEB | R0142 | |
| ligase | NEB | B0202S | |
| liquid-handling robotic system | TECAN | EVO 100, MCA 96-channel | |
| Matlab analysis software | Mathworks | ||
| multi- pipette 8 lanes | Axygen | BR703710 | |
| N-butanoyl-L-homoserine lactone (C4-HSL) | cayman | K40982552 019 | |
| PBS buffer | Biological Industries | 020235A | |
| platereader | TECAN | Infinite F200 PRO | |
| Q5 HotStart Polymerase | NEB | M0493 | |
| RBP seqeunces | Addgene | 27121 & 40650 | see Table 2 |
| SODIUM CHLORIDE (NaCL) | BIO LAB | 190305 | |
| SV Gel and PCR Clean-Up System | Promega | A9281 | |
| Tryptone | BD | 211705 |
参考文献
- Cerretti, D. P., Mattheakis, L. C., Kearney, K. R., Vu, L., Nomura, M. Translational regulation of the spc operon in Escherichia coli. Identification and structural analysis of the target site for S8 repressor protein. Journal of Molecular Biology. 204 (2), 309-329 (1988).
- Babitzke, P., Baker, C. S., Romeo, T. Regulation of translation initiation by RNA binding proteins. Annual Review of Microbiology. 63, 27-44 (2009).
- Van Assche, E., Van Puyvelde, S., Vanderleyden, J., Steenackers, H. P. RNA-binding proteins involved in post-transcriptional regulation in bacteria. Frontiers in Microbiology. 6, 141 (2015).
- Chappell, J., Watters, K. E., Takahashi, M. K., Lucks, J. B. A renaissance in RNA synthetic biology: new mechanisms, applications and tools for the future. Current Opinion in Chemical Biology. 28, 47-56 (2015).
- Wagner, T. E., et al. Small-molecule-based regulation of RNA-delivered circuits in mammalian cells. Nature Chemical Biology. 14 (11), 1043 (2018).
- Bendak, K., et al. A rapid method for assessing the RNA-binding potential of a protein. Nucleic Acids Research. 40 (14), e105 (2012).
- Strein, C., Alleaume, A. -. M., Rothbauer, U., Hentze, M. W., Castello, A. A versatile assay for RNA-binding proteins in living cells. RNA. 20 (5), 721-731 (2014).
- Ule, J., Jensen, K. B., Ruggiu, M., Mele, A., Ule, A., Darnell, R. B. CLIP identifies Nova-regulated RNA networks in the brain. Science. 302 (5648), 1212-1215 (2003).
- Lee, F. C. Y., Ule, J. Advances in CLIP Technologies for Studies of Protein-RNA Interactions. Molecular Cell. 69 (3), 354-369 (2018).
- Katz, N., et al. An in Vivo Binding Assay for RNA-Binding Proteins Based on Repression of a Reporter Gene. ACS Synthetic Biology. 7 (12), 2765-2774 (2018).
- Watters, K. E., Yu, A. M., Strobel, E. J., Settle, A. H., Lucks, J. B. Characterizing RNA structures in vitro and in vivo with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Methods. 103, 34-48 (2016).
- Saito, H., et al. Synthetic translational regulation by an L7Ae-kink-turn RNP switch. Nature Chemical Biology. 6 (1), 71-78 (2010).
- Gott, J. M., Wilhelm, L. J., Uhlenbeck, O. C. RNA binding properties of the coat protein from bacteriophage GA. Nucleic Acids Research. 19 (23), 6499-6503 (1991).
- Peabody, D. S. The RNA binding site of bacteriophage MS2 coat protein. The EMBO Journal. 12 (2), 595-600 (1993).
- Lim, F., Peabody, D. S. RNA recognition site of PP7 coat protein. Nucleic Acids Research. 30 (19), 4138-4144 (2002).
- Lim, F., Spingola, M., Peabody, D. S. The RNA-binding Site of Bacteriophage Qβ Coat Protein. Journal of Biological Chemistry. 271 (50), 31839-31845 (1996).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Optimizing Restriction Endonuclease Reactions Available from: https://international.neb.com/tools-and-resources/usage-guidelines/optimizing-restriction-endonuclease-reactions (2018)
- . Wizard® SV Gel and PCR Clean-Up System Protocol Available from: https://worldwide.promega.com/resources/protocols/technical-bulletins/101/wizard-sv-gel-and-pcr-cleanup-system-protocol/ (2018)
- . Ligation Protocol with T4 DNA Ligase (M0202) Available from: https://international.neb.com/protocols/0001/01/01/dna-ligation-with-t4-dna-ligase-m0202 (2018)
- . Routine Cloning Using Top10 Competent Cells - US Available from: https://www.thermofisher.com/us/en/home/references/protocols/cloning/competent-cells-protocol/routine-cloning-using-top10-competent-cells.html (2018)
- . NucleoSpin Plasmid - plasmid Miniprep kit Available from: https://www.mn-net.com/ProductsBioanalysis/DNAandRNApurification/PlasmidDNApurificationeasyfastreliable/NucleoSpinPlasmidplasmidMiniprepkit/tabid/1379/language/en-US/Default.aspx (2018)
- Sanger, F., Coulson, A. R., Barrell, B. G., Smith, A. J. H., Roe, B. A. Cloning in single-stranded bacteriophage as an aid to rapid DNA sequencing. Journal of Molecular Biology. 143 (2), 161-178 (1980).
- . Protocol - How to Create a Bacterial Glycerol Stock Available from: https://www.addgene.org/protocols/create-glycerol-stock/ (2018)
- . Making your own chemically competent cells Available from: https://international.neb.com/protocols/2012/06/21/making-your-own-chemically-competent-cells (2018)
- . Luria-Bertani (LB) Medium Preparation · Benchling Available from: https://benchling.com/protocols/gdD7XI0J/luria-bertani-lb-medium-preparation (2018)
- Delebecque, C. J., Silver, P. A., Lindner, A. B. Designing and using RNA scaffolds to assemble proteins in vivo. Nature Protocols. 7 (10), 1797-1807 (2012).
- Hocine, S., Raymond, P., Zenklusen, D., Chao, J. A., Singer, R. H. Single-molecule analysis of gene expression using two-color RNA labeling in live yeast. Nature Methods. 10 (2), 119-121 (2013).
- Espah Borujeni, A., et al. Precise quantification of translation inhibition by mRNA structures that overlap with the ribosomal footprint in N-terminal coding sequences. Nucleic Acids Research. 45 (9), 5437-5448 (2017).
- Ding, Y., et al. In vivo genome-wide profiling of RNA secondary structure reveals novel regulatory features. Nature. 505, (2013).
- Rouskin, S., Zubradt, M., Washietl, S., Kellis, M., Weissman, J. S. Genome-wide probing of RNA structure reveals active unfolding of mRNA structures in vivo. Nature. 505 (7485), 701-705 (2014).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2’-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proceedings of the National Academy of Sciences of the United States of America. 108 (27), 11063-11068 (2011).
- Spitale, R. C., et al. Structural imprints in vivo decode RNA regulatory mechanisms. Nature. 519 (7544), 486 (2015).
- Watters, K. E., Abbott, T. R., Lucks, J. B. Simultaneous characterization of cellular RNA structure and function with in-cell SHAPE-Seq. Nucleic Acids Research. 44 (2), e12 (2016).
- Flynn, R. A., et al. Transcriptome-wide interrogation of RNA secondary structure in living cells with icSHAPE. Nature Protocols. 11 (2), 273-290 (2016).
- Bernardi, A., Spahr, P. -. F. Nucleotide Sequence at the Binding Site for Coat Protein on RNA of Bacteriophage R17. Proceedings of the National Academy of Sciences of the United States of America. 69 (10), 3033-3037 (1972).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved