A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
قياس ديناميات الأنابيب المجهرية بواسطة المجهر القرص الغزل في المغزل ميتوتيك القطب
In This Article
Summary
نقدم هنا طريقه قويه ومفصله لتحليل ديناميات البولية المجهرية في الخلايا المتزامنة في طليعة باستخدام الخلية الحية الغزل القرص المجهري المجهر ومعالجه الصور المستندة إلى matlab.
Abstract
ونحن نقدم وصفا لتعديل طريقه محدده لتحديد ديناميات البولية الصغيرة في الخلايا الحية. ويستند البروتوكول علي التعبير عن علامة المشفرة وراثيا لغايات ايجابيه من الأنابيب الدقيقة (EB3 الموسومة مع بروتين الفلورسنت tdTomato) وعاليه السرعة, عاليه الدقة, التصوير الخلوي الحي باستخدام القرص الدوار المجهر المجهري. يتم تحقيق تزامن دوره الخلية وزيادة كثافة الأنابيب الدقيقة من خلال تثبيط الانفصال الوسطي في الخلايا المتصوقيه ، ويتم اجراء تحليل النمو باستخدام برنامج المسار المفتوح المصدر. ان استخدام بروتين فلوري مشرق واحمر اللون ، بالاشتراك مع طاقة الليزر المنخفضة ووقت التعرض المنخفض المطلوب لدوران القرص المجهري يقلل من السمية الضوئية واحتمال القطع الاثريه المستحثة ضوئيا. وهذا يسمح بتصوير عدد أكبر من الخلايا في نفس الاعداد مع الحفاظ علي الخلايا في وسط النمو في ظل ظروف الثقافة القياسية. ونظرا لان التحليل يتم بطريقه تلقائية خاضعه للاشراف ، فان النتائج قويه إحصائيا وقابله للتكرار.
Introduction
الأنابيب المجهرية (MTs) هي هياكل ديناميكية للغاية وجدت في جميع الخلايا الحقيقية النواة تقريبا وفي بعض البكتيريا1. مع [اكتين] وخيوط متوسطه, ينحت هم [ستوسيكلتون]2,3. تقسيم الخلية4، جزيء النقل5، الضرب الصواري6، والإحساس من البيئة المحيطة من خلال الابتدائية هدب7، السمع (kinocilium)8،9، الاجنه10،11،12، الغزو وورم خبيث13،14، وحتى تكوين الذاكرة15،16،17،18، والعديد من العمليات الأخرى تعتمد أساسا علي MTs. سيكون من المستحيل مشاركه الmts في جميع هذه الاحداث دون قدرتها الملحوظة علي التحول بسرعة بين النمو (البلمره) والانكماش (التحلل). وصفت هذه الخاصية بأنها عدم الاستقرار الديناميكي19. يتم تغيير MT ديناميستي في العديد من الحالات المرضية20،21،22. التالي ، فان تحديد طبيعة هذه الخاصية يمكن ان يساعد علي فهم أليات المرض وبعد ذلك معالجتها.
وقد وضعت قائمه طويلة من الأساليب لتحليل ديناميات MT, معظمها تستند إلى تقنيات التصوير23. في البداية ، تم استخدام المجاهر الضوئية واسعه المجال لمراقبه تشكيل أنابيب البوليمرات في المختبر24. الاكتشاف من [ند-كندينغ] ([ب])-بروتينات ان يجمع في [مت] [بلس-نهاي] والتطوير الطرق إلى فلوريسسينتلي تسميه بروتينات جعل هو يمكن ان يلاحظ التصرف ال [MTs] مباشره في [ليفينغ سل] مع مجال واسعه و [كونككلر] مجاهر مجهر25,26,27. واحده [آيب-بروتين] [ند-بيندينغ] بروتين 3 (EB3)28; من خلال التعبير المفرط وتتبع EB3 تنصهر إلى بروتين الفلورسنت ، يمكن تحديد معدلات التجميع MT plus-نهاية29،30.
وكثيرا ما يستخدم المجهر الضوئي الليزر البؤري المجهري (CLSM) لمتابعه ديناميات MT. ومع ذلك ، فان تقنيه التصوير هذه تشكل خطرا عاليا من السمية الضوئية والرشح الضوئي ، وهما عمليتان غير مرغوب فيهما للخلايا الحية وعينات التصوير الخافتة31. من أجل الحصول علي نسبه أفضل من الاشاره إلى الضوضاء ، يجب ان تكون قوه الليزر ومده التعرض عاليه بما يكفي في حين لا تضر العينات ، وهذا يتطلب التضحية بالقرار في مقابل السرعة. بديل مناسب ل CLSM هو الغزل القرص المجهر32. ويستند هذا الأسلوب التصوير علي استخدام القرص Nipkow33، والذي يتكون من قرص متحرك تحمل مجموعه من الثقوب ، ويعمل اي ما يعادل إلى العديد من المجاهر CLS التصوير نفس العينة في وقت واحد34. ولذلك ، فان الضوء من الليزر سوف تضيء عده مناطق في العينة في وقت واحد ولكن الحفاظ علي الطبيعة المحورية. التالي ، فان قرص Nipkow يسمح بالحصول علي صور مشابهه ل CLSM ولكن بشكل أسرع وباستخدام طاقة ليزر اقل. تم تحسين القرص Nipkow كذلك من قبل Yokogawa الكهربائية, التي أدخلت القرص الثاني مع مجموعه من العدسات الدقيقة علي ذلك ان الضوء المباشر بشكل فردي في ثقب الدبوس المعنية, مزيد من الحد من السمية الضوئية والتصوير الضوئي35. وهكذا, الغزل القرص الليزر المسح المجهري أصبحت طريقه الاختيار للتصوير الخلية الحية, ويجعل من الممكن الحصول علي الصور مع ارتفاع نسبه الاشاره إلى الضوضاء في سرعه عاليه31,36, وهو أمر حاسم لحل الإشارات مثل تلك من نهايات MT سريعة النمو.
ديناميات MT تختلف مؤقتا. علي سبيل المثال ، ميتيك MTs هي أكثر ديناميكية من تلك المرحلة البينية37،38. المثل ، لوحظت الاختلافات في معدل النمو والانكماش حتى داخل نفس مرحله دوره الخلية ، مثل انقسام39،40. لذلك ، لتجنب جمع البيانات الزائفة ، يجب ان يقتصر قياس ديناميات MT علي اطار زمني ضيق اثناء دوره الخلية. علي سبيل المثال ، يمكن تحقيق قياس ديناميات MT في طليعة عن طريق علاج الخلايا مع dimethylenastron (DME) ، وهو التناظرية monastrol الذي يمنع المحرك كينيسن Eg541 ويمنع تشكيل المغزل القطبين القطبية42. تثبيط الخلايا في طليعة مع مثبطات Eg5 DME وغيرها من المشتقات monastrol لا يؤثر علي mt ديناميات43،44،45، مما يجعل من DME أداه مفيده لدراسة ديناميات mt علي حد سواء في الخلايا الثابتة والحية44.
هنا نحن الجمع بين طريقه تحليل ديناميات MT في الخلايا بروتافاز الموصوفة من قبل Ertych وآخرون44 مع التصوير المزدوج القرص الغزل. يسمح هذا الأسلوب قياس ديناميات MT في الخلايا prometase التي تم جمعها من المستوي البؤري واحد مع معدل التصوير اعلي ، ولكن دون الرشح الضوئي والحد الأدنى من السمية الضوئية. وعلاوة علي ذلك ، وكمراسل الفلورسنت ، ونحن نستخدم البروتينات الفلورية ديمر الطماطم (tdtomato) التي تحسنت السطوع والاستقرار الضوئي بالمقارنة مع البروتين الفلوري الأخضر (egfp) ومتحمس مع انخفاض الطاقة الخفيفة46. لذلك ، يتطلب tdTomato طاقة ليزر اقل للاثاره واقل سميه ضوئيه. الإجمال ، فاننا نقوم بتحسين الطريقة من خلال الحد من السمية الضوئية وتحسين الدقة والمعالجة اللاحقة المطلوبة لتحليل ديناميات MT. بالاضافه إلى ذلك ، نقوم بإنشاء أساس للتعديلات المستقبلية للأسلوب عن طريق الجمع بينها وبين تقنيات التزامن الأخرى.
Access restricted. Please log in or start a trial to view this content.
Protocol
1. بذر الخلايا هيلا
- اعداد 2 مل من 5 ميكروغرام/مل الفيبرونكتين جنيني الحل في الفوسفات مخزنه المالحة (التلفزيونية) وأضافه 450 μl منه في كل بئر من 4 بشكل جيد مكفرشفه (#1.5). احتضان الشريحة لمده 15 دقيقه في 37 درجه مئوية و 5 ٪ CO2.
- شطف خلايا هيلا المتنامية بشكل غير متزامن مع الفوسفات المخزن في دولبيكو المالحة (DPBS) واحتضان مع تريبسين-أدتا (0.05 ٪: 0.02 ٪ ؛ w:v) لمده 5 دقائق في 37 درجه مئوية. وقف رد الفعل الانزيمي باضافه معهد روزويل بارك التذكاري (RPMI) 1640 المتوسطة تستكمل مع 10% الحرارة المعطلة الجنين الساق المصل (FCS) في 3:1 (v:v) نسبه المضافة التريبسين-أدتا.
ملاحظه: تم الحفاظ علي الخلايا هيلا في RPMI 1640 المتوسطة تستكمل مع 10 ٪ FCS الحرارة المعطلة في 37 درجه مئوية و 5 ٪ CO2 وكانت تمر بشكل روتيني مره واحده وصلوا 80-90 ٪ كونفلوينسي كما هو موضح أعلاه. - تحديد تركيز الخلية باستخدام غرفه نيوبوير. اخلط 50 μL الخاصة بتعليق الخلية باللون الأزرق التريبين عند 1:1 (v:v) نسبه ، أعاده التعليق ، ونقل 10 μL من التعليق إلى الغرفة. احسب فقط الخلايا السالبة الزرقاء الموجودة داخل المربعات الاربعه الكبيرة (للاطلاع علي التفاصيل ، انظر Phelan et al.47). اشتقاق تركيز الخلية من رقم الخلية المحسوب باستخدام الصيغة التالية:
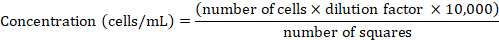
- بيليه الخلايا بواسطة طرد في 300 x g لمده 2 دقيقه. أعاده التعليق مع rpmi 1640 الطازجة من أجل الحصول علي 1 × 106 خلايا/مل.
- قم بازاله الفيبروكتين من المشبك الذي تم تنظيفه ، واغسل الآبار مرتين باستخدام DPBS ، والبذور 50,000 خلايا لكل بئر.
- العودة المشتركة في الزنزانات مع الخلايا إلى الحاضنة وتنمو لهم ل 24 ح في 37 درجه مئوية و 5 ٪ CO2.
2. التعبير عن pEB3-tdTomato في الخلايا هيلا
- اعداد أنبوب الطرد المركزي الصغير 1.5 mL. لكل أنبوب ، تمييع 2 ميكروغرام من pEB3-tdTomato48 مع العازلة ترانسفيكشن (المنتج الاصطناعية في محلول مائي) إلى حجم النهائي من 396 μl.
- أضافه 4 μL من الكاشف المتحول (غير الدهون ، التي تحتوي علي بوليثيلينمين) إلى الأنبوب الأول ، ودوامه الخليط علي الفور لمده 10 ليالي بالبالضبط.
- تدور لفتره وجيزة أسفل الأنبوب مع الطرد المركزي الصغير واحتضان في درجه حرارة الغرفة (RT) لمده 10 دقيقه.
- أزاله الخلايا هيلا من الحاضنة. القطرات ، أضافه 100 μL من خليط التحويل إلى كل بئر من 4 بالاضافه إلى ذلك ، وأعاده الخلايا إلى الحاضنة.
- بعد 4 ح من الحضانة في 37 درجه مئوية و 5 ٪ CO2، تكمله الخلايا مع النمو الطازج المتوسطة واحتضان لمده 24 ساعة علي الأقل في 37 درجه مئوية و 5 ٪ co2.
ملاحظه: من الضروري تحسين ظروف التحويل لكل نوع من أنواع الخلايا. يجب ان تكون مستويات التعبير منخفضه بما يكفي للسماح بتحديد نهايات النامية MT واحد. بدلا من ذلك ، يمكن استخدام خط الخلية التعبير بثبات EB3-tdTomato في التجارب. هذا من شانه ان يقلل من التباين في مستويات التعبير من EB3 بين الاستعدادات وبين الخلايا من نفس الاعداد49.
3. التزامن والتصوير الخلوي الحي لpEB3-التعبير عن الخلايا هيلا
- اعداد محلول 2.5 μM من ديميثيلسيناستراو (DME) في الفينول الأحمر الحرة دولبيكو النسر المعدلة المتوسطة (DMEM) تكمله مع 10 ٪ FCS و 2 مم L-الجلوتامين أو العرض الجلوتامين البديلة.
- استبدال المتوسطة النمو في الكوفيرسليب مع 500 μl من متوسط النمو التي تحتوي علي 2.5 μM DME واحتضان الخلايا في 37 درجه مئوية و 5 ٪ CO2.
- بعد 3.5 h من الحضانة مع DME ، نقل الخلايا إلى المجهر ، وجبل كوفيرسليب حجره في غرفه البيئية مع لوحات الظلام للتصوير في 37 درجه مئوية و 5 ٪ CO2، ومزيد من احتضان حتى وقت الحضانة الإجمالي هو 4 ساعة.
ملاحظه: الحفاظ علي درجه الحرارة عند 37 درجه مئوية بدون تذبذب أمر حاسم بالنسبة للتجربة. - أداء التصوير الفاصل الزمني علي المجهر المقلوب مجهزه 100x 1.49 N.A. الغمر النفط الهدف ، والمزدوج الغزل القرص الدوار النظام ، ونظام التركيز البؤري التلقائي موثوق بها للصيانة المستمرة للمستوي البؤري. تحديد معلمات التصوير كما يلي.
ملاحظه: نحن نستخدم الكترون ضرب الجهاز بالاضافه إلى اله الكاميرا (EM-CCD).- ل EB3 الاثاره ، واستخدام خط ليزر 561 nm مع الوقت التعرض 200 ms. جمع الضوء المنبعث من خلال تمريره رباعيه (405 ، 488 ، 561 ، 640 nm) مراه ديشروريك ومرشح الانبعاثات 600/52 nm.
ملاحظه: ويمكن تعديل قوه الليزر لكل خليه العمر لمنع تشبع الصورة. في جميع الأفلام الفاصلة الزمنيه المعطية هنا تم تعيين طاقة الليزر إلى 5.3 ميغاواط. - العثور علي خليه في طور والتركيز في Z-الطائرة المقابلة لمركز المغزل القطب الدوار. الحصول علي الصور كل 0.5 s علي ما مجموعه 1 دقيقه مع عدم التسلق ولا الاضاءه بين التعرض.
- ل EB3 الاثاره ، واستخدام خط ليزر 561 nm مع الوقت التعرض 200 ms. جمع الضوء المنبعث من خلال تمريره رباعيه (405 ، 488 ، 561 ، 640 nm) مراه ديشروريك ومرشح الانبعاثات 600/52 nm.
4. تحليل ديناميات MT باستخدام U-المسار الخامس 2.2.0
- لتحليل ديناميات MT البرمجيات الرقمية بيئة الحوسبة مطلوب (علي سبيل المثال ، MATLAB).
ملاحظه: الفهم الأساسي للبرمجيات كافيه للتحليل. مواد المساعدة الشاملة والدروس المتاحة علي موقع المطور (https://uk.mathworks.com/products/matlab/getting-started.html). - تحميل (https://github.com/DanuserLab/u-track) وتثبيت البرنامج المفتوح المصدر u-المسار الخامس 2.2.0 البرمجيات بعد التعليمات المفصلة الواردة في ملف "Readme_u-المسار. pdf"50,51,52.
- بدء تشغيل برنامج التحليل العددي وأضافه مجلد 2.2.0 v المسار مع الملفات الفرعية في مسار البحث عن البرامج.
- من اطار الأوامر المكالمة "Movieselectorgui". وهذا يفتح نافذه الحوار التي يمكن من خلالها استيراد الملفات الخام التي تم إنشاؤها بواسطة برنامج الحصول علي الصور في المجهر (الشكل التكميلي 1، الشكل 2، الشكل 3، الشكل 4).
ملاحظه: يتوافق برنامج U-Track مع تنسيقات بيانات الصور الأخرى. ويستخدم الاشكال الحيوية ، والتي تعترف مختلف اشكال البيانات العلوم الحياة53. - يتم قراءه حجم كل صوره من بيانات التعريف تلقائيا. ادخل الفتحة العددية للهدف (في هذه الحالة 1.49) والفترة الزمنيه (0.5 s) المستخدمة في التصوير يدويا (الشكل التكميلي 1B). بالاضافه إلى ذلك ، يمكن أيضا توفير معلومات عن الطول الموجي للاثاره ، والفلوريد ، ووقت التعرض ، ولكنها ليست حاسمه لمزيد من التحليل.
- بمجرد تحميل جميع الصور ، احفظ سلسله الفواصل الزمنيه التي تم إدخالها كقائمه أفلام عن طريق تحديد "حفظ باسم قائمه الأفلام". علي الجانب الأيمن من نافذه الحوار حدد الخيار "U-المسار" واضغط علي "مواصله" (الشكل التكميلي 1c).
ملاحظه: يتم تحسين القيم للخلايا هيلا. في حاله التبديل إلى خط خليه مختلف ، يجب تعريف القيم مره أخرى. بدلا من ذلك ، استخدم الإعدادات الموصي بها من قبل مطوري البرامج. ويمكن الاطلاع علي شرح مفصل لكل من المعلمات وكيفيه تعريفها في التقرير الفني المقدمة مع الإصدار السابق من البرنامج ، plusTipTracker50. - من النافذة المنبثقة حدد "ميكروتوبولي زائد نهايات" واضغط علي "Ok" (الشكل التكميلي 1c). وتتيح نافذه الحوار الجديدة تحديد بارامترات الخطوات الثلاث للتحليل (الشكل التكميلي 1 د) ، وهي الكشف والتتبع وتحليل المسار.
- في الخطوة 1 اختر "إعدادات" ومن القائمة المنسدلة حدد "الكشف عن المذنب" كطريقه الكشف (الشكل التكميلي 2B).
- من نافذه الحوار الجديدة تحديد المعلمات للفرق من فلتر Gaussians وتقسيم مستجمعات المياه علي النحو التالي (الشكل التكميلي 2C): عمليه قناع لاستخدامها للكشف = لا شيء ؛ انخفاض تمرير الانحراف المعياري غوسيه = 1 بكسل; عاليه تمريره غوسيه الانحراف المعياري = 3 بكسل; عتبه الحد الأدنى = 3 الانحرافات القياسية; عتبه حجم الخطوة = 0.25 الانحرافات القياسية. حدد "تطبيق الإعدادات علي كل الأفلام" و "تطبيق".
- في الخطوة 2 ، يتم تعريف المعلمات للربط ، وإغلاق الفجوة ، ودمج وتقسيم ، ووظائف التصفية كالمان في ثلاث خطوات كما هو موضح في الوردي والأخضر والأزرق ، وفقا لذلك (الشكل التكميلي 3B). لهذه الخطوات ، حدد "ميكروتوبولي بلس-النهاية ديناميات" ومن الخيار "اعداد" تعريف القيم كما هو موضح في الشكل التكميلي 3C-E، علي التوالي.
- بالنسبة للمشكلات المتعلقة بالابعاد ، اختر "2" من القائمة المنسدلة. استخدام الحد الأقصى للفجوة لإغلاق = 5 إطارات; الحد الأدنى لطول مقاطع المسار من الخطوة الاولي = 3 إطارات. كما كان من قبل ، حدد "تطبيق الإعدادات علي جميع الأفلام" وانقر علي "تطبيق".
- في الخطوة 3 من التحليل ، يتم تصنيف مسارات MT المكتشفة (الشكل التكميلي 4). وكطريقه لتحليل المسارات ، اختر "تصنيف ديناميات الميكروبوليه" وحدد المعلمات من خلال زر "الاعداد" كما هو مبين في الشكل التكميلي 4 ب ، ج. بعد ذلك ، اختر مربع "تطبيق الإعدادات علي جميع الأفلام" وانقر علي "تطبيق".
- مره واحده يتم تعريف جميع المعلمات ، من "لوحه التحكم-U-المسار" نافذه (الشكل التكميلي 1d) حدد "تطبيق الاختيار/إلغاء التاشير علي جميع الأفلام" و "تشغيل جميع الأفلام" مربعات واضغط علي "تشغيل". سيبدا هذا التحليل الخاص بسلسلة الفواصل الزمنيه.
- بمجرد اكتمال معالجه الفيلم ، يتم عرض رسالة "تمت معالجه الفيلم (الأفلام) الخاصة بك بنجاح". اضغط علي "Ok" ، ثم "حفظ".
- الآن فمن الأمن لإنهاء برنامج التحليل العددي. يتم تخزين النتائج من معالجه الفيلم في بنيه المجلدات الفرعية كملفات m في المجلد حيث يتم تخزين الملفات الاوليه.
5. التحليل الإحصائي لديناميات MT
- استيراد الملفات m إلى برنامج تحليل إحصائي المفضل.
ملاحظه: في حالتنا ، نقوم أولا باستيراد الملفات في جدول بيانات قياسي لجعلها قابله للقراءة. تحتوي الملفات m علي المعلومات الاحصائيه (المتوسط والمتوسط والانحراف المعياري) علي معلمات مختلفه (علي سبيل المثال ، سرعه النمو ، دينامسيتي MT). وترد قائمه مفصله من المعلمات في التقرير الفني المقدمة مع النسخة السابقة من البرنامج ، plustiptracker50،52. يمكن أيضا استيراد ملفات m التي تم إنشاؤها إلى برامج معالجه البيانات الأخرى. - اختر المعلمة "متوسط سرعه النمو" واستيرادها إلى جدول للإحصاءات والعرض. ادخل المعلومات الخاصة بالمعلمات الأخرى ، (علي سبيل المثال ، "ديناميستي") اما في جدول جديد أو في عمود جديد من نفس الجدول والمؤامرة المجمعة.
Access restricted. Please log in or start a trial to view this content.
النتائج
وبعد البروتوكول المحدد المبين في الشكل 1A، تم التعبير عن pEB3 في خلايا هيلا المتنامية بصوره غير متزامنة. تمت مزامنة الخلايا 48 h بعد التحويل في طليعة من خلال العلاج DME (الشكل 1b). وكفلت هذه الخطوة ان يكون قياس ديناميات MT دائما في نفس المرحلة من دوره الخلية. وتمت معا...
Access restricted. Please log in or start a trial to view this content.
Discussion
هنا ، ونحن وصف تعديلا للأسلوب الذي انشاته Ertych وآخرون44. مع العديد من التعديلات الأخرى ، ونحن الجمع بين هذه التقنية من تحليل ديناميات MT مع المزدوج الغزل القرص التصوير البؤري. استخدام القرص الدوار المزدوج يحسن دقه النامية MTs مع الحد من السمية الضوئية36. كما نقوم بتقلي...
Access restricted. Please log in or start a trial to view this content.
Disclosures
وليس لدي المؤلفين ما يفصحون عنه.
Acknowledgements
ونشكر أعضاء مرفق المجهر الضوئي ، معهد ماكس بلانك للطب التجريبي ، علي مشورتهم المتخصصة ودعمهم.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
References
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved