È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazione della microtubule Dynamics mediante microscopia disco rotante in spindle mitotici monopolari
In questo articolo
Riepilogo
Qui presentiamo un metodo robusto e dettagliato di analisi delle dinamiche dei microtubuli nelle cellule sincronizzate nella prometafase utilizzando la microscopia confocale del disco di filatura a cellule vive e l'elaborazione delle immagini basata su MATLAB.
Abstract
Descriviamo una modifica di un metodo stabilito per determinare la dinamica dei microtubuli nelle cellule viventi. Il protocollo si basa sull'espressione di un marcatore geneticamente codificato per le estremità positive dei microtubuli (EB3 etichettato con proteina fluorescente tdTomato) e dell'imaging ad alta velocità, ad alta risoluzione e a cellule vive utilizzando la microscopia confocale del disco rotante. La sincronizzazione del ciclo cellulare e l'aumento della densità dei microtubuli si ottengono inibendo la separazione centrosomica nelle cellule mitotiche e l'analisi della crescita viene eseguita utilizzando il software U-Track open-source. L'uso di una proteina fluorescente luminosa e rossa, in combinazione con la minore potenza laser e la riduzione del tempo di esposizione necessaria per la microscopia su disco rotante riduce la fototossicità e la probabilità di artefatti indotti dalla luce. Ciò consente di imaging di un maggior numero di cellule nella stessa preparazione, mantenendo le cellule in un mezzo di crescita in condizioni di coltura standard. Poiché l'analisi viene eseguita in modo automatico supervisionato, i risultati sono statisticamente robusti e riproducibili.
Introduzione
I microtubuli (MT) sono strutture altamente dinamiche che si trovano praticamente in tutte le cellule eucariotiche e in alcuni batteri1. Insieme ad actin e filamenti intermedi, scolpiscono il citoscheletro2,3. Divisione cellulare4, trasporto molecolare5, flagellir battendo6, la sensazione dell'ambiente circostante attraverso il ciglio primario7, udito (kinocio)8,9, embriogenesi10,11,12, invasione e metastasi13,14, e anche la formazione della memoria15,16,17,18, e molti altri processi si basano principalmente sulle MT. La partecipazione degli MT in tutti questi eventi sarebbe impossibile senza la loro notevole capacità di passare rapidamente dalla crescita (polimerizzazione) al restringimento (depolimerizzazione). Questa proprietà è descritta come instabilità dinamica19. La dinamicità MT è alterata in molte condizioni patologiche20,21,22. Quindi, determinare la natura di questa proprietà può aiutare a comprendere i meccanismi della malattia e successivamente il loro trattamento.
È stata sviluppata una lunga lista di metodi per l'analisi delle dinamiche MT, la maggior parte dei quali si basa noto su tecniche di imaging23. Inizialmente, sono stati utilizzati microscopi a luce a campo largo per osservare la formazione di polimeri di tubulina in vitro24. La scoperta di proteine di legame finale (EB) che si raccolgono a MT plus-end e lo sviluppo di metodi per etichettare fluorescenti proteine ha permesso di osservare il comportamento delle MT direttamente nelle cellule viventi con ampi campi e microscopi a confocalizzazione della confraternita25,26,27. Una proteina EB è la proteina di legame finale 3 (EB3)28; sovraesprimendo e tracciando EB3 fuso a una proteina fluorescente, MT più tassi di assemblaggio end possono essere determinati29,30.
La microscopia a fluorescenza a scansione laser confocale (CLSM) viene spesso utilizzata per seguire la dinamica MT. Tuttavia, questa tecnica di imaging rappresenta un alto rischio di fototossicità e fotosbiancamento, due processi indesiderati per l'imaging campione di cellule vive e dim31. Al fine di ottenere un migliore rapporto segnale-rumore, la potenza del laser e la durata dell'esposizione dovrebbero essere sufficientemente elevate senza danneggiare i campioni, e questo richiede di sacrificare la risoluzione in cambio di velocità. Un'alternativa adatta al CLSM è la microscopia a disco rotante32. Questa modalità di imaging si basa sull'uso di un disco Nipkow33, che consiste in un disco in movimento recante una serie di fori, e funziona in modo equivalente a molti microscopi CLS che immaginino lo stesso campione contemporaneamente34. Pertanto, la luce del laser illuminerà diverse regioni del campione contemporaneamente, ma manterrà la natura confocale. Il disco Nipkow, quindi, permette di ottenere immagini simili a CLSM ma più velocee e utilizzando meno potenza laser. Il disco Nipkow è stato ulteriormente migliorato da Yokogawa Electric, che ha introdotto un secondo disco con una serie di microlenti su di esso che dirigere individualmente la luce in un rispettivo foro stenopeico, riducendo ulteriormente la fototossicità e il fotosbiancamento35. Così, la microscopia a scansione laser su disco rotante è diventata un metodo di scelta per l'imaging delle cellule vive, e permette di ottenere immagini con alto rapporto segnale-rumore ad alta velocità31,36, che è fondamentale per risolvere segnali come quelli provenienti dalle estremità MT in rapida crescita.
Le dinamiche MT differiscono temporaneamente. Ad esempio, gli MT mitotici sono più dinamici di quelli interfase37,38. Analogamente, sono state osservate differenze nel tasso di crescita e nel restringimento anche all'interno della stessa fase del ciclo cellulare, come la mitosi39,40. Pertanto, per evitare la falsa raccolta dei dati, la misurazione delle dinamiche MT dovrebbe essere limitata a una stretta finestra di tempo durante il ciclo cellulare. Ad esempio, la misurazione della dinamica di MT nella prometafase può essere ottenuta trattando le cellule con dimetilnastronastron (DME), un analogo monastrolo che inibisce la cinematosina Eg541 e impedisce la formazione del mandrino mitotico bipolare42. L'inibizione delle cellule alla prometafase con l'inibitore Di Eg5 DME e altri derivati monastrol non influisce sulla dinamica MT43,44,45, che rende DME uno strumento utile per studiare la dinamica MT sia nelle cellule fisse che vive44.
Qui combiniamo il metodo di analisi della dinamica MT nelle cellule prometafase descritto da Ertych et al.44 con doppia imaging del disco rotante. Questo metodo consente di misurazione della dinamica MT nelle cellule prometaphase raccolte da un singolo piano focale con una maggiore frequenza di imaging, ma senza fotosbiancamento e fototossicità minima. Inoltre, come reporter fluorescente, utilizziamo proteine fluorescenti tomato (tdTomato) che hanno migliorato luminosità e fotostabilità rispetto alla proteina fluorescente verde (EGFP) ed è entusiasta di luce a bassa energia46. Pertanto, tdTomato richiede meno potenza laser per l'eccitazione ed è meno fototossico. Complessivamente, miglioriamo ulteriormente il metodo riducendo la fototossicità e migliorando la risoluzione e post-elaborazione necessarie per l'analisi delle dinamiche MT. Inoltre, creiamo una base per le modifiche future del metodo combinandolo con altre tecniche di sincronizzazione.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Semina delle cellule HeLa
- Preparare 2 mL di 5 soluzione fibronectina a 5 g/mL in salina tampobuffer di fosfato (PBS) e aggiungerne 450 l in ogni pozzetti di un coperchio 4 ben camerato (#1.5). Incubare lo scivolo per 15 min a 37 gradi centigradi e il 5% di CO2.
- Risciacquare le celle HeLa in crescita asincrona con Dulbecco's Phosped Buffered Saline (DPBS) e incubare con trypsin-EDTA (0,05%: 0,02%; w:v) per 5 min a 37 . Fermare la reazione enzimatica con l'aggiunta del Roswell Park Memorial Institute (RPMI) 1640 medio integrato con 10% di siero di vitello fetale inattivato dal calore (FCS) a 3:1 (v:v) rapporto di tripsina-EDTA aggiunto.
NOT: Le cellule HeLa sono state mantenute in RPMI 1640 medio integrato con 10% di fSIattiva di calore a 37 e 5% di CO2 e sono state regolarmente passaggiate una volta raggiunto l'80-90% di confluenza come descritto sopra. - Determinare la concentrazione cellulare utilizzando una camera Neubauer. Mescolare un'aliquota di 50 l'uno con il rapporto trypan blue a 1:1 (v:v) ( Contare solo le celle trypan blu-negative all'interno dei quattro quadrati grandi (per i dettagli vedere Phelan et al.47). Derivare la concentrazione di cella dal numero di cella conteggiato utilizzando la seguente formula:
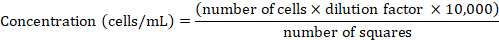
- Pellet le cellule per centrifugazione a 300 x g per 2 min. Risospendere con RPMI 1640 fresco al fine di ottenere 1 x 106 cellule / mL.
- Rimuovere la fibronectina dallo scivolo ricucibile, lavare i pozzetti due volte con DPBS e semire50.000 cellule per pozzo.
- Riportare l'incubatura con le cellule all'incubatrice e farle crescere per 24 h a 37 e 5% di CO2.
2. Espressione di pEB3-tdTomato nelle cellule HeLa
- Preparare un tubo di microcentrifuga da 1,5 ml. Per ogni tubo, diluire 2 g di pEB3-tdTomato48 con tampone di trasfezione (prodotto sintetico in soluzione acquosa) ad un volume finale di 396 gradi centigradi.
- Aggiungere 4 -L di reagente di trasfezione (non lipidico, contenente polietilenimine) al primo tubo e vorticare immediatamente la miscela per esattamente 10 s.
- Ruotare brevemente il tubo con una microcentrifuga e incubare a temperatura ambiente (RT) per 10 min.
- Rimuovere le cellule HeLa dall'incubatrice. A goccia, aggiungere 100 l' l della miscela di trasfezione ad ogni pozzetto di un 4 vetrino ben camerato, e riportare le cellule all'incubatrice.
- Dopo 4 h di incubazione a 37 e 5% di CO2, integrare le cellule con mezzo di crescita fresco e incubare per almeno 24 h a 37 e 5% CO2.
NOT: È necessario ottimizzare le condizioni di trasfezione per ogni tipo di cellula. I livelli di espressione devono essere sufficientemente bassi da consentire l'identificazione di singole estremità di crescita MT. In alternativa, negli esperimenti è possibile utilizzare una linea cellulare che esprime stabilmente EB3-tdTomato; ciò ridurrebbe la variabilità nei livelli di espressione di EB3-tdTomato tra i preparati e tra le cellule della stessa preparazione49.
3. Sincronizzazione e imaging a celle vive di cellule heLa che esprimono pEB3
- Preparare una soluzione di 2,5 M di dimetililnastron (DME) in privo di fenolo Dulbecco's Modified Eagle Medium (DMEM) completata con 10% FCS e 2 mM di glutammina o una fornitura alternativa di glutammina.
- Sostituire il mezzo di crescita nel coperchio camerato con 500 -L del mezzo di crescita contenente 2,5 M DME e incubare le cellule a 37 e 5% di CO2.
- Dopo 3,5 h di incubazione con DME, trasferire le cellule al microscopio, montare lo scivolo camerato in una camera ambientale con pannelli scuri per l'imaging a 37 e 5% di CO2e incubare ulteriormente fino a quando il tempo totale di incubazione è di 4 h.
NOT: Il mantenimento della temperatura a 37 gradi centigradi senza fluttuazioni è fondamentale per l'esperimento. - Eseguire l'imaging time-lapse su un microscopio invertito dotato di un obiettivo di immersione dell'olio 1.49 N.A.100x, un sistema confocale a doppio disco rotante e un sistema di messa a fuoco automatica affidabile per la manutenzione continua del piano focale. Definire i parametri di imaging come indicato di seguito.
NOT: Usiamo una fotocamera elettronica moltiplicante (EM-CCD).- Per l'eccitazione EB3-tdTomato, utilizzare una linea laser da 561 nm con tempi di esposizione di 200 ms. Raccogliere la luce emessa attraverso un quadruplo passa banda (405, 488, 561, 640 nm) specchio dicroico e un filtro di emissione 600/52 nm.
NOT: La potenza del laser può essere regolata per ogni cella immagine per evitare la saturazione dell'immagine. In tutti i film time-lapse dati qui la potenza laser è stata impostata a 5,3 mW. - Trovare una cella nella profase e mettere a fuoco nel piano z corrispondente al centro del mandrino mitotico monopolare. Acquisire immagini ogni 0,5 s su un totale di 1 min senza binning e senza illuminazione tra le esposizioni.
- Per l'eccitazione EB3-tdTomato, utilizzare una linea laser da 561 nm con tempi di esposizione di 200 ms. Raccogliere la luce emessa attraverso un quadruplo passa banda (405, 488, 561, 640 nm) specchio dicroico e un filtro di emissione 600/52 nm.
4. Analisi delle dinamiche MT utilizzando U-Track v2.2.0
- Per analizzare la dinamica MT è necessario un software di ambiente di elaborazione numerico (ad esempio, MATLAB).
NOT: La comprensione di base del software è sufficiente per l'analisi. Il materiale di aiuto completo e le esercitazioni sono disponibili sul sito Web dello sviluppatore (https://uk.mathworks.com/products/matlab/getting-started.html). - Scaricare (https://github.com/DanuserLab/u-track) e installare il software open-source U-Track v2.2.0 seguendo le istruzioni dettagliate fornite nel file "Readme_u-track.pdf"50,51,52.
- Avviare il software di analisi numerica e aggiungere la cartella U-Track v2.2.0 con sottocartelle nel percorso di ricerca del software.
- Dalla finestra di comando chiamare "movieSelectorGUI". Si apre una finestra di dialogo da cui è possibile importare i file non elaborati generati dal software di acquisizione delle immagini al microscopio (Figura supplementare 1, Figura 2, Figura 3, Figura 4).
NOT: Il software U-Track è compatibile con altri formati di dati immagine. Utilizza Bio-Formati, che riconosce diversi formati di dati di life science53 . - Le dimensioni di ogni immagine vengono lette automaticamente dai metadati. Immettere manualmente l'apertura numerica dell'obiettivo (in questo caso 1,49) e l'intervallo di tempo (0,5 s) utilizzato per l'imaging (Figura supplementare 1B). Inoltre, possono essere fornite informazioni sulla lunghezza d'onda dell'eccitazione, sul fluoroforo e sul tempo di esposizione, ma non sono fondamentali per ulteriori analisi.
- Una volta caricate tutte le immagini, salvare la serie time-lapse inserita come elenco di film selezionando l'"elenco " Salva come filmato". Sul lato destro della finestra di dialogo selezionare l'opzione "U-Track" e premere "Continua" (Figura supplementare 1C).
NOT: I valori sono ottimizzati per le celle HeLa. Se si passa a una linea di celle diversa, i valori devono essere definiti di nuovo. In alternativa, utilizzare le impostazioni consigliate dagli sviluppatori di software. La spiegazione dettagliata di ciascuno dei parametri e come dovrebbero essere definiti può essere trovata nella relazione tecnica fornita con la versione precedente del software, plusTipTracker50. - Dalla finestra pop-up selezionare "Microtubule Plus-Ends" e premere "Ok" (Figura supplementare 1C). La nuova finestra di dialogo consente di determinare i parametri per i tre passaggi dell'analisi (Supplementary Figure 1D), che sono il rilevamento, il rilevamento e l'analisi del tracciato.
- Nel passaggio 1 scegliere "Impostazioni" e da un menu a discesa selezionare "Rilevamento cometa" come metodo di rilevamento (Figura supplementare 2B).
- Dalla nuova finestra di dialogo definire i parametri per la differenza del filtro gaussiani e la segmentazione spartiacque come segue (Supplementary Figure 2C): Processo di maschera da utilizzare per il rilevamento - Nessuno; Deviazione standard gaussiana a passate basse: 1 pixel; Deviazione standard gaussiana ad alto passaggio : 3 pixel; Soglia minima: 3 deviazioni standard; Dimensione del gradino della soglia: 0,25 deviazioni standard. Selezionare "Applica impostazioni a tutti i filmati" e "Applica".
- Nel passaggio 2, i parametri per il collegamento, la chiusura dello spazio, l'unione e la divisione e le funzioni di filtro Kalman sono definiti in tre passaggi come evidenziato in rosa, verde e blu, di conseguenza (Figura supplementare 3B). Per questi passaggi, selezionare "Microtubule Plus-end Dynamics" e dall'opzione "Impostazione" definire i valori indicati rispettivamente nella figura supplementare 3C-E.
- Per i problemi di dimensionalità, scegliere "2" dal menu a discesa. Utilizzare Il divario massimo per chiudere 5 fotogrammi; Lunghezza minima dei segmenti di traccia dal primo passaggio : 3 fotogrammi. Come prima, selezionare "Applica impostazioni a tutti i film" e fare clic su "Applica".
- Nel passaggio 3 dell'analisi, le tracce MT rilevate sono classificate (Figura supplementare 4). Come metodo di analisi della traccia, scegliere "Microtubule Dynamics Classification" e definire i parametri tramite il pulsante "Impostazione" come indicato nella Figura supplementare 4B,C. Dopo di che, scegliere la "Applica impostazioni a tutti i film" casella e fare clic su "Applica".
- Una volta definiti tutti i parametri, dalla finestra " Pannello dicontrollo–U-Track" (Figura supplementare 1D) selezionare le caselle "Applica controllo/Deseleziona a tutti i film" e " Esegui tutti ifilm" e premere "Esegui". In questo modo verrà avviata l'analisi MT della serie time-lapse.
- Una volta completata l'elaborazione del filmato, viene visualizzato il messaggio "I film sono stati elaborati correttamente". Premere "Ok", quindi"Salva".
- Ora è sicuro di uscire dal software di analisi numerica. I risultati dell'elaborazione del filmato vengono memorizzati in strutture di sottocartelle come file m nella cartella in cui sono archiviati i file non elaborati.
5. Analisi statistica della MT Dynamics
- Importare i file m in un programma di analisi statistica preferito.
NOT: Nel nostro caso, importiamo prima i file in un foglio di calcolo standard per renderli leggibili. I file m contengono informazioni statistiche (mediana, media e deviazione standard) su diversi parametri (ad esempio, velocità di crescita, dinamicità MT). L'elenco dettagliato dei parametri è riportato nella relazione tecnica fornita con la versione precedente del software, plusTipTracker50,52. I file m generati possono anche essere importati in altri software di elaborazione dati. - Scegliere il parametro "media velocità di crescita" e importarlo in una tabella per le statistiche e la visualizzazione. Immettere le informazioni su altri parametri (ad esempio, "dynamicity") in una nuova tabella o in una nuova colonna della stessa tabella raggruppata e del grafico.
Access restricted. Please log in or start a trial to view this content.
Risultati
Seguendo il protocollo descritto nella figura 1A, il plasmidp pEB3-tdTomato è stato espresso in modo temporaneo in celle HeLa in crescita asincrona. Le cellule sono state sincronizzate 48 h dopo la trasfezione a prometafase attraverso il trattamento DME(Figura 1B). Questo passaggio ha assicurato che la misurazione della dinamica MT è sempre stata eseguita nella stessa fase del ciclo cellulare. I filmati time-lapse sono stati ulteriormente elaborati e analizzat...
Access restricted. Please log in or start a trial to view this content.
Discussione
Qui, descriviamo una modifica di un metodo stabilito per la prima volta da Ertych et al.44. Insieme a diverse altre modifiche, combiniamo questa tecnica di analisi della dinamica MT con l'imaging confocale a doppio disco rotante. L'uso del doppio disco rotante migliora la risoluzione delle MT in crescita riducendo la fototossicità36. Riduciamo ulteriormente il fotosbiancamento e i danni indotti dalla luce laser delle cellule passando a un reporter fluorescente a lunghezza ...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Ringraziamo i membri della Light Microscopy Facility, Max-Planck Institute of Experimental Medicine, per la loro consulenza e supporto di esperti.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
Riferimenti
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon