Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Mesure de la dynamique des microtubules par la microscopie du disque filature dans des spindles mitotiques monopolaires
Dans cet article
Résumé
Ici nous présentons une méthode robuste et détaillée d'analyse de dynamique de microtubule dans les cellules synchronisées dans prometaphase utilisant la microscopie confocale de disque de filature de cellules vivantes et le traitement d'image MATLAB-basé.
Résumé
Nous décrivons une modification d'une méthode établie pour déterminer la dynamique de microtubule dans les cellules vivantes. Le protocole est basé sur l'expression d'un marqueur génétiquement codé pour les extrémités positives des microtubules (EB3 étiquetés avec la protéine fluorescente tdTomato) et à haute vitesse, haute résolution, imagerie à cellules vivantes à l'aide de la microscopie confocale du disque filature. La synchronisation du cycle cellulaire et l'augmentation de la densité des microtubules sont obtenues en inhibant la séparation centrosomale dans les cellules mitotiques, et l'analyse de la croissance est effectuée à l'aide du logiciel Open-Track. L'utilisation d'une protéine fluorescente brillante et rouge décalée, en combinaison avec la puissance laser inférieure et le temps d'exposition réduit requis pour la microscopie de disque de rotation réduisent la phototoxicité et la probabilité des artefacts induits par la lumière. Cela permet d'imagerie d'un plus grand nombre de cellules dans la même préparation tout en maintenant les cellules dans un milieu de croissance dans des conditions de culture standard. Étant donné que l'analyse est effectuée de façon automatique supervisée, les résultats sont statistiquement robustes et reproductibles.
Introduction
Les microtubules (MT) sont des structures très dynamiques que l'on trouve dans pratiquement toutes les cellules eucaryotes et dans certaines bactéries1. Avec l'actine et les filaments intermédiaires, ils sculptent le cytosquelette2,3. Division cellulaire4, transport de molécules5, flagellateur battant6, la sensation de l'environnement environnant à travers le cilium primaire7, audition (kinocilium)8,9, embryogenèse10,11,12, invasion et métastes13,14, et même la formation de la mémoire15,16,17,18, et beaucoup d'autres processus reposent principalement sur les MT. La participation des MT à tous ces événements serait impossible sans leur remarquable capacité à passer rapidement de la croissance (polymérisation) au rétrécissement (dépolymérisation). Cette propriété est décrite comme l'instabilité dynamique19. La dynamique MT est altérée dans de nombreuses conditions pathologiques20,21,22. Par conséquent, la détermination de la nature de cette propriété peut aider à comprendre les mécanismes de la maladie et, par la suite, leur traitement.
Une longue liste de méthodes a été développée pour l'analyse de la dynamique MT, dont la plupart sont basées sur des techniques d'imagerie23. Initialement, des microscopes à lumière à large champ ont été utilisés pour observer la formation de polymères de tubuline in vitro24. La découverte de protéines liantes finales (EB) qui s'accumulent à MT plus-ends et le développement de méthodes pour étiqueter les protéines fluorescentes ont permis d'observer le comportement des MT directement dans les cellules vivantes avec large champ et microscopes à fluorescence confocale25,26,27. Une protéine EB est la protéine liant fin 3 (EB3)28; en surexprimant et en suivant EB3 fusionné à une protéine fluorescente, les taux d'assemblage MT plus-extrémité peuvent être déterminés29,30.
La microscopie par fluorescence par laser confocal (CLSM) est fréquemment utilisée pour suivre la dynamique du MT. Cependant, cette technique d'imagerie pose un risque élevé de phototoxicité et de photoblanchiment, deux processus indésirables pour les cellules vivantes et l'imagerie de l'échantillon faible31. Afin d'obtenir un meilleur rapport signal-bruit, la puissance laser et la durée d'exposition doivent être suffisamment élevées tout en n'endommageant pas les échantillons, ce qui nécessite de sacrifier la résolution en échange de la vitesse. Une alternative appropriée au CLSM est la microscopie de disque tournante32. Cette modalité d'imagerie est basée sur l'utilisation d'un disque Nipkow33, qui se compose d'un disque en mouvement portant un tableau de trous d'épingle, et fonctionne de façon équivalente à de nombreux microscopes CLS imagerie du même échantillon simultanément34. Par conséquent, la lumière du laser illuminera plusieurs régions de l'échantillon simultanément, mais conservera la nature confocale. Le disque Nipkow permet donc d'obtenir des images similaires à CLSM, mais plus rapidement et en utilisant moins de puissance laser. Le disque Nipkow a été encore amélioré par Yokogawa Electric, qui a introduit un deuxième disque avec un tableau de microlentilles sur elle que la lumière individuellement diriger dans un trou d'épingle respectif, réduisant encore la phototoxicité et photoblanchiment35. Ainsi, la microscopie de balayage laser de disque de rotation est devenue une méthode de choix pour l'imagerie cellulaire vivante, et il permet d'obtenir des images avec le rapport signal-bruit élevé à une vitesse élevée31,36, qui est crucial pour résoudre des signaux tels que ceux des extrémités de MT à croissance rapide.
La dynamique MT diffère temporairement. Par exemple, les MT mitotiques sont plus dynamiques que les MTinterphases 37,38. De même, des différences dans le taux de croissance et le rétrécissement ont été observées même au sein de la même phase du cycle cellulaire, comme la mitose39,40. Par conséquent, pour éviter la collecte de fausses données, la mesure de la dynamique MT devrait être limitée à une fenêtre temporelle étroite pendant le cycle cellulaire. Par exemple, la mesure de la dynamique de MT dans prometaphase peut être réalisée en traitant les cellules avec le diméthylenastron (DME), un analogue monastrol qui inhibe la kinésine de moteur Eg541 et empêche la formation du fuseau mitotique bipolaire42. L'inhibition des cellules à proméaphase avec Eg5 inhibiteur DME et autres dérivés monastrol n'affecte pas la dynamique MT43,44,45, ce qui rend DME un outil utile pour étudier la dynamique MT à la fois dans les cellules fixes et vivantes44.
Ici nous combinons la méthode de l'analyse de dynamique de MT dans les cellules de proméaphase décrites par Ertych et autres44 avec la formation image de disque de double rotation. Cette méthode permet de mesurer la dynamique mT dans les cellules proméaphase recueillies à partir d'un seul plan focal avec un taux d'imagerie plus élevé, mais sans photoblanchiment et phototoxicité minimale. En outre, en tant que journaliste fluorescent, nous utilisons tandem dimer protéine fluorescente tomate (tdTomato) qui a amélioré la luminosité et la photostabilité par rapport à la protéine fluorescente verte (EGFP) et est excité avec une lumière d'énergie plus faible46. Par conséquent, tdTomato nécessite moins de puissance laser pour l'excitation et est moins phototoxique. Au total, nous améliorons encore la méthode en réduisant la phototoxicité et en améliorant la résolution et le post-traitement requis pour l'analyse de la dynamique MT. En outre, nous créons une base pour les modifications futures de la méthode en la combinant avec d'autres techniques de synchronisation.
Access restricted. Please log in or start a trial to view this content.
Protocole
1. Ensemencement des cellules HeLa
- Préparer 2 ml de solution de fibronectin de 5 g/mL dans la saline tamponnée au phosphate (PBS) et en ajouter 450 l dans chaque puits d'un bordereau de couverture de 4 puits bien chambré (#1,5). Incuber la glissière pendant 15 min à 37 oC et 5 % de CO2.
- Rincer asynchronement les cellules HeLa en croissance asynchrone avec la saline tamponnée de phosphate (DPBS) de Dulbecco et incuber avec la trypsine-EDTA (0,05 % : 0,02 %; w:v) pendant 5 min à 37 oC. Arrêtez la réaction enzymatique par l'ajout de Roswell Park Memorial Institute (RPMI) 1640 moyen complété par 10% de sérum fœtal de veau (FCS) inactivé par la chaleur à 3:1 (v:v) ratio de trypsin-EDTA ajouté.
REMARQUE: Les cellules de HeLa ont été maintenues dans le milieu de RPMI 1640 complété avec 10% DE FCS inactivé par la chaleur à 37 oC et 5% de CO2 et ont été systématiquement passages une fois qu'ils ont atteint 80-90% de confluency comme décrit ci-dessus. - Déterminer la concentration cellulaire à l'aide d'une chambre Neubauer. Mélanger un aliquot de 50 l de la suspension de la cellule avec le bleu trypan à 1:1 (v:v) ratio, resuspendre, et transférer 10 L de la suspension dans la chambre. Ne comptez que les cellules trypan bleu-négatif à l'intérieur des quatre grands carrés (pour plus de détails voir Phelan et al.47). Détirez la concentration cellulaire du numéro de cellule compté à l'aide de la formule suivante :
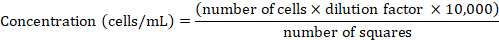
- Pelleter les cellules par centrifugation à 300 x g pendant 2 min. Resuspendre avec LE RPMI 1640 frais afin d'obtenir 1 x 106 cellules/mL.
- Retirer la fibronectine de la couverture chambrée, laver les puits deux fois avec DPBS, et les graines 50.000 cellules par puits.
- Remettre le bordereau chambré avec les cellules à l'incubateur et les faire pousser pendant 24 h à 37 oC et 5 % de CO2.
2. Expression de pEB3-tdTomato dans les cellules HeLa
- Préparer un tube microcentrifuge de 1,5 ml. Pour chaque tube, diluer 2 g de pEB3-tdTomato48 avec un tampon de transfection (produit synthétique en solution aqueuse) à un volume final de 396 l.
- Ajouter 4 ll de réactif transfection (non lipidique, contenant de la polyéthylène) au premier tube, et vortexler le mélange immédiatement pour exactement 10 s.
- Faites tourner brièvement le tube à l'eau à l'air microcentrifugeet incubez à température ambiante (RT) pendant 10 min.
- Retirez les cellules HeLa de l'incubateur. Dans le sens du drop, ajouter 100 L du mélange de transfection à chaque puits d'un bordereau de couverture de 4 bien chambrés, et retourner les cellules à l'incubateur.
- Après 4 h d'incubation à 37 oC et 5 % de CO2,complétez les cellules avec un milieu de croissance frais et incubez pendant au moins 24 h à 37 oC et 5 % CO2.
REMARQUE: Il est nécessaire d'optimiser les conditions de transfection pour chaque type de cellule. Les niveaux d'expression doivent être suffisamment bas pour permettre l'identification des extrémités de croissance de MT simples. Alternativement, une ligne cellulaire exprimant de façon stable EB3-tdTomato peut être utilisée dans les expériences; cela réduirait la variabilité des niveaux d'expression de EB3-tdTomato entre les préparations et entre les cellules de la même préparation49.
3. Synchronisation et imagerie à cellules vivantes des cellules HeLa pEB3-tdTomato-expressing
- Préparer une solution de 2,5 M de diméthylenastron (DME) dans le milieu d'aigle modifié (DMEM) de Dulbecco, sans phénol-rouge, complétée par 10 % de FCS et 2 mM de L-glutamine ou un autre approvisionnement en glutamine.
- Remplacer le milieu de croissance dans le bordereau chambré par 500 L du milieu de croissance contenant 2,5 M DME et incuber les cellules à 37 oC et 5 % de CO2.
- Après 3,5 h d'incubation avec DME, transférer les cellules au microscope, monter le bordereau chambré dans une chambre environnementale avec des panneaux sombres pour l'imagerie à 37 oC et 5% CO2, et incuber jusqu'à ce que le temps d'incubation total soit de 4 h.
REMARQUE: Le maintien de la température à 37 oC sans fluctuation est crucial pour l'expérience. - Effectuez l'imagerie en time-lapse sur un microscope inversé équipé d'un objectif d'immersion d'huile N.A. 100x 1,49, d'un système confocal à double rotation du disque et d'un système d'autofocus fiable pour l'entretien continu du plan focal. Définissez les paramètres d'imagerie comme suit.
REMARQUE: Nous utilisons une caméra Electron Multiplying Charge-Coupled Device (EM-CCD).- Pour eb3-tdTomato excitation, utilisez une ligne laser de 561 nm avec un temps d'exposition de 200 ms. Recueillir la lumière émise à travers un quadruple bandpass (405, 488, 561, 640 nm) miroir dichroic et un filtre d'émission de 600/52 nm.
REMARQUE: La puissance laser peut être ajustée pour chaque cellule image afin d'éviter la saturation de l'image. Dans tous les films en time-lapse donnés ici la puissance laser a été fixé à 5,3 mW. - Trouvez une cellule en prophase et concentrez-vous dans le plan Z correspondant au centre du fuseau mitotique monopolaire. Acquérir des images tous les 0,5 s sur un total de 1 min sans binning et sans éclairage entre les expositions.
- Pour eb3-tdTomato excitation, utilisez une ligne laser de 561 nm avec un temps d'exposition de 200 ms. Recueillir la lumière émise à travers un quadruple bandpass (405, 488, 561, 640 nm) miroir dichroic et un filtre d'émission de 600/52 nm.
4. Analyse de la dynamique MT à l'aide de U-Track v2.2.0
- Pour analyser la dynamique MT, un logiciel d'environnement informatique numérique est nécessaire (p. ex., MATLAB).
REMARQUE: La compréhension de base du logiciel est suffisante pour l'analyse. Des documents d'aide complets et des tutoriels sont disponibles sur le site Web du développeur (https://uk.mathworks.com/products/matlab/getting-started.html). - Téléchargez (https://github.com/DanuserLab/u-track) et installez le logiciel open-source U-Track v2.2.0 suivant les instructions détaillées données dans le fichier "Readme_u-track.pdf"50,51,52.
- Lancez le logiciel d'analyse numérique et ajoutez le dossier U-Track v2.2.0 avec des sous-dossiers dans le chemin de recherche du logiciel.
- De la fenêtre de commande appeler "movieSelectorGUI". Cela ouvre une fenêtre de dialogue à partir de laquelle les fichiers bruts générés par le logiciel d'acquisition d'images au microscope peuvent être importés (Figure supplémentaire 1, Figure 2, Figure 3, Figure 4).
REMARQUE: Le logiciel U-Track est compatible avec d'autres formats de données d'image. Il utilise Bio-Formats, qui reconnaît différents formats de données des sciences de la vie53. - La taille de chaque image est lue automatiquement à partir des métadonnées. Entrez manuellement l'ouverture numérique de l'objectif (dans ce cas 1,49) et l'intervalle de temps (0,5 s) utilisé pour l'imagerie(figure supplémentaire 1B). En outre, des informations sur la longueur d'onde de l'excitation, le fluorophore et le temps d'exposition peuvent également être fournies, mais elles ne sont pas essentielles pour une analyse plus approfondie.
- Une fois que toutes les images sont chargées, enregistrez la série time-lapse saisie comme une liste de films en sélectionnant la "Save As Movie List". Sur le côté droit de la fenêtre de dialogue sélectionnez l'option "U-Track" et appuyez sur "Continuer" (Figure supplémentaire 1C).
REMARQUE: Les valeurs sont optimisées pour les cellules HeLa. Si vous passez à une autre ligne cellulaire, les valeurs doivent être définies à nouveau. Vous pouvez également utiliser les paramètres recommandés par les développeurs de logiciels. L'explication détaillée de chacun des paramètres et comment ils doivent être définis peut être trouvée dans le rapport technique fourni avec la version précédente du logiciel, plusTipTracker50. - De la fenêtre pop-up sélectionnez "Microtubule Plus-Ends" et appuyez sur "Ok"(figure supplémentaire 1C). La nouvelle fenêtre de dialogue permet de déterminer les paramètres des trois étapes de l'analyse(figure supplémentaire 1D), qui sont la détection, le suivi et l'analyse de la voie.
- À l'étape 1, choisissez « Paramètres » et à partir d'un menu déroulant sélectionnez « Détection de comètes » comme méthode de détection(figure supplémentaire 2B).
- À partir de la nouvelle fenêtre de dialogue, définissez les paramètres de la différence du filtre Gaussians et la segmentation du bassin versant comme suit (Figure 2C supplémentaire) : Processus de masque à utiliser pour la détection - Aucun; Déviation standard Gaussian à faible passage 1 pixel; Déviation standard gaussienne à passage élevé de 3 pixels; Seuil minimum : 3 écarts-types; Taille de l'étape de seuil : 0,25 écart séminence. Sélectionnez "Appliquer paramètres à tous les films" et "Appliquer".
- À l'étape 2, les paramètres de liaison, de fermeture d'écart, de fusion et de fractionnement, et les fonctions de filtre Kalman sont définis en trois étapes comme surligné en rose, vert et bleu, en conséquence (Figure supplémentaire 3B). Pour ces étapes, sélectionnez la « dynamique Microtubule Plus-end » et de l'option « Réglage » définissent les valeurs indiquées dans la figure 3C-E supplémentaire,respectivement.
- Pour les problèmes de dimensionnalité, choisissez "2" dans le menu déroulant. Utilisez l'écart maximal pour fermer 5 images; Longueur minimale des segments de voie à partir de la première étape et 3 images. Comme avant, sélectionnez "Appliquer paramètres à tous les films" et cliquez sur "Appliquer".
- À l'étape 3 de l'analyse, les traces MT détectées sont classées (Figure supplémentaire 4). Comme méthode d'analyse de piste, choisissez « Classification dela dynamique microtubule» et définissez les paramètres à travers le bouton «Réglage» tel qu'indiqué dans la figure supplémentaire 4B,C. Après cela, choisissez le "Apply Settings to All Movies" box and click on "Apply".
- Une fois que tous les paramètres sont définis, à partir de la fenêtre "Control Panel-U-Track" (Additional Figure 1D) sélectionnez le " ApplyCheck/Uncheck to All Movies" et " Run AllMovies" cases and press "Run". Cela permettra d'initier l'analyse MT de la série time-lapse.
- Une fois le traitement du film terminé, un message "Votre film a été traité avec succès" est affiché. Appuyez sur "Ok", puis "Save".
- Maintenant, il est sûr de quitter le logiciel d'analyse numérique. Les résultats du traitement du film sont stockés dans des structures de sous-dossier sous-fichiers dans le dossier où les fichiers bruts sont stockés.
5. Analyse statistique de la dynamique MT
- Importer les fichiers m dans un programme d'analyse statistique préféré.
REMARQUE: Dans notre cas, nous importons d'abord les fichiers dans une feuille de calcul standard pour les rendre lisibles. Les fichiers m contiennent des informations statistiques (écart médian, moyen et standard) sur différents paramètres (p. ex., vitesse de croissance, dynamique MT). La liste détaillée des paramètres est donnée dans le rapport technique fourni avec la version précédente du logiciel, plusTipTracker50,52. Les fichiers m générés peuvent également être importés dans d'autres logiciels de traitement de données. - Choisissez le paramètre « moyenne de vitesse de croissance » et importez-le dans un tableau pour les statistiques et l'affichage. Entrez les informations sur d'autres paramètres (p. ex., « dynamique ») soit dans une nouvelle table, soit dans une nouvelle colonne de la même table et d'une même parcelle groupées.
Access restricted. Please log in or start a trial to view this content.
Résultats
Suivant le protocole donné décrit dans la figure 1A,le plasmide pEB3-tdTomato a été exprimé transitoirement dans les cellules De HeLa en croissance asynchrone. Les cellules ont été synchronisées 48 h après la transfection à proméaphase par le traitement DME (Figure 1B). Cette étape a permis de s'assurer que la mesure de la dynamique mT était toujours effectuée à la même phase du cycle cellulaire. Les films en time-lapse ont été traités et anal...
Access restricted. Please log in or start a trial to view this content.
Discussion
Ici, nous décrivons une modification d'une méthode établie pour la première fois par Ertych et al.,44. Avec plusieurs autres modifications, nous combinons cette technique d'analyse de dynamique de MT avec l'imagerie confocale de disque de rotation double. L'utilisation du disque à double filature améliore la résolution des MT en croissance tout en réduisant la phototoxicité36. Nous réduisons davantage les dommages causés par le photoblanchiment et la lumière las...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Nous remercions les membres de l'installation de microscopie légère, Max-Planck Institute of Experimental Medicine, pour leurs conseils et leur soutien.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
Références
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon