Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Messung der Mikrotubuli-Dynamik durch Spinnen der Disk-Mikroskopie in Monopolar-Mitotischen Spindeln
In diesem Artikel
Zusammenfassung
Hier stellen wir eine robuste und detaillierte Methode der Mikrotubuli-Dynamikanalyse in Zellen vor, die in der Prometaphase mit Live-Zell-Spinnscheibe konfokaler Mikroskopie und MATLAB-basierter Bildverarbeitung synchronisiert werden.
Zusammenfassung
Wir beschreiben eine Modifikation einer etablierten Methode zur Bestimmung der Mikrotubuli-Dynamik in lebenden Zellen. Das Protokoll basiert auf der Expression eines genetisch kodierten Markers für die positiven Enden von Mikrotubuli (EB3 mit tdTomato fluoreszierendem Protein) und hochgeschwindigkeits-, hochauflösender Live-Zell-Bildgebung mittels konokaler Spinnenvon-Mikroskopie. Die Zellzyklussynchronisierung und die erhöhte Dichte von Mikrotubuli werden durch Hemmung der zentrosomalen Trennung in mitotischen Zellen erreicht, und die Analyse des Wachstums wird mit Open-Source-U-Track-Software durchgeführt. Die Verwendung eines hellen und rot verschiebbaren fluoreszierenden Proteins in Kombination mit der geringeren Laserleistung und der reduzierten Belichtungszeit, die für die Spinnendere benötigt wird, reduziert die Phototoxizität und die Wahrscheinlichkeit lichtinduzierter Artefakte. Dies ermöglicht die Abbildung einer größeren Anzahl von Zellen in der gleichen Zubereitung unter Beibehaltung der Zellen in einem Wachstumsmedium unter Standardkulturbedingungen. Da die Analyse in einer überwachten automatischen Weise durchgeführt wird, sind die Ergebnisse statistisch robust und reproduzierbar.
Einleitung
Mikrotubuli (MTs) sind hochdynamische Strukturen, die in praktisch allen eukaryotischen Zellen und in einigen Bakterien gefunden werden1. Zusammen mit Aktin und Zwischenfilamenten formen sie das Zytoskelett2,3. Zellteilung4, Molekültransport5, Flagellar schlagen6, das Gefühl der Umgebung durch primärecilium7, Hören (Kinocilium)8,9, Embryogenese10,11,12, Invasion und Metastasierung13,14, und sogar Gedächtnisbildung15,16,17,18, und viele andere Prozesse verlassen sich in erster Linie auf MTs. Die Teilnahme von MTs an all diesen Ereignissen wäre ohne ihre bemerkenswerte Fähigkeit, schnell zwischen Wachstum (Polymerisation) und Schrumpfung (Depolymerisation) zu wechseln, nicht möglich. Diese Eigenschaft wird als dynamische Instabilität19beschrieben. MT-Dynamik wird unter vielen pathologischen Bedingungen verändert20,21,22. Daher kann die Bestimmung der Art dieser Eigenschaft helfen, Krankheitsmechanismen und anschließend ihre Behandlung zu verstehen.
Für die MT-Dynamikanalyse wurde eine lange Liste von Methoden entwickelt, von denen die meisten auf bildgebenden Verfahren basieren23. Ursprünglich wurden Weitfeldlichtmikroskope zur Beobachtung der Bildung von Tubulinpolymeren in vitro24eingesetzt. Die Entdeckung von Endbindungsproteinen (EB)-Proteinen, die sich an MT-Plus-Ends sammeln, und die Entwicklung von Methoden zur fluoreszierenden Kennzeichnung von Proteinen ermöglichten es, das Verhalten von MTs direkt in lebenden Zellen mit Weitfeld- und konfokalen Fluoreszenzmikroskopen25,26,27zu beobachten. Ein EB-Protein ist endbindendes Protein 3 (EB3)28; durch Überexpression und Verfolgung von EB3, das zu einem fluoreszierenden Protein verschmolzen wird, können MT-Plus-End-Montageraten bestimmt werden29,30.
Die konfokale Laserscanning-Fluoreszenzmikroskopie (CLSM) wird häufig verwendet, um der MT-Dynamik zu folgen. Diese bildgebende Technik stellt jedoch ein hohes Risiko für Phototoxizität und Photobleaching, zwei unerwünschte Prozesse für lebende Zellen und dim Probenbildgebung31. Um ein besseres Signal-Rausch-Verhältnis zu erreichen, sollten die Laserleistung und die Belichtungsdauer hoch genug sein, ohne die Proben zu beschädigen, und dies erfordert eine Aufblätung der Auflösung im Austausch für Geschwindigkeit. Eine geeignete Alternative zu CLSM ist die Spinnscheibenmikroskopie32. Diese bildgebende Modalität basiert auf der Verwendung einer Nipkow-Scheibe33, die aus einer beweglichen Scheibe besteht, die eine Reihe von Lochlöchern trägt, und arbeitet äquivalent zu vielen CLS-Mikroskopen, die die gleiche Probe gleichzeitig darstellen34. Daher leuchtet das Licht des Lasers mehrere Bereiche in der Probe gleichzeitig, behält aber die konfokale Natur bei. Die Nipkow-Festplatte ermöglicht es daher, Bilder zu erhalten, die CLSM ähneln, aber schneller und mit weniger Laserleistung. Die Nipkow-Scheibe wurde von Yokogawa Electric weiter verbessert, die eine zweite Scheibe mit einer Reihe von Mikrolinsen auf sie einführte, die einzeln Licht in ein entsprechendes Loch leiten, wodurch Phototoxizität und Photobleichung weiter reduziert werden35. So wurde die Spinnscheiben-Laserscanmikroskopie zu einer Methode der Wahl für die Live-Zell-Bildgebung, und es ermöglicht, Bilder mit einem hohen Signal-Rausch-Verhältnis bei einer hohen Geschwindigkeitvon 31,36zu erhalten, was für das Auflösen von Signalen wie denen von den schnell wachsenden MT-Enden entscheidend ist.
Mt-Dynamik unterscheidet sich vorübergehend. Beispielsweise sind die mitotischen MTs dynamischer als die Interphase-MTs37,38. In ähnlicher Weise wurden Unterschiede in der Wachstumsrate und Schrumpfung sogar innerhalb derselben Zellzyklusphase beobachtet, wie Mitose39,40. Um eine falsche Datenerfassung zu vermeiden, sollte die Messung der MT-Dynamik daher während des Zellzyklus auf ein enges Zeitfenster beschränkt werden. Zum Beispiel kann die Messung der MT-Dynamik in der Prometaphase durch die Behandlung der Zellen mit Dimethylenastron (DME) erreicht werden, einem monastrolen Analogon, das die Motorkinus eg541 hemmt und die Bildung der bipolaren Mitotischen Spindel42verhindert. Die Hemmung von Zellen in der Prometaphase mit Eg5-Hemmer DME und anderen monastrolen Derivaten hat keinen Einfluss auf die MT-Dynamik43,44,45, was DME zu einem nützlichen Werkzeug für die Untersuchung der MT-Dynamik sowohl in festen als auch in lebenden Zellen macht44.
Hier kombinieren wir die von Ertych etal. 44 beschriebene Methode der MT-Dynamikanalyse in Prometaphasenzellen mit dualspinnierender Scheibenbildgebung. Diese Methode ermöglicht die Messung der MT-Dynamik in Prometaphasenzellen, die von einer einzigen Fokalebene mit einer höheren Bildrate, jedoch ohne Photobleichung und minimale Phototoxizität gesammelt wurden. Darüber hinaus verwenden wir als fluoreszierender Reporter Tandem-Dimer Tomato fluorescent protein (tdTomato), das die Helligkeit und Photostabilität im Vergleich zum grünen Fluoreszenzprotein (EGFP) verbessert hat und mit weniger Energielicht46angeregt wird. Daher benötigt tdTomato weniger Laserleistung für die Anregung und ist weniger fototoxisch. Insgesamt verbessern wir die Methode weiter, indem wir die Phototoxizität reduzieren und die Auflösung und Nachbearbeitung verbessern, die für die MT-Dynamikanalyse erforderlich sind. Darüber hinaus schaffen wir eine Grundlage für zukünftige Änderungen der Methode, indem wir sie mit anderen Synchronisationstechniken kombinieren.
Access restricted. Please log in or start a trial to view this content.
Protokoll
1. Aussaat von HeLa-Zellen
- Bereiten Sie 2 ml mit 5 g/ml Fibronectin-Lösung in Phosphatgepufferter Saline (PBS) vor und fügen Sie 450 l davon in jeden Brunnen eines 4 gut gekammerten Deckellips (#1.5) ein. Inkubieren Sie den Schlitten 15 min bei 37 °C und 5%CO2.
- Asynchron wachsende HeLa-Zellen mit Dulbeccos Phosphatgepufferte Saline (DPBS) abspülen und mit Trypsin-EDTA (0,05%: 0,02%; w:v) 5 min bei 37 °C inkubieren. Stoppen Sie die enzymatische Reaktion durch die Zugabe von Roswell Park Memorial Institute (RPMI) 1640 Medium ergänzt mit 10% hitzeinaktiviertem fetalem Kalbsserum (FCS) bei 3:1 (v:v) Verhältnis von hinzugefügten Trypsin-EDTA.
HINWEIS: HeLa-Zellen wurden in RPMI 1640 medium gehalten, ergänzt mit 10% wärmeinaktiviertem FCS bei 37 °C und 5%CO2 und wurden routinemäßig durchzogen, sobald sie wie oben beschrieben eine Konfluenz von 80–90% erreichten. - Bestimmen Sie die Zellkonzentration mit einer Neubauer-Kammer. Mischen Sie ein 50 L Aliquot der Zellsuspension mit Trypan blau bei 1:1 (v:v) Verhältnis, resuspendieren, und übertragen Sie 10 l der Suspension in die Kammer. Zählen Sie nur die Trypan blau-negativen Zellen innerhalb der vier großen Quadrate (Details siehe Phelan et al.47). Leiten Sie die Zellkonzentration von der gezählten Zellzahl mit der folgenden Formel ab:
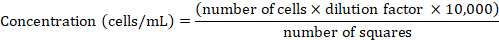
- Pellet die Zellen durch Zentrifugation bei 300 x g für 2 min. Resuspend mit frischem RPMI 1640, um 1 x 106 Zellen/ml zu erhalten.
- Entfernen Sie das Fibronectin aus dem kammerierten Deckel, waschen Sie die Brunnen zweimal mit DPBS und säen Sie 50.000 Zellen pro Brunnen.
- Den kammerierten Deckelrutsch mit den Zellen in den Inkubator zurückgeben und 24 h bei 37 °C und 5%CO2anbauen.
2. Expression von pEB3-tdTomato in HeLa-Zellen
- Bereiten Sie ein 1,5 ml Mikrozentrifugenrohr vor. Für jedes Rohr 2 g pEB3-tdTomato48 mit Transfektionspuffer (synthetisches Produkt in wässriger Lösung) auf ein Endvolumen von 396 l verdünnen.
- Fügen Sie dem ersten Röhrchen 4 l Transfektionsreagenz (nicht-lipidisch, polyethylenimin enthaltend) hinzu und wirbeln Sie die Mischung sofort für genau 10 s aus.
- Drehen Sie das Rohr mit einer Mikrozentrifuge kurz herunter und brüten bei Raumtemperatur (RT) 10 min.
- Entfernen Sie die HeLa-Zellen aus dem Inkubator. Tropfend fügen Sie jedem Brunnen eines 4 gut kammerigen Deckels 100 l des Transfektionsgemisches hinzu und geben Sie die Zellen an den Inkubator zurück.
- Nach 4 h Inkubation bei 37 °C und 5%CO2die Zellen mit frischem Wachstumsmedium ergänzen und mindestens 24 h bei 37 °C und 5%CO2brüten.
HINWEIS: Es ist notwendig, die Transfektionsbedingungen für jeden Zelltyp zu optimieren. Die Ausdrucksebenen müssen niedrig genug sein, um die Identifizierung einzelner MT-Wachstumsenden zu ermöglichen. Alternativ kann in den Experimenten eine Zelllinie verwendet werden, die EB3-tdTomato stabil ausdrückt; dies würde die Variabilität der Expressionsniveaus von EB3-tdTomato zwischen Präparaten und zwischen Zellen aus der gleichen Zubereitung verringern49.
3. Synchronisation und Live-Zell-Bildgebung von pEB3-tdTomato-expressing HeLa Cells
- Bereiten Sie eine 2,5-M-Lösung aus Dimethylenastron (DME) in phenolrot-freiem Dulbecco es Modified Eagle Medium (DMEM) vor, ergänzt durch 10% FCS und 2 mM L-Glutamin oder eine alternative Glutaminversorgung.
- Ersetzen Sie das Wachstumsmedium im kammerierten Deckelschlupf durch 500 l des Wachstumsmediums, das 2,5 M DME enthält, und inkubieren Sie die Zellen bei 37 °C und 5%CO2.
- Nach 3,5 h Inkubation mit DME die Zellen auf das Mikroskop übertragen, den kammerierten Abdeckungsslip in eine Umweltkammer mit dunklen Paneelen für die Bildgebung bei 37 °C und 5%CO2montieren und weiter inkubieren, bis die gesamte Inkubationszeit 4 h beträgt.
HINWEIS: Die Aufrechterhaltung der Temperatur bei 37 °C ohne Schwankungen ist für das Experiment entscheidend. - Führen Sie die Zeitraffer-Bildgebung auf einem invertierten Mikroskop durch, das mit einem 100x 1,49 N.A. Öl-Tauchobjektiv, einem Konfokalsystem für Zweispinnscheiben und einem zuverlässigen Autofokussystem für die kontinuierliche Wartung der Brennebene ausgestattet ist. Definieren Sie die Bildparameter wie folgt.
HINWEIS: Wir verwenden eine Elektronenmultiplizierende Charge-Coupled Device Kamera (EM-CCD).- Für EB3-tdTomato Anregung, verwenden Sie eine 561 nm Laserlinie mit 200 ms Belichtungszeit. Sammeln Sie das emittierte Licht durch einen vierfachen Bandpass (405, 488, 561, 640 nm) dichroitischen Spiegel und einen 600/52 nm Emissionsfilter.
HINWEIS: Die Laserleistung kann für jede abgebildete Zelle eingestellt werden, um eine Bildsättigung zu verhindern. In allen hier angegebenen Zeitrafferfilmen wurde die Laserleistung auf 5,3 mW eingestellt. - Finden Sie eine Zelle in Prophase und Fokus in der Z-Ebene, die dem Zentrum der monopolaren mitotischen Spindel entspricht. Erfassen Sie Bilder alle 0,5 s über insgesamt 1 min ohne Binning und ohne Beleuchtung zwischen den Belichtungen.
- Für EB3-tdTomato Anregung, verwenden Sie eine 561 nm Laserlinie mit 200 ms Belichtungszeit. Sammeln Sie das emittierte Licht durch einen vierfachen Bandpass (405, 488, 561, 640 nm) dichroitischen Spiegel und einen 600/52 nm Emissionsfilter.
4. Analyse der MT-Dynamik mit U-Track v2.2.0
- Zur Analyse der MT-Dynamik ist eine numerische Computerumgebungssoftware erforderlich (z.B. MATLAB).
HINWEIS: Grundlegendes Verständnis der Software reicht für die Analyse aus. Umfassendes Hilfematerial und Tutorials finden Sie auf der Website des Entwicklers (https://uk.mathworks.com/products/matlab/getting-started.html). - Laden Sie (https://github.com/DanuserLab/u-track) herunter und installieren Sie die Open-Source-Software U-Track v2.2.0 gemäß den detaillierten Anweisungen in der Datei "Readme_u-track.pdf"50,51,52.
- Starten Sie die numerische Analysesoftware und fügen Sie dem Softwaresuchpfad den Ordner U-Track v2.2.0 mit Unterordnern hinzu.
- Rufen Sie im Befehlsfenster "movieSelectorGUI" auf. Dadurch wird ein Dialogfenster geöffnet, aus dem die von der Bildaufnahmesoftware am Mikroskop erzeugten Rohdateien importiert werden können (Zusatzabbildung 1, Abbildung 2, Abbildung 3, Abbildung 4).
HINWEIS: Die U-Track Software ist mit anderen Bilddatenformaten kompatibel. Es verwendet Bio-Formate, die verschiedene Life Science Datenformate erkennen53. - Die Größe jedes Bildes wird automatisch aus den Metadaten gelesen. Geben Sie manuell die numerische Blende des Objektivs (in diesem Fall 1,49) und des Zeitintervalls (0,5 s) ein, das für die Bildgebung verwendet wird (ergänzende Abbildung 1B). Zusätzlich können Informationen über die Anregungswellenlänge, das Fluorophor und die Belichtungszeit bereitgestellt werden, aber sie sind für die weitere Analyse nicht entscheidend.
- Sobald alle Bilder geladen sind, speichern Sie die eingegebene Zeitrafferserie als Filmliste, indem Sie die "Speichern als Filmliste "auswählen. Wählen Sie auf der rechten Seite des Dialogfensters die Option "U-Track" und drücken Sie "Continue" (Supplementary Figure 1C).
HINWEIS: Die Werte sind für HeLa-Zellen optimiert. Wenn Sie zu einer anderen Zelllinie wechseln, sollten die Werte erneut definiert werden. Alternativ können Sie die von den Softwareentwicklern empfohlenen Einstellungen verwenden. Die detaillierte Erläuterung der einzelnen Parameter und deren Definition finden Sie im technischen Bericht mit der vorherigen Version der Software, plusTipTracker50. - Wählen Sie im Pop-up-Fenster "Microtubule Plus-Ends" und drücken Sie "Ok" (Ergänzende Abbildung 1C). Das neue Dialogfenster ermöglicht die Bestimmung der Parameter für die drei Schritte der Analyse (Ergänzende Abbildung 1D), die Erkennung, Nachverfolgung und Spuranalyse sind.
- Wählen Sie in Schritt 1 "Einstellungen" und in einem Dropdown-Menü "Kometenerkennung" als Erkennungsmethode aus (Zusatzabbildung 2B).
- Aus dem neuen Dialogfenster definieren Sie die Parameter für die Differenz des Gaußer Filters und die Einzugsgebietsegmentierung wie folgt (Zusatzabbildung 2C): Maskenprozess für die Erkennung = Keine; Niedrigpass Gaußsche Standardabweichung = 1 Pixel; Hochpass Gaußsche Standardabweichung = 3 Pixel; Mindestschwelle = 3 Standardabweichungen; Schwellenwertschrittgröße = 0,25 Standardabweichungen. Wählen Sie "Einstellungen auf alle Filmeanwenden " und "Anwenden".
- In Schritt 2 werden die Parameter zum Verknüpfen, Schließen von Spalt, Zusammenführen und Aufteilen und Kalman-Filterfunktionen in drei Schritten definiert, wie in Rosa, Grün und Blau hervorgehoben , entsprechend(Ergänzende Abbildung 3B). Wählen Sie für diese Schritte die Option "Microtubule Plus-End Dynamics" aus, und definieren Sie aus der Option "Einstellen" die Werte, wie in der Zusatzabbildung 3C–Eangegeben.
- Bei Problemen mit der Dimensionalität wählen Sie "2" aus dem Dropdown-Menü. Verwenden Sie Maximale Lücke zum Schließen = 5 Frames; Minimale Länge der Spursegmente aus dem ersten Schritt = 3 Frames. Wählen Sie wie bisher "Einstellungen auf alle Filmeanwenden " und klicken Sie auf "Anwenden".
- In Schritt 3 der Analyse werden die erkannten MT-Spuren klassifiziert (Ergänzende Abbildung 4). Wählen Sie als Trackanalysemethode "Microtubule Dynamics Classification" und definieren Sie die Parameter über die Schaltfläche "Einstellen", wie in Der Ergänzenden Abbildung 4B,Cangegeben. Danach wählen Sie das Feld "Einstellungen auf alle Filme anwenden" und klicken Sie auf "Anwenden".
- Sobald alle Parameter definiert sind, wählen Sie im Fenster "Systemsteuerung –U-Track" (Ergänzende Abbildung 1D) die Felder "Check/Uncheck to All Moviesanwenden " und "Alle Filme ausführen" drücken und drücken Sie "Ausführen". Damit wird die MT-Analyse der Zeitrafferserie eingeleitet.
- Sobald die Filmverarbeitung abgeschlossen ist, wird die Meldung "Ihre Filme wurden erfolgreich verarbeitet" angezeigt. Drücken Sie "Ok", dann "Save".
- Jetzt ist es sicher, die numerische Analyse-Software zu beenden. Die Ergebnisse der Filmverarbeitung werden in Unterordnerstrukturen als m-Dateien in dem Ordner gespeichert, in dem die Rohdateien gespeichert sind.
5. Statistische Analyse der MT-Dynamik
- Importieren Sie die m-Dateien in ein bevorzugtes Statistikanalyseprogramm.
HINWEIS: In unserem Fall importieren wir zuerst die Dateien in eine Standardtabelle, um sie lesbar zu machen. Die m-Dateien enthalten statistische Informationen (Median, Mittelwert und Standardabweichung) zu verschiedenen Parametern (z.B. Wachstumsgeschwindigkeit, MT-Dynamik). Die detaillierte Liste der Parameter ist im technischen Bericht mit der vorherigen Version der Software, plusTipTracker50,52angegeben. Die generierten m-Dateien können auch in andere Datenverarbeitungssoftware importiert werden. - Wählen Sie den Parameter "Wachstumsgeschwindigkeit bedeutet" und importieren Sie ihn in eine Tabelle für Statistiken und Anzeige. Geben Sie die Informationen zu anderen Parametern (z. B. "Dynamik") entweder in einer neuen Tabelle oder in einer neuen Spalte derselben gruppierten Tabelle und demselben Diagramm ein.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Nach dem in Abbildung 1Abeschriebenen Protokoll wurde das pEB3-tdTomato-Plasmid vorübergehend in asynchron wachsenden HeLa-Zellen exprimiert. Die Zellen wurden 48 h nach der Transfektion bei Prometaphase durch DME-Behandlung synchronisiert (Abbildung 1B). Dieser Schritt sorgte dafür, dass die Messung der MT-Dynamik immer in der gleichen Phase des Zellzyklus durchgeführt wurde. Die Zeitrafferfilme wurden mit U-Track v2.2.0, wie in der Ergänzenden Dokumentatio...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Hier beschreiben wir eine Modifikation einer Methode, die zuerst von Ertych et al.44etabliert wurde. Zusammen mit einigen anderen Modifikationen kombinieren wir diese Technik der MT-Dynamikanalyse mit der konfokalen Bildgebung der Doppelspinnscheibe. Die Verwendung der Doppelspinnscheibe verbessert die Auflösung wachsender MTs und reduziert gleichzeitig die Phototoxizität36. Wir reduzieren die Photobleaching- und Laserlichtschäden der Zellen weiter, indem wir zu einem fl...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Wir danken den Mitgliedern der Lichtmikroskopieanlage, dem Max-Planck-Institut für Experimentelle Medizin, für die fachkundige Beratung und Unterstützung.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
Referenzen
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten