このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
単極有糸状スピンドルにおけるスピンディスク顕微鏡による微小管ダイナミクスの測定
要約
ここでは、生細胞紡糸ディスク共焦点顕微鏡法とMATLABベースの画像処理を用いてプロメタフェイズで同期した細胞における微小管ダイナミクス解析の堅牢かつ詳細な方法を提示する。
要約
生細胞における微小管ダイナミクスを決定するための確立された方法の改変について説明する。このプロトコルは、微小管(TdTomato蛍光タンパク質で標識されたEB3)の陽性端に対する遺伝的にコードされたマーカーの発現と、紡糸ディスク共焦点顕微鏡を用いた高速、高解像度、生細胞イメージングに基づいている。細胞周期同期と微小管の密度の増加は、有糸分裂細胞における遠心分離を阻害することによって達成され、そして成長の分析はオープンソースのU-Trackソフトウェアを用いて行われる。明るく赤ずれた蛍光タンパク質を使用すると、レーザーパワーが低く、スピンディスク顕微鏡検査に必要な露光時間が短縮され、光毒性や光誘起アーティファクトの確率が低下します。これにより、標準的な培養条件下で増殖培地中の細胞を維持しながら、同じ調製物でより多くの細胞をイメージングすることができます。解析は教師付き自動で実行されるため、結果は統計的に堅牢で再現可能です。
概要
微小管(MT)は、事実上すべての真核細胞およびいくつかの細菌1に見られる非常に動的な構造である。アクチンおよび中間フィラメントと共に、それらは細胞骨格2、3を彫刻する。細胞分裂4、分子輸送5、フラッラー打ち負かす6、原発性シリウム7を介した周囲環境の感覚、聴覚(キノチリウム)8、9、胚発生10、11、12、浸潤および転移13、14、さらには記憶形成15、16、17、そして、他の多くのプロセスは、主にMTに依存しています。このプロパティは、動的不安定性 19 として記述されます。MTの動的性は、多くの病理学的条件20、21、22で変化する。したがって、この特性の性質を決定することは、疾患のメカニズムとその後の治療を理解するのに役立ちます。
MTダイナミクス解析のための方法の長いリストが開発されており、そのほとんどはイメージング技術23に基づいています。当初、広視野光顕微鏡は、インビトロ24におけるチューブリンポリマーの形成を観察するために使用された。MTプラスエンドで集まるエンド結合(EB)タンパク質の発見と、タンパク質を蛍光標識する方法の開発により、広視野および共焦点蛍光顕微鏡25、26、27を有する生細胞におけるMTの挙動を直接観察することが可能となった。1つのEBタンパク質は、エンド結合タンパク質3(EB3)28である。蛍光タンパク質に融合したEB3を過剰発現および追跡することにより、MTプラスエンド組み立て率は29、30を決定することができる。
共焦点レーザー走査蛍光顕微鏡(CLSM)は、MTダイナミクスに従うために頻繁に使用されます。しかしながら、この撮像技術は、光毒性および光脱色のリスクが高く、生細胞および薄暗い試料イメージング31に対する2つの望ましくないプロセスを有する。より良い信号対雑音比を得るためには、レーザーパワーと露光時間はサンプルにダメージを与えない状態で十分に高くする必要があり、速度と引き換えに解像度を犠牲にする必要があります。CLSMに代わる適切な代替手段は、回転ディスク顕微鏡32である。このイメージングモダリティは、ピンホールの配列を有する移動ディスクからなるニプコウディスク33の使用に基づいており、同じサンプルを同時に34個撮像する多くのCLS顕微鏡と同等に動作する。したがって、レーザーからの光は、同時にサンプル内のいくつかの領域を照らすが、共焦点性を保持します。したがって、Nipkowディスクは、CLSMに似た画像を取得できますが、より高速で、より少ないレーザーパワーを使用することができます。ニプコウディスクは、個別に光をそれぞれのピンホールに導くマイクロレンズの配列を持つ第2のディスクを導入した横川電機によってさらに改良され、光毒性と光脱色35をさらに低減しました。このように、紡糸ディスクレーザー走査顕微鏡は生細胞イメージングの選択肢となり、高速31、36で高い信号対雑音比の画像を得ることが可能となり、急速に成長するMT末端からの信号等の解決に不可欠である。
MT ダイナミクスは一時的に異なります。たとえば、有糸分裂性の M は、相間のM7、38よりも動的です。同様に、有糸分裂39、40などの同じ細胞周期期内においても増殖速度および収縮の違いが観察されている。したがって、誤ったデータ収集を避けるために、MTダイナミクスの測定は、細胞周期中の狭い時間枠に制限する必要があります。例えば、プロメタフェイズにおけるMTダイナミクスの測定は、モータキネシンEg541を阻害し、バイポーラ有糸状スピンドル42の形成を防止するモナストロル類似体であるジメチルエナストロン(DME)で細胞を処理することによって達成することができる。Eg5阻害剤DMEおよび他のモナストロル誘導体を有するプロメタフェイズにおける細胞の阻害は、MTダイナミクス43、44、45に影響を及ぼさない、これはDMEを固定細胞および生細胞44の両方でMTダイナミクスを研究するための有用なツールとなる。
ここでは、Ertych et al.44で説明したプロメタフェイズ細胞におけるMTダイナミクス解析の方法とデュアルスピニングディスクイメージングを組み合わせる。この方法は、光漂白および最小限の光毒性なしで、より高いイメージング速度を有する単一の焦点面から収集されたプロメタフェイズ細胞におけるMTダイナミクスの測定を可能にする。さらに、蛍光レポーターとして、緑色蛍光タンパク質(EGFP)に比べて明るさや光安定性が向上し、エネルギー光46を低くして励起されるタンデムダイマートマト蛍光タンパク質(tdTomato)を用いた。したがって、tdTomatoは励起のためのより少ないレーザーパワーを必要とし、より少ない光毒性です。全体として、光毒性を低減し、MTダイナミクス解析に必要な分解能と後処理を改善することで、この方法をさらに改善します。さらに、メソッドを他の同期手法と組み合わせることで、メソッドを将来変更するための基礎を作成します。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. HeLa細胞の播種
- リン酸緩衝生理食塩水(PBS)に5μg/mLフィブロネクチン溶液の2mLを調製し、4ウェルチャンバーカバースリップ(#1.5)の各ウェルに450 μLを加えます。スライドを37°Cで15分間インキュベートし、5%CO2.
- リンスは、ダルベッコのリン酸緩衝生理食塩酸(DPBS)でHeLa細胞を非同期的に増殖させ、トリプシンEDTA(0.05%:0.02%;w:v)を37°Cで5分間インキュベートします。ロズウェルパーク記念研究所(RPMI)1640培地を添加して、10%熱不活性化胎児胎児血清(FCS)を添加したトリプシン-EDTAの比率(v:v)で添加することにより酵素反応を停止します。
注:HeLa細胞は、37°Cおよび5%CO2で10%熱不活性化FCSを添加したRPMI 1640培地中に維持され、上述したように80〜90%の結合に達すると日常的に通過した。 - ノイバウアーチャンバーを使用して細胞濃度を決定します。セル懸濁液の50μLアリコートをトリパンブルーで1:1(v:v)比で混合し、再サスペンドし、懸濁液の10°Lをチャンバーに移します。4つの大きな正方形の中のトリパン青陰性細胞のみを数えます(詳細はPhelan et al.47を参照)。次の式を使用して、カウントされたセル番号からセル濃度を導出します。
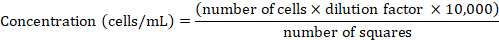
- 細胞を300 x gで2分間遠心分離してペレット化し、1 x 106セル/mLを得るために新鮮なRPMI 1640で再サスペンドします。
- チャンバー付きカバースリップからフィブロネクチンを取り出し、DPBSでウェルを2回洗浄し、井戸あたり50,000細胞を種子します。
- 細胞と一緒にチャンバ入りのカバースリップをインキュベーターに戻し、37°Cと5%CO2で24時間成長させます。
2. HeLa細胞におけるpEB3-tdTomatoの発現
- 1.5 mLマイクロ遠心管を準備します。チューブごとに、トランスフェクションバッファ(水溶液中の合成物)でpEB3-tdTomato48を2μg希釈し、最終体積396μLにします。
- トランスフェクション試薬(非脂質、ポリエチレニミンを含む)を第1チューブに4μL添加し、直ちに10sのボルテックスを加える。
- マイクロ遠心分離機でチューブを短時間スピンダウンし、室温(RT)で10分間インキュベートします。
- インキュベーターから HeLa 細胞を取り除きます。ドロップワイズ、トランスフェクション混合物の100μLを4ウェルチャンバーカバースリップの各ウェルに加え、細胞をインキュベーターに戻す。
- 37°Cおよび5%CO2で4時間インキュベートした後、細胞を新鮮な増殖培地で補い、37°Cと5%CO2で少なくとも24時間インキュベートする。
注:細胞タイプごとにトランスフェクション条件を最適化する必要があります。式レベルは、単一の MT 成長端の識別を可能にするのに十分な低さである必要があります。あるいは、EB3-tdTomatoを安定して発現する細胞株を実験に使用することもできる。これは、同じ調製物49からの調製物間および細胞間のEB3-tdTomatoの発現レベルの変動性を低下させるであろう。
3. pEB3-tdTomato発現HeLa細胞の同期と生細胞イメージング
- フェノールレッドフリーダルベッコの修飾イーグル培地(DMEM)にジメチルエナストロン(DME)の2.5μM溶液を調製し、10%FCSおよび2mM L-グルタミンまたは代替グルタミン供給を補充します。
- チャンバ入りカバースリップの成長培地を2.5μM DMEを含む成長培地の500°Lに交換し、細胞を37°Cおよび5%CO2でインキュベートします。
- DMEによるインキュベーションを3.5時間行った後、細胞を顕微鏡に移し、チャンバカバースリップを37°Cおよび5%CO2で撮像するためのダークパネルを備えた環境室に取り付け、さらに全インキュベーション時間が4時間になるまでインキュベートする。
注:変動のない37°Cでの温度維持は、実験に不可欠です。 - 100x 1.49 N.A. オイル浸漬目的、二重紡糸ディスク共焦点システム、および焦点面の継続的なメンテナンスのための信頼性の高いオートフォーカスシステムを搭載した反転顕微鏡でタイムラプスイメージングを実行します。イメージング パラメータを次のように定義します。
注:電子乗算電荷結合デバイスカメラ(EM-CCD)を使用しています。- EB3-tdTomato励起の場合は、200ミリ秒の露光時間を持つ561 nmレーザーラインを使用します。4倍のバンドパス(405、488、561、640 nm)の二色ミラーと600/52 nmの発光フィルタを介して放出された光を収集します。
注:レーザーパワーは、画像の飽和を防ぐために、画像化されたセルごとに調整することができます。ここで与えられたすべてのタイムラプス映画では、レーザーパワーは5.3 mWに設定されました。 - プロフェーズの細胞を見つけ、単極有糸状スピンドルの中心に対応するZ平面に焦点を合わせます。ビニングなし、露出間の照明なしの合計1分にわたって0.5 sごとに画像を取得します。
- EB3-tdTomato励起の場合は、200ミリ秒の露光時間を持つ561 nmレーザーラインを使用します。4倍のバンドパス(405、488、561、640 nm)の二色ミラーと600/52 nmの発光フィルタを介して放出された光を収集します。
4. Uトラックv2.2.0を用いたMTダイナミクスの解析
- MTダイナミクスを解析するには、数値計算環境ソフトウェア(例えばMATLAB)が必要である。
注:ソフトウェアの基本的な理解は、分析のために十分です。包括的なヘルプ資料とチュートリアルは、開発者の Web サイト (https://uk.mathworks.com/products/matlab/getting-started.html) で入手できます。 - 「https://github.com/DanuserLab/u-track Readme_u-track.pdf」ファイル50、51、52に記載されている詳細な指示に従って、オープンソースのU-Track v2.2.0ソフトウェアをダウンロードしてインストールします。
- 数値解析ソフトウェアを起動し、サブフォルダを含む U-Track v2.2.0 フォルダをソフトウェア検索パスに追加します。
- コマンド ウィンドウから "movieSelectorGUI" を呼び出します。これにより、顕微鏡で画像取得ソフトウェアによって生成された生ファイルをインポートできるダイアログ ウィンドウが開きます (補足図 1、図 2、図 3、図 4)。
注:Uトラックソフトウェアは、他の画像データ形式と互換性があります。これは、異なるライフサイエンスデータ形式53を認識するバイオフォーマットを使用しています。 - 各イメージのサイズは、メタデータから自動的に読み取られます。目的の開口数 (この場合は 1.49) とイメージングに使用する時間間隔 (0.5 秒) を手動で入力します (補足図 1B)。さらに、励起波長、蛍光色素、露光時間に関する情報も提供できますが、さらなる分析には重要ではありません。
- すべての画像が読み込まれたら、「ムービーリストとして保存」を選択して、入力したタイムラプスシリーズをムービーリストとして保存します。ダイアログ ウィンドウの右側にある[U-Track]オプションを選択し、[続行]をクリックします (補足図 1C)。
注:値は HeLa セル用に最適化されます。別のセルラインに切り替える場合は、値を再度定義する必要があります。または、ソフトウェア開発者が推奨する設定を使用します。各パラメータとその定義方法の詳細な説明は、以前のバージョンのソフトウェアに付属の技術レポート plusTipTracker50にあります。 - ポップアップ ウィンドウで [マイクロチューブプラスエンド]を選択し、[ >OK」(補足図 1C) を押します。新しいダイアログ ウィンドウでは、検出、追跡、および追跡分析である分析の 3 つのステップ (補足図 1D)のパラメーターを決定できます。
- 手順 1 で [設定] を選択し、ドロップダウン メニューから検出方法として [彗星検出] を選択します (補足図 2B)。
- 新しいダイアログ ウィンドウから、ガウス フィルタと集水域セグメンテーションの違いのパラメータを次のように定義します (補足図 2C):検出に使用するマスク プロセス = なし;低パスガウス標準偏差 = 1ピクセル。ハイパスガウス標準偏差 = 3ピクセル。最小しきい値 = 3 標準偏差。しきい値ステップサイズ = 0.25 標準偏差。[すべてのムービーに設定を適用する]を選択し、[適用]を選択します。
- 手順 2 では、リンク、ギャップの終了、マージと分割、および Kalman フィルタ関数のパラメータは、ピンク、緑、青で強調表示された 3 つのステップで定義されます (補足図 3B)。これらの手順では、「マイクロチューブ・プラスエンド・ダイナミクス」を選択し、「設定」オプションから、補足図 3C-Eに示すように値を定義します。
- 次元に関する問題については、ドロップダウンメニューから「2」を選択します。[最大ギャップ] を使用して閉じる = 5 フレーム。最初のステップからのトラック セグメントの最小長 = 3 フレーム。前と同様に、[すべてのムービーに設定を適用]を選択し、[適用]をクリックします。
- 分析のステップ3では、検出されたMTトラックが分類される(補足図4)。トラック分析方法として、「微小管ダイナミクス分類」を選択し、補足図 4B,Cに示すように「設定」ボタンを使用してパラメータを定義します。その後、[すべてのムービーに設定を適用]ボックスを選択し、[適用]をクリックします。
- すべてのパラメータを定義したら、[コントロール パネル-U-Track]ウィンドウ (補足図 1D)で [すべてのムービーにチェック/チェックを適用]チェック ボックスをオンにし、[すべてのムービーを実行]ボックスをオンにして[実行]をクリックします。これにより、タイムラプスシリーズのMT解析が開始されます。
- ムービーの処理が完了すると、「ムービーが正常に処理されました」というメッセージが表示されます。「OK」を押してから、「保存」を押します。
- 今、数値解析ソフトウェアを終了しても安全です。ムービー処理の結果は、未加工ファイルが格納されているフォルダ内の m ファイルとしてサブフォルダ構造に格納されます。
MTダイナミクスの統計解析
- m ファイルを優先統計分析プログラムにインポートします。
注:この例では、まず標準スプレッドシートにファイルをインポートして読みやすくします。Mファイルには、異なるパラメータ(成長率、MTダイナミクスなど)の統計情報(中央値、平均、標準偏差)が含まれます。パラメータの詳細なリストは、以前のバージョンのソフトウェアで提供された技術レポートに記載されています。生成されたMファイルは、他のデータ処理ソフトウェアにインポートすることもできます。 - 「成長速度平均」パラメータを選択し、統計および表示用のテーブルにインポートします。新しいテーブルまたは同じグループ化されたテーブルとプロットの新しい列に、他のパラメータ("動的性"など)に関する情報を入力します。
Access restricted. Please log in or start a trial to view this content.
結果
図1Aに概説された所定のプロトコルに続いて、pEB3-tdTomatoプラスミドは、非同期増殖HeLa細胞で一過性に発現した。細胞をDME処理を介してプロメタフェイズでトランスフェクションした後48時間同期させた(図1B)。このステップにより、MTダイナミクスの測定は常に細胞周期の同じ段階で行われました。タイムラプスムービーは、補足文書5...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
ここでは、Ertychら44によって最初に確立された方法の改変について説明する。他のいくつかの変更と共に、MTダイナミクス解析のこの技術とデュアルスピニングディスク共焦点イメージングを組み合わせます。デュアルスピニングディスクの使用は、光毒性36を低減しながら、成長するMTの解像度を向上させます。さらに、より長波長の蛍光レポーターに切?...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者たちは何も開示する必要はない。
謝辞
私たちは、彼らの専門家のアドバイスとサポートのために、光顕微鏡施設、マックスプランク実験医学研究所のメンバーに感謝します。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
参考文献
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved