Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Измерение динамики микротрубок с помощью вращающейся микроскопии диска в монополярных митотическом спинное
В этой статье
Резюме
Здесь мы представляем надежный и подробный метод анализа микротрубной динамики в клетках, синхронизированных в прометафазе с помощью живой клетки вращающейся дисковой конфокальной микроскопии и обработки изображений на основе MATLAB.
Аннотация
Мы описываем модификацию установленного метода определения динамики микротрубв в живых клетках. Протокол основан на экспрессии генетически закодированного маркера для положительных концов микротрубок (EB3, маркированных флуоресцентным белком tdTomato) и высокоскоростной, с высоким разрешением, живоклеточной визуализации с помощью вращающейся дисковой конфокальной микроскопии. Синхронизация клеточного цикла и увеличение плотности микротрубок достигается путем ингибирования центросомального разделения в митотических клетках, а анализ роста проводится с помощью программного обеспечения U-Track с открытым исходным кодом. Использование яркого и красного сдвига флуоресцентного белка в сочетании с более низкой мощностью лазера и сокращением времени экспозиции, необходимого для вращающейся дисковой микроскопии, снижает фототоксичность и вероятность световых артефактов. Это позволяет для визуализации большего количества клеток в том же препарате при сохранении клеток в среде роста в стандартных условиях культуры. Поскольку анализ выполняется контролируемым автоматическим способом, результаты являются статистически надежными и воспроизводимыми.
Введение
Микротрубочки (МТ) являются высокодинамическими структурами, встречаемыми практически во всех эукариотических клетках и в некоторых бактериях1. Вместе с актином и промежуточными нитями они лепят цитоскелет2,3. Ячейка разделение4, молекула транспорт5, flagellar избиение6, ощущение окружающей среды через первичный цилиум7, слух (киноцилиум)8,9, эмбриогенез10,11,12, вторжение и метастазы13,14, и даже формирование памяти15,16,17,18, и многие другие процессы в первую очередь опираются на МТ. Участие МТ во всех этих событиях было бы невозможно без их замечательной способности быстро переключаться между ростом (полимеризация) и усадки (деполимеризация). Это свойство описывается как динамическая нестабильность19. Mt динамика изменяется во многих патологических условиях20,21,22. Таким образом, определение характера этого свойства может помочь понять механизмы болезни, а затем их лечение.
Для анализа динамики МТ был разработан длинный список методов, большинство из которых основаны на методах визуализации23. Первоначально для наблюдения за образованием полимеров тубулина в пробирке24использовались широкоугольные световые микроскопы. Открытие конечных связывания (EB)-белки, которые собирают на МТ плюс-концы и развитие методов флуоресцентно этикетки белков позволило наблюдать за поведением MTs непосредственно в живых клетках с широким полем и конфокальные флуоресценции микроскопов25,26,27. Один EB-белок является конечным связывающим белком 3 (EB3)28; путем переэкспрессинга и отслеживания EB3, слитого в флуоресцентный белок, MT плюс конец сборки ставки могут быть определены29,30.
Конфокальная лазерная сканирующая флуоресцентная микроскопия (CLSM) часто используется для наблюдения за динамикой МТ. Тем не менее, этот метод визуализации представляет высокий риск фототоксичности и фотоотбеления, два нежелательных процессов для живых клеток и тусклый образец изображения31. Для того, чтобы получить лучшее соотношение сигнала к шуму, мощность лазера и продолжительность экспозиции должны быть достаточно высокими, не повреждая образцы, и это требует ущерба разрешением в обмен на скорость. Подходящей альтернативой CLSM является спиннинг дискмикроскопия32. Эта модальность изображения основана на использовании диска Nipkow33, который состоит из движущегося диска, несущего массив пинхолов, и работает эквивалентно многим микроскопам CLS, изображая тот же образец одновременно34. Таким образом, свет от лазера будет освещать несколько областей в образце одновременно, но сохранить конфокальный характер. Таким образом, диск Nipkow позволяет получать изображения, похожие на CLSM, но быстрее и используя меньше лазерной энергии. Диск Nipkow был дополнительно улучшен Yokogawa Electric, который представил второй диск с массивом микролинз на нем, что индивидуально направлять свет в соответствующую пинхол, дальнейшее снижение фототоксичности и photobleaching35. Таким образом, вращающийся диск лазерное сканирование микроскопии стал методом выбора для живой визуализации клеток, и это позволяет получать изображения с высоким соотношением сигнала к шуму на высокой скорости31,36, что имеет решающее значение для решения сигналов, таких как от быстрорастущих концов МТ.
Динамика МТ временно отличается. Например, митотические МТ более динамичны, чем межфазные37,38. Аналогичным образом, различия в темпах роста и усадке наблюдались даже в пределах одной и той же фазы клеточного цикла, например, митоза39,40. Поэтому, чтобы избежать ложного сбора данных, измерение динамики МТ должно ограничиваться узким временем во время клеточного цикла. Например, измерение динамики МТ в прометафазе может быть достигнуто путем лечения клеток с диметиленастроном (DME), монострольным аналогом, который подавляет моторный кинезин Eg541 и предотвращает образование биполярного митоtic шпинделя42. Ингибирование клеток на прометафазе с ингибитором Eg5 DME и другими производными монастрола не влияет на динамику МТ43,44,45, что делает DME полезным инструментом для изучения динамики МТ как в стационарных, так и в живых клетках44.
Здесь мы объединяем метод анализа динамики МТ в прометафазных клетках, описанных Ertych et al.44, с двойной вращающейся визуализацией диска. Этот метод позволяет измерять динамику МТ в прометафазных клетках, собранных из одного фокусного плана с более высокой частотой изображений, но без фотоотбелевания и минимальной фототоксичности. Кроме того, как флуоресцентный репортер, мы используем тандем димер томатный флуоресцентный белок (tdTomato), который улучшил яркость и фотостабильность по сравнению с зеленым флуоресцентным белком (EGFP) и возбуждается с более низким энергетическим светом46. Таким образом, tdTomato требует меньше лазерной энергии для возбуждения и менее фототоксичным. В целом, мы дополнительно улучшаем метод за счет снижения фототоксичности и улучшения разрешения и постобработки, необходимых для анализа динамики Mt. Кроме того, мы создаем основу для будущих модификаций метода, объединив его с другими методами синхронизации.
Access restricted. Please log in or start a trial to view this content.
протокол
1. Посев клеток HeLa
- Приготовьте 2 мл 5 мл/мл фибронектина раствор афериста (PBS) и добавьте 450 л его в каждую скважину 4 хорошо камерного покрытия (#1,5). Инкубировать слайд в течение 15 мин при 37 градусах по Цельсию и 5% CO2.
- Промыть асинхронно растущие клетки HeLa с фосфатным солей Dulbecco (DPBS) и инкубировать с трипсин-EDTA (0.05%: 0.02%; w:v) в течение 5 мин при 37 градусах Цельсия. Остановить ферментативную реакцию добавлением Розуэлл Парк Мемориальный институт (RPMI) 1640 средних дополнен 10% тепло-инактивированной сыворотки плода теленка (FCS) на 3:1 (v:v) соотношение добавил трипсин-EDTA.
ПРИМЕЧАНИЕ: Клетки HeLa были сохранены в среде RPMI 1640, дополненной 10% теплоинактивированной FCS при температуре 37 градусов по Цельсию и 5% CO2 и регулярно проходились, как только они достигли 80-90% непредвиденных обстоятельств, как описано выше. - Определить концентрацию клеток с помощью камеры Нойбауэра. Смешайте 50 qL aliquot клеточной подвески с трипан овым итога в соотношении 1:1 (v:v), resuspend, и перенесите 10 зл. Подсчитайте только трипан сине-отрицательных клеток внутри четырех больших квадратов (для деталей см. Фелан и др.47). Извлеките концентрацию клетки из подсчитанного числа клеток с помощью следующей формулы:
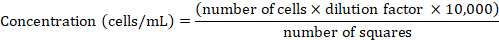
- Пеллети теки центрифугированием при 300 х г в течение 2 мин. Отдохните свежим RPMI 1640 для того, чтобы получить 1 х 106 клеток/мл.
- Удалите фибронектин из камерной крышкой, мыть колодцы дважды с DPBS, и семена 50000 клеток на хорошо.
- Вернуть камерный покрывало с клетками в инкубатор и выращивать их в течение 24 ч при 37 градусах Цельсия и 5% CO2.
2. Выражение pEB3-tdTomato в клетках HeLa
- Подготовьте микроцентрифугную трубку мощностью 1,5 мл. Для каждой трубки разбавьте 2 мкг pEB3-tdTomato48 с трансфекционным буфером (синтетический продукт в водином растворе) до конечного объема 396 qL.
- Добавьте в первую трубку 4 л трансфекционного реагента (нелипидного, содержащего полиэтиленина), и сразу же на 10 с.
- Кратко вращать трубку с микроцентрифугой и инкубировать при комнатной температуре (RT) в течение 10 минут.
- Удалите клетки HeLa из инкубатора. Dropwise, добавить 100 л трансфекционной смеси к каждому колодцу 4 хорошо камерной крышкой, и вернуть клетки в инкубатор.
- После 4 ч инкубации при 37 и 5% CO2, дополнить клетки со свежим средством роста среды и инкубировать, по крайней мере 24 ч при 37 КС и 5% CO2.
ПРИМЕЧАНИЕ: Необходимо оптимизировать условия трансфекции для каждого типа клеток. Уровни выражения должны быть достаточно низкими, чтобы определить одиночные концы роста МТ. Кроме того, в экспериментах может быть использована клеточная линия, четко выражающая EB3-tdTomato; это уменьшит изменчивость в уровнях экспрессии EB3-tdTomato между препаратами и между клетками из одного и того же препарата49.
3. Синхронизация и live-cell Imaging pEB3-tdTomato-выражение клеток HeLa
- Подготовьте раствор диметиленастрона (DME) в феноло-красном безварном орел-медиуме (DMEM) дополненном 10% FCS и 2 ММ L-глутамина или альтернативным запасом глутамина.
- Замените среду роста в камерном coverslip с 500 qL среды роста, содержащей 2,5 ММ DME и инкубировать клетки на 37 градусов по Цельсию и 5% CO2.
- После 3,5 ч инкубации с DME, передача клеток в микроскоп, смонтировать камерный покрывало в экологическую камеру с темными панелями для визуализации при 37 КС и 5% CO2, и далее инкубировать до тех пор, пока общее время инкубации составляет 4 ч.
ПРИМЕЧАНИЕ: Поддержание температуры при температуре 37 градусов без колебаний имеет решающее значение для эксперимента. - Выполните замедленное изображение на перевернутом микроскопе, оснащенном целью погружения масла 100x 1.49 N.A., двойной вращающейся дисковой конфокальной системой и надежной системой автофокусировки для непрерывного обслуживания фокусной плоскости. Определите параметры изображения следующим образом.
ПРИМЕЧАНИЕ: Мы используем камеру Electron Multiplying Charge-Coupled Device (EM-CCD).- Для EB3-tdTomato возбуждение, используйте 561 нм лазерной линии с 200 мс время экспозиции. Соберите излучаемый свет через четырехместный диапазон (405, 488, 561, 640 нм) дихроическое зеркало и фильтр для выбросов 600/52 нм.
ПРИМЕЧАНИЕ: Лазерная мощность может быть скорректирована для каждой изображенной ячейки, чтобы предотвратить насыщение изображения. Во всех фильмах замедленного пребывания здесь лазерная мощность была установлена на 5,3 мВт. - Найти клетку в профазе и сосредоточиться в плоскости, соответствующей центру монополярного митотического шпинделя. Приобретайте изображения каждые 0,5 с в общей сложности 1 мин без связывания и без освещения между экспозициями.
- Для EB3-tdTomato возбуждение, используйте 561 нм лазерной линии с 200 мс время экспозиции. Соберите излучаемый свет через четырехместный диапазон (405, 488, 561, 640 нм) дихроическое зеркало и фильтр для выбросов 600/52 нм.
4. Анализ динамики MT с использованием U-Track v2.2.0
- Для анализа динамики MT требуется программное обеспечение для расчетной среды (например, MATLAB).
ПРИМЕЧАНИЕ: Базового понимания программного обеспечения достаточно для анализа. Всеобъемлющий справочный материал и учебные материалы доступны на веб-сайте разработчика (https://uk.mathworks.com/products/matlab/getting-started.html). - Скачать (https://github.com/DanuserLab/u-track) и установить с открытым исходным кодом U-Track v2.2.0 программное обеспечение после подробных инструкций, приведенных в "Readme_u-track.pdf" файл50,51,52.
- Запустите программное обеспечение для количественного анализа и добавьте папку U-Track v2.2.0 с субфолиорами в путь поиска программного обеспечения.
- Из окна команды звоните "movieSelectorGUI". Это открывает окно диалога, из которого сырые файлы, генерируемые программным обеспечением приобретения изображения в микроскоп, могут быть импортированы(Дополнительная диаграмма 1, Рисунок 2, Рисунок 3, Рисунок 4).
ПРИМЕЧАНИЕ: Программное обеспечение U-Track совместимо с другими форматами данных изображений. Он использует Bio-Formats, который распознает различные форматы данных науки о жизни53. - Размер каждого изображения считывается из метаданных автоматически. Вручную введите численное отверстие цели (в данном случае 1,49) и временной интервал (0,5 с), используемый для визуализации(Дополнительная диаграмма 1B). Кроме того, информация о длине волн возбуждения, фторфоре и времени воздействия также может быть предоставлена, но они не являются критическими для дальнейшего анализа.
- После загрузки всех изображений сохраните вводимые временные промежутки в качестве списка фильмов, выбрав «Сохранить как список фильмов». На правой стороне окна диалога выберите вариант"U-Track"инажмите" Продолжить "(Дополнительная рисунок 1С).
ПРИМЕЧАНИЕ: Значения оптимизированы для клеток HeLa. При переключении на другую клеточную линию значения следует определить еще раз. Кроме того, используйте параметры, рекомендованные разработчиками программного обеспечения. Подробное объяснение каждого из параметров и того, как они должны быть определены, можно найти в техническом отчете, представленном предыдущей версией программного обеспечения, plusTipTracker50. - Из всплывающее окно выберите "Microtubule Plus-Ends" и нажмите "Ok"(Дополнительная рисунок 1С). Новое окно диалога позволяет определить параметры для трех этапов анализа(Дополнительная цифра 1D), которые являются обнаружение, отслеживание и отслеживание анализа.
- В шаге 1 выберите "Настройки" и из выпадающего меню выберите "Обнаружение кометы" в качестве метода обнаружения(Дополнительная диаграмма 2B).
- Из нового окна диалога определяются параметры разницы гауссийского фильтра и сегментации водораздела следующим образом(Дополнительная диаграмма 2C):Процесс маски, который будет использоваться для обнаружения и нет; Низкопроходивное стандартное отклонение Гауссиана - 1 пиксель; Высокопроходивное гауссское стандартное отклонение - 3 пикселя; Минимальный порог - 3 стандартных отклонения; Пороговый размер шага - 0,25 стандартных отклонений. Выберите "Применить настройки для всех фильмов" и "Применить".
- В шаге 2, параметры для соединения, закрытия зазора, слияния и расщепления, и Калман фильтр функции определяются в три этапа, как выделено в розовый, зеленый и синий, соответственно(Дополнительный рисунок 3B). Для этих шагов выберите "Microtubule Plus-end Dynamics" и из опции "Установка" определить значения, указанные в дополнительном рисунке 3C-E, соответственно.
- Для проблем с размерностью выберите "2" из выпадающего меню. Используйте максимальный разрыв, чтобы закрыть 5 кадров; Минимальная длина трековых сегментов от первого шага - 3 кадра. Как и прежде, выберите "Применить настройки для всех фильмов" и нажмите на "Применить".
- В шаге 3 анализа, обнаруженные треки МТ классифицируются(Дополнительный рисунок 4). В качестве метода анализа треков выберите «Классификацию динамики микротрубок»и определите параметры через кнопку«Установка»,указанную на дополнительной диаграмме 4B,C. После этого выберите поле«Применить настройки ко всем фильмам»и нажмите накнопку «Применить».
- После того, как все параметры определены, из окна "Control Panel-U-Track"(Дополнительная цифра 1D) выберите "Применить Check/Uncheck для всех фильмов" и "Запустите все фильмы" коробки и нажмите "Run". Это инициирует MT анализ замедленной серии.
- После завершения обработки фильма отображается сообщение "Ваш фильм (ы) успешно обработаны". Пресса"Ок",затем "Сохранить".
- Теперь можно смело выйти из программного обеспечения для количественного анализа. Результаты обработки фильмов хранятся в подфайлных структурах в виде m-файлов в папке, где хранятся необработанные файлы.
5. Статистический анализ динамики МТ
- Импортm-файлов в предпочтительную программу статистического анализа.
ПРИМЕЧАНИЕ: В нашем случае мы сначала импортируем файлы в стандартной электронной таблице, чтобы сделать их читаемыми. M-файлы содержат статистическую информацию (средний, средний и стандартный отклонение) по различным параметрам (например, скорость роста, mt-динамика). Подробный перечень параметров приведен в техническом отчете, представленном предыдущей версией программного обеспечения, plusTipTracker50,52. Генерируемые m-файлы также могут быть импортированы в другое программное обеспечение для обработки данных. - Выберите параметр "средняя скорость роста" и импортируйте его в таблицу для статистики и отображения. Введите информацию о других параметрах (например, "динамическость") либо в новой таблице, либо в новой колонке той же сгруппированной таблицы и сюжета.
Access restricted. Please log in or start a trial to view this content.
Результаты
Следуя данному протоколу, изложенному на рисунке 1A,плазмида pEB3-tdTomato была транслеэффектно выражена в асинхронно растущих клетках HeLa. Клетки были синхронизированы 48 ч после трансфекции на прометафазе через лечение DME(рисунок 1B). Этот шаг обеспечил, чтобы и...
Access restricted. Please log in or start a trial to view this content.
Обсуждение
Здесь мы описываем модификацию метода, впервые установленного Ertych et al.44. Наряду с несколькими другими модификациями, мы сочетаем эту технику анализа динамики МТ с двойной вращающейся дискковой конфокальной визуализации. Использование двойного вращающегося диска улучшае?...
Access restricted. Please log in or start a trial to view this content.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Мы благодарим членов Фонда световой микроскопии, Института экспериментальной медицины Макса-Планка, за их экспертные советы и поддержку.
Access restricted. Please log in or start a trial to view this content.
Материалы
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
Ссылки
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены