JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
단극성 미토틱 스핀들에서 디스크 현미경 검사법에 의한 미세소관 역학 측정
요약
여기에서 우리는 살아있는 세포 회전 디스크 공초점 현미경 검사법 및 MATLAB 기지를 둔 심상 처리를 사용하여 prometaphase에서 동기화된 세포에 있는 microtubule 역학 분석의 강력하고 상세한 방법을 제시합니다.
초록
우리는 살아있는 세포에 있는 microtubule 역학을 결정하기 위하여 확립된 방법의 수정을 기술합니다. 이 프로토콜은 마이크로소관(tdTomato 형광 단백질로 표시된 EB3)의 양성 말단과 회전 디스크 공초점 현미경을 사용하여 고속, 고해상도, 라이브 셀 이미징에 대한 유전자 부호 인코딩 된 마커의 발현을 기반으로합니다. 세포 주기 동기화 및 미세소관의 증가 된 밀도는 유사분열 세포에서 중추 분리를 억제함으로써 달성되며, 성장 분석은 오픈 소스 U-Track 소프트웨어를 사용하여 수행됩니다. 밝고 적색 이동형 형광 단백질을 사용하여 낮은 레이저 파워와 함께 디스크 현미경 검사법에 필요한 노출 시간을 줄여 광독성과 빛으로 인한 유물의 확률을 감소시입니다. 이것은 표준 배양 조건의 성장 배지에서 세포를 유지하면서 동일한 제제에서 더 많은 수의 세포를 이미징할 수 있게 한다. 분석은 감독된 자동 방식으로 수행되므로 통계적으로 강력하고 재현 가능한 결과를 제공합니다.
서문
미세소관 (MTs)은 거의 모든 진핵 세포와 일부 박테리아1에서발견되는 매우 역동적 인 구조입니다. 액틴 및 중간 필라멘트와 함께 세포 골격2,3을조각합니다. 세포 분열4,분자 수송5,6을치는 편모, 1 차 실륨7,청각 (kinocilium)8,9,배아 발생10,11,12,침략 및 전이13,14,심지어 기억 형성15,16,17,18, 그리고 다른 많은 프로세스는 주로 MTs에 의존. 이러한 모든 이벤트에 있는 MTs의 참여는 급속하게 성장 (중합) 및 수축 (탈중합) 사이를 전환하는 그들의 놀라운 능력 없이는 불가능할 것입니다. 이 속성은 동적불안정성 19로설명됩니다. MT 역학은 많은 병리학 적 조건20,21,22에서변경됩니다. 따라서이 속성의 특성을 결정하는 것은 질병 메커니즘과 그 후의 치료를 이해하는 데 도움이 될 수 있습니다.
MT 역학 분석을 위한 방법의 긴 목록은, 대부분은 화상 진찰 기술23에근거를 둔 개발되었습니다. 초기에, 넓은 필드 광 현미경은 시험관 내 의 tubulin 중합체의 형성을 관찰하기 위해 사용되었다24. MT 플러스 엔드에서 수집되는 단자 결합(EB)-단백질의 발견과 형광성 단백질을 표지하는 방법의 개발은 넓은 필드 및 공초점 형광 현미경25,26,27를가진 살아있는 세포에서 직접 MT의 거동을 관찰할 수 있게 하였다. EB-단백질 1개(EB3)는 말단결합 단백질 3(EB3)28; 형광 단백질에 융합된 EB3를 과발현 및 추적함으로써, MT 플러스 엔드 조립속도는 29,30으로결정될 수 있다.
공초점 레이저 스캐닝 형광 현미경 검사법(CLSM)은 MT 역학을 따르기 위해 자주 사용됩니다. 그러나, 이 화상 진찰 기술은 광독성 및 광표백의 고위험, 살아있는 세포 및 희미한 견본 화상 진찰을 위한 2개의 바람직하지 않은 프로세스를 포즈31. 더 나은 신호 대 잡음 비를 얻으려면 레이저 전력과 노출 기간이 충분히 높아야하며 샘플을 손상시키지 않아야하며 속도를 대가로 해상도를 희생해야합니다. CLSM에 적합한 대안은 디스크현미경(32)을회전하는 것입니다. 이 이미징 양식은 핀홀배열을 포함하는 움직이는 디스크로 구성된 Nipkow디스크(33)의사용을 기반으로 하며, 동일한 샘플을 동시에34개시료를 이미징하는 많은 CLS 현미경과 동등하게 작동한다. 따라서 레이저의 빛은 시료의 여러 영역을 동시에 비추지만 공초점 특성을 유지합니다. 따라서 Nipkow 디스크를 사용하면 CLSM과 유사하지만 더 빠르고 레이저 전력을 적게 사용하여 이미지를 얻을 수 있습니다. Nipkow 디스크는 요코가와 일렉트릭에 의해 더욱 개선되었으며, 각 핀홀에 개별적으로 빛을 직접 전달하는 마이크로 렌즈 배열이있는 두 번째 디스크를 도입하여 광독성및 광 표백35를더욱 감소시켰습니다. 따라서, 회전 디스크 레이저 스캐닝 현미경 은 라이브 셀 이미징을위한 선택의 방법이되었고, 고속31,36에서높은 신호 대 잡음 비로 이미지를 얻을 수있게하며, 이는 빠르게 성장하는 MT 끝에서와 같은 신호를 해결하는 데 중요합니다.
MT 역학은 일시적으로 다릅니다. 예를 들어, 유사분열 MT는37,38. 유사하게, 유사하게, 유사하게, 유사하게, 유사하게, 유사하게 증가율 및 수축의 차이는 유사분열39,40과같은 동일한 세포 주기 단계 내에서도 관찰되었다. 따라서 잘못된 데이터 수집을 방지하기 위해 MT 역학의 측정은 셀 주기 동안 좁은 시간 창으로 제한되어야 합니다. 예를 들어, 프로메타상에서의 MT 역학의 측정은 모터 키네신 Eg541을 억제하고 양극성 유사분열스핀들(42)의형성을 방지하는 모나스트랄 유사체인 디메틸레나스트론(DME)으로 세포를 처리함으로써 달성될 수 있다. Eg5 억제제 DME 및 다른 모나스트랄 유도체와 함께 프로메타상에서 세포의 억제는 MT 역학43,44,45에영향을 미치지 않으며, 이는 DME를 고정 및 살아있는 세포 모두에서 MT 역학을 연구하는 데 유용한도구(44)로만든다.
여기에서 우리는 Ertych 등44에 의해 기술된 prometaphase 세포에 있는 MT 역학 분석의 방법을 이중 회전 디스크 화상 진찰을 결합합니다. 이 방법을 사용하면 광표백과 최소한의 광독성 없이 단일 초점 평면에서 수집된 전미상 세포에서 MT 역학을 측정할 수 있습니다. 또한, 형광 리포터로서, 녹색 형광 단백질(EGFP)에 비해 밝기와 광안정성을 향상시키고 저에너지라이트(46)로흥분하는 탠덤 디머 토마토 형광 단백질(tdTomato)을 사용합니다. 따라서 tdTomato는 여기를 위해 레이저 전력을 덜 필요로하며 광독성이 적습니다. MT 역학 분석에 필요한 광독성을 줄이고 분해능 및 후처리를 개선하여 방법을 더욱 개선합니다. 또한 다른 동기화 기술과 결합하여 메서드를 향후 수정할 수 있는 기반을 만듭니다.
Access restricted. Please log in or start a trial to view this content.
프로토콜
1. 헬라 세포의 파종
- 인산완식염수(PBS)에 5 μg/mL 섬유넥틴 용액 2mL를 준비하고 4개의 잘 챔버된 커버슬립(#1.5)의 각 우물에 450 μL을 추가합니다. 슬라이드를 37 °C 및 5 %CO2에서15 분 동안 배양합니다.
- 덜베코의 인산완충식염수(DPBS)로 비동기적으로 성장하는 헬라 세포를 헹구고 트립신-EDTA(0.05%: 0.02%,w:v)로 배양하여 37°C에서 5분 동안 배양합니다. 로스웰 공원 기념 연구소의 추가에 의해 효소 반응을 중지 (RPMI) 1640 중간 보충 10% 열 불활성화 태아 송아지 혈 청 (FCS) 에 3:1 (v:v) 추가 트립신-EDTA의 비율.
참고: HeLa 세포는 37°C 및 5%CO2에서 10% 열 불활성화 FCS로 보충된 RPMI 1640 배지에서 유지되었고, 전술한 바와 같이 80-90% 합류에 도달한 후에 일상적으로 통과되었다. - 노이바우어 챔버를 사용하여 세포 농도를 결정합니다. 1:1(v:v) 비율로 트리판 블루와 셀 서스펜션의 50 μL aliquot를 혼합하고, 다시 중단하고, 챔버 내로 10 μL을 전달한다. 4개의 큰 사각형 안에 있는 트라이판 청음 세포만 카운트합니다(자세한 내용은 Phelan 등47참조). 다음 공식을 사용하여 계수 된 셀 번호에서 세포 농도를 유도하십시오.
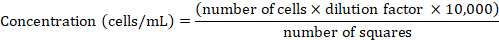
- 300 x g에서 2 분 동안 원심 분리하여 세포를 펠렛. 신선한 RPMI 1640으로 다시 일시 중단하여 1 x 106 세포 / mL을 얻습니다.
- 챔버 커버 슬립에서 섬유넥틴을 제거하고, DPBS로 우물을 두 번 씻고, 잘 당 50,000 세포를 시드하십시오.
- 챔버된 커버슬립을 세포와 함께 인큐베이터로 되돌리고 37°C 및 5%CO2에서24시간 동안 성장시다.
2. 헬라 세포에서 pEB3-tdTomato의 발현
- 1.5 mL 미세 원심 분리튜브를 준비합니다. 각 튜브에 대해, pEB3-tdTomato48의 2 μg를 형질전환 완충제(수성 용액의 합성 제품)로 396 μL의 최종 부피로 희석합니다.
- 첫 번째 튜브에 4 μL의 형질 감염 시약 (비 지질, 폴리에틸렌민 함유)을 추가하고 정확히 10 초 동안 혼합물을 즉시 소용돌이치십시오.
- 미세 원심 분리기로 튜브를 간단히 회전시키고 실온 (RT)에서 10 분 동안 배양하십시오.
- 인큐베이터에서 HeLa 세포를 제거합니다. 드롭와이즈, 4 개의 잘 챔버 커버 슬립의 각 웰에 형질 감염 혼합물의 100 μL을 추가하고, 인큐베이터에 세포를 반환합니다.
- 37°C 및 5%CO2에서4시간 배양한 후, 신선한 성장 배지로 세포를 보충하고 37°C 및 5%CO2에서적어도 24시간 동안 배양한다.
참고: 각 세포 유형에 대한 형질 감염 조건을 최적화 할 필요가있다. 단일 MT 성장 끝을 식별할 수 있을 만큼 표현 수준이 충분히 낮아야 합니다. 대안적으로, EB3-tdTomato를 안정적으로 발현하는 세포주가 실험에 사용될 수 있다; 이는 동일한 제제49에서제제 와 세포 들 사이의 EB3-tdTomato의 발현 수준에서 의 가변성을 감소시킬 것이다.
3. pEB3-tdTomato의 동기화 및 라이브 셀 이미징 - 발현 HeLa 세포
- 페놀-레드 프리 덜베코의 변형 된 독수리 배지 (DMEM)에 디메틸레나스트론 (DME)의 2.5 μM 용액을 10 % FCS 및 2 mM L-글루타민 또는 대체 글루타민 공급으로 보충하십시오.
- 챔버형 커버슬립의 성장 배지를 2.5 μM DME를 함유하는 성장 배지의 500 μL로 교체하고 37°C 및 5%CO2에서세포를 배양한다.
- DME로 3.5 시간 의 인큐베이션 후, 세포를 현미경으로 옮기고, 챔버형 커버슬립을 37°C 및 5%CO2에서이미징을 위한 어두운 패널이 있는 환경 챔버에 장착하고, 총 배양 시간이 4시간될 때까지 추가로 인큐베이션한다.
참고: 변동없이 37 °C에서 온도의 유지는 실험에 매우 중요합니다. - 100x 1.49 N.A. 오일 침지 목표, 이중 방적 디스크 공초점 시스템 및 초점 평면의 지속적인 유지 관리를 위한 신뢰할 수 있는 자동 초점 시스템이 장착된 반전 현미경으로 타임랩스 이미징을 수행합니다. 다음과 같이 이미징 매개 변수를 정의합니다.
참고: 우리는 전자 증식 충전 결합 장치 카메라 (EM-CCD)를 사용합니다.- EB3-tdTomato 여기의 경우, 200 ms 노출 시간과 561 nm 레이저 라인을 사용합니다. 4중 대역통과(405, 488, 561, 640 nm) 이색 거울과 600/52 nm 방출 필터를 통해 방출된 빛을 수집합니다.
참고: 이미지 채도를 방지하기 위해 각 이미지 셀에 대해 레이저 전력을 조정할 수 있습니다. 여기에 주어진 모든 시간 경과 영화에서 레이저 전력은 5.3 mW로 설정되었습니다. - 단극 유사분열 스핀들의 중심에 해당하는 Z 평면에서 위상 및 초점에서 셀을 찾습니다. 노출 사이에 비닝과 조명없이 총 1 분 동안 0.5 초마다 이미지를 수집합니다.
- EB3-tdTomato 여기의 경우, 200 ms 노출 시간과 561 nm 레이저 라인을 사용합니다. 4중 대역통과(405, 488, 561, 640 nm) 이색 거울과 600/52 nm 방출 필터를 통해 방출된 빛을 수집합니다.
4. U-트랙 v2.2.0을 이용한 MT 역학 분석
- MT 역학을 분석하려면 수치 컴퓨팅 환경 소프트웨어가 필요합니다(예: MATLAB).
참고: 소프트웨어에 대한 기본적인 이해는 분석에 충분합니다. 포괄적인 도움말 자료및 자습서는 개발자 웹 사이트(https://uk.mathworks.com/products/matlab/getting-started.html)에서 확인할 수 있습니다. - 다운로드 (https://github.com/DanuserLab/u-track) 및 "Readme_u-track.pdf"파일50,51,52에주어진 자세한 지침에 따라 오픈 소스 U-트랙 v2.2.0 소프트웨어를 설치합니다.
- 수치 분석 소프트웨어를 시작하고 소프트웨어 검색 경로에 하위 폴더가 있는 U-Track v2.2.0 폴더를 추가합니다.
- 명령 창 에서 호출 "영화선택기". 이는 현미경에서 이미지 수집 소프트웨어에 의해 생성된 원시 파일을 가져올 수 있는 대화 창을 열어줍니다(보충도 1, 도 2, 도 3, 도 4).
참고: U-Track 소프트웨어는 다른 이미지 데이터 형식과 호환됩니다. 그것은 다른 생명 과학 데이터 형식을 인식 하는 바이오 형식을 사용 하 여53. - 각 이미지의 크기는 메타데이터에서 자동으로 읽습니다. 수동으로 영상에 사용되는 목표의 수치 조리개(이 경우 1.49)와 시간 간격(0.5s)을 입력합니다(보조도 1B). 또한, 여기 파장, 형광부 및 노출 시간에 대한 정보도 제공될 수 있지만 추가 분석을 위해 중요하지는 않습니다.
- 모든 이미지가 로드되면"영화 목록으로 저장"을선택하여 입력한 타임랩스 시리즈를 동영상 목록으로 저장합니다. 대화 창의 오른쪽에있는"U-트랙" 옵션을 선택하고"계속"(보충 그림 1C)를누릅니다.
참고: 이 값은 HeLa 셀에 최적화되어 있습니다. 다른 세포줄로 전환하는 경우 값을 다시 정의해야 합니다. 또는 소프트웨어 개발자가 권장하는 설정을 사용합니다. 각 매개 변수에 대한 자세한 설명과 매개 변수를 정의해야하는 방법은 소프트웨어의 이전 버전과 함께 제공되는 기술 보고서에서 찾을 수 있습니다, 플러스팁 Tracker50. - 팝업 창에서"마이크로스터플러스-엔드"를선택하고 "확인"(보조 그림 1C)를누릅니다. 새 대화 창을 사용하면 검색, 추적 및 추적 분석인 분석의 세단계(추가 그림 1D)에대한 매개 변수를 결정할 수 있습니다.
- 1단계에서는 "설정"을 선택하고 드롭다운 메뉴에서 "혜성 감지"를 감지 방법으로 선택합니다(보조그림 2B).
- 새 대화 창으로부터 가우시안 필터와 유역 세분화의 차이에 대한 파라미터를 다음과 같이 정의한다(보조도 2C):검출에 사용할 마스크 공정 =없음; 로우 패스 가우시안 표준 편차 = 1 픽셀; 하이 패스 가우시안 표준 편차 = 3 픽셀; 최소 임계값 = 3개의 표준 편차; 임계값 단계 크기 = 0.25 표준 편차입니다. "모든동영상에 설정 적용"및"적용"을선택합니다.
- 2단계에서는 연결, 간격 닫기, 병합 및 분할 및 Kalman 필터 함수에 대한 매개 변수가 분홍색, 녹색 및 파란색으로 강조 표시된 대로 세 단계로 정의됩니다(추가그림 3B). 이러한 단계의 경우 "마이크로스터울 플러스 엔드 다이내믹스"를 선택하고 "설정" 옵션에서 각각 추가 그림 3C-E에표시된 값을 정의합니다.
- 차원에 문제가 있는 경우 드롭다운 메뉴에서 "2"를 선택합니다. 최대 간격을 사용하여 닫으려면 = 5프레임; 첫 번째 단계의 최소 트랙 세그먼트 길이 = 3프레임입니다. 이전과 마찬가지로 "모든 동영상에 설정 적용"을선택하고"적용"을클릭합니다.
- 분석의 3단계에서, 검출된 MT 트랙은분류된다(보충 도 4). 트랙 분석 방법으로 ,"미세 관 역학 분류"를 선택하고 보충 그림 4B, C에표시된 대로"설정"버튼을 통해 매개 변수를 정의합니다. 그런 다음 "모든 동영상에 설정 적용" 상자를 선택하고"적용"을클릭합니다.
- 모든 매개 변수가 정의되면,"제어판 - U-트랙" 창(보충 그림 1D)에서"모든 동영상에 선택 / 선택 취소적용 "을 선택하고 " 모든 영화를실행" 상자를 실행하고 "실행 "를누릅니다. 이렇게 하면 타임랩스 계열의 MT 분석이 시작됩니다.
- 동영상 처리가 완료되면 "동영상이 성공적으로 처리되었습니다"라는 메시지가 표시됩니다. "확인"을누르면 "저장".
- 이제 수치 분석 소프트웨어를 종료하는 것이 안전합니다. 동영상 처리 의 결과는 원시 파일이 저장되는 폴더의 m 파일로 하위 폴더 구조에 저장됩니다.
5. MT 역학의 통계 분석
- m-파일을 기본 통계 분석 프로그램으로 가져옵니다.
참고: 이 경우 먼저 표준 스프레드시트에서 파일을 가져와 읽을 수 있도록 합니다. m-files에는 다양한 매개 변수(예: 성장 속도, MT 동태도)에 대한 통계 정보(중앙값, 평균 및 표준 편차)가 포함되어 있습니다. 매개 변수의 자세한 목록은 소프트웨어의 이전 버전과 함께 제공되는 기술 보고서에 제공되며, plusTipTracker50,52. 생성된 m-파일은 다른 데이터 처리 소프트웨어로 가져올 수도 있습니다. - "성장 속도 평균" 매개변수를 선택하고 통계 및 표시를 위해 테이블로 가져옵니다. 새 테이블이나 동일한 그룹화 된 테이블 및 플롯의 새 열에 다른 매개 변수(예: "dynamicity")에 대한 정보를 입력합니다.
Access restricted. Please log in or start a trial to view this content.
결과
도 1A에설명된 주어진 프로토콜에 따라, pEB3-tdTomato 플라스미드는 비동기적으로 성장하는 HeLa 세포에서 일시적으로 발현되었다. 세포는 DME 처리를 통해 프로메타상에서 형질전환 후 48시간 동기화되었다(도1B). 이 단계는 MT 역학의 측정이 항상 세포 주기의 동일한 단계에서 수행되었다는 것을 보장했습니다. 타임랩스 동영상은 보충 문서
Access restricted. Please log in or start a trial to view this content.
토론
여기서, 우리는 Ertych 등44에의해 처음 설립 된 방법의 수정을 설명합니다. 몇 가지 다른 수정과 함께, 우리는 이중 회전 디스크 공초점 이미징MT 역학 분석의이 기술을 결합합니다. 듀얼 스피닝 디스크를 사용하면 증가하는 MT의 해상도를 향상시키는 동시에 광독성36을줄입니다. 우리는 더 긴 파장 형광 리포터로 전환하여 광 표백 및 레이저 광 유도 세포의 손상?...
Access restricted. Please log in or start a trial to view this content.
공개
저자는 공개 할 것이 없다.
감사의 말
우리는 그들의 전문가 적인 조언 및 지원에 대한 빛 현미경 검사시설, 실험 의학의 막스 플랑크 연구소의 회원들에게 감사드립니다.
Access restricted. Please log in or start a trial to view this content.
자료
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
참고문헌
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유