É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Medição da dinâmica do microtúbulo girando microscopia do disco em fusos mitocticos monopolares
Neste Artigo
Resumo
Aqui apresentamos um método robusto e detalhado de análise da dinâmica dos microtúbulos em células sincronizadas em prometaphase usando microscopia confocal de disco giratório de células vivas e processamento de imagem baseado em MATLAB.
Resumo
Descrevemos uma modificação de um método estabelecido para determinar a dinâmica dos microtúbulos nas células vivas. O protocolo é baseado na expressão de um marcador geneticamente codificado para as extremidades positivas dos microtúbulos (EB3 rotulado com proteína fluorescente tdTomato) e imagens de células vivas de alta velocidade, alta resolução, usando microscopia confocal de disco giratório. A sincronização do ciclo celular e o aumento da densidade de microtúbulos são alcançados inibindo a separação centrosomal em células mitocômicas, e a análise do crescimento é realizada usando software u-track de código aberto. O uso de uma proteína fluorescente brilhante e em vermelho, em combinação com o menor poder laser e o tempo de exposição reduzido necessário para a microscopia de disco giratório, reduzem a fototoxicidade e a probabilidade de artefatos induzidos pela luz. Isso permite a imagem de um número maior de células na mesma preparação, mantendo as células em um meio de crescimento condições de cultura padrão. Como a análise é realizada de forma automática supervisionada, os resultados são estatisticamente robustos e reprodutíveis.
Introdução
Microtubules (MTs) são estruturas altamente dinâmicas encontradas em praticamente todas as células eucarióticas e em algumas bactérias1. Juntamente com a actina e filamentos intermediários, eles esculpem o citoesqueleto2,3. Divisão celular4,transporte demoléculas 5,flagelar batendo6,a sensação do ambiente circundante através do clácia primário7, audição (kinocilium)8,9,embrionênese10,11,12, invasão e metástase13,14,e até mesmo formação de memória15,16,17,18, e muitos outros processos dependem principalmente de MTs. A participação de MTs em todos esses eventos seria impossível sem sua notável capacidade de alternar rapidamente entre crescimento (polimerização) e encolhimento (despolimerização). Esta propriedade é descrita como instabilidade dinâmica19. A dinâmica do MT é alterada em muitas condições patológicas20,21,22. Assim, determinar a natureza desta propriedade pode ajudar a compreender mecanismos da doença e subseqüentemente seu tratamento.
Uma longa lista de métodos foi desenvolvida para a análise da dinâmica MT, a maioria dos quais são baseados em técnicas de imagem23. Inicialmente, microscópios de luz de campo largo foram utilizados para observar a formação de polímeros de tubulina in vitro24. A descoberta de proteínas end-binding (EB) que se coletam em MT plus-ends e o desenvolvimento de métodos para rotular fluorescentemente proteínas possibilitou observar o comportamento das MTs diretamente em células vivas com amplo campo e microscópios de fluorescência confocal25,26,27. Uma proteína EB é a proteína de ligação final 3 (EB3)28; expressando e rastreando eB3 com uma proteína fluorescente, as taxas de montagem mt plus-end podem ser determinadas29,30.
A microscopia de fluorescência da exploração a laser confocal (CLSM) é frequentemente usada para acompanhar a dinâmica do MT. No entanto, esta técnica de imagem representa um alto risco de fototoxicidade e fotobranqueamento, dois processos indesejáveis para células vivas e imagens de amostra dim31. A fim de obter uma melhor relação sinal-ruído, a potência do laser e a duração da exposição devem ser altas o suficiente, sem danificar as amostras, e isso requer sacrificar a resolução em troca de velocidade. Uma alternativa adequada ao CLSM é a microscopia de disco giratório32. Esta modalidade de imagem é baseada no uso de um disco Nipkow33, que consiste em um disco em movimento com uma matriz de furos, e funciona de forma equivalente a muitos microscópios CLS imagens da mesma amostra simultaneamente34. Portanto, a luz do laser iluminará várias regiões da amostra simultaneamente, mas manterá a natureza confocal. O disco nipkow, portanto, permite a obtenção de imagens semelhantes ao CLSM, mas mais rápido e usando menos energia a laser. O disco Nipkow foi ainda melhor pelo Yokogawa Electric, que introduziu um segundo disco com uma variedade de microlentes sobre ele que individualmente direcionar a luz em um pinhole respectivos, reduzindo ainda mais fototoxicidade e fotobranqueamento35. Assim, a microscopia de digitalização a laser de disco giratório tornou-se um método de escolha para imagens de células vivas, e torna possível obter imagens com alta relação sinal-ruído em alta velocidade31,36, o que é crucial para resolver sinais como os das extremidades de rápido crescimento do MT.
A dinâmica do MT difere temporariamente. Por exemplo, as MTs mitocôticas são mais dinâmicas do que asinterfases 37,38. Da mesma forma, diferenças na taxa de crescimento e encolhimento têm sido observadas mesmo dentro da mesma fase do ciclo celular, como a mitose39,40. Portanto, para evitar a coleta de dados falsos, a medição da dinâmica do MT deve ser limitada a uma janela de tempo estreita durante o ciclo celular. Por exemplo, a medição da dinâmica mt na prometaphase pode ser alcançada através do tratamento das células com dimenastron (DME), um análogo monastrol que inibe o motor kinesin Eg541 e impede a formação do fuso mitomático42. Inibição de células em prometaphase com DME inibidor eg5 e outros derivados monastrois não afeta a dinâmica MT43,44,45, o que torna DME uma ferramenta útil para estudar a dinâmica mt tanto em células fixas e vivas44.
Aqui combinamos o método de análise de dinâmica MT em células prometaphase descritas por Ertych et al.44 com imagens de disco de fiação dupla. Este método permite a medição da dinâmica mt em células prometaphase coletadas a partir de um único plano focal com uma maior taxa de imagem, mas sem fotobranqueamento e fototoxicidade mínima. Além disso, como repórter fluorescente, usamos proteína fluorescente de tomate tandem dimer (tdTomato), que melhorou o brilho e a fotostabilidade em comparação com a proteína fluorescente verde (EGFP) e está animada com luz de energia mais baixa46. Portanto, tdTomato requer menos energia laser para excitação e é menos fototóxico. Ao todo, melhoramos ainda mais o método, reduzindo a fototoxicidade e melhorando a resolução e pós-processamento necessários para a análise da dinâmica do MT. Além disso, criamos uma base para futuras modificações do método, combinando-o com outras técnicas de sincronização.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Semeadura de células HeLa
- Prepare 2 mL de 5 μg/mL solução de fibronectina em soro fisiológico tampão de fosfato (PBS) e adicione 450 μL dele em cada poço de um coverslip de 4 poços com câmara (#1,5). Incubar o slide por 15 min a 37 °C e 5% CO2.
- Enxágüe células HeLa de crescimento assíncrono com a soro-sinodiada de Dulbecco (DPBS) e incubada com trippsina-EDTA (0,05%: 0,02%; w:v) por 5 min a 37 °C. Pare a reação enzimática pela adição do Roswell Park Memorial Institute (RPMI) 1640 médio complementado com 10% de calor inativado soro de vitela fetal (FCS) em 3:1 (v:v) relação de trippsina adicionada-EDTA.
Nota: As células HeLa foram mantidas no rpmi 1640 médio complementado com 10% de calor inativado FCS em 37 °C e 5% CO2 e foram rotineiramente passados uma vez que atingiu 80-90% de confluência, conforme descrito acima. - Determine a concentração celular usando uma câmara de Neubauer. Misture um alíquota de 50 μL da suspensão celular com trippan azul em 1:1 (v:v) relação, suspender e transferir 10 μL da suspensão para a câmara. Conte apenas as células azul-negativas trippan dentro dos quatro grandes quadrados (para detalhes ver Phelan et al.47). Derivar a concentração celular do número de células contados usando a seguinte fórmula:
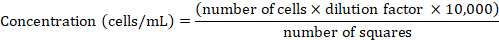
- Pelotas as células por centrífuga a 300 x g para 2 min. Resuspende com RPMI 1640 fresco, a fim de obter 1 x 106 células / mL.
- Retire a fibronectina do coverslip com câmara, lave os poços duas vezes com DPBS, e sementes 50.000 células por poço.
- Devolva o coverslip com câmara com as células para a incubadora e cultivá-los por 24 h a 37 °C e 5% CO2.
2. Expressão de pEB3-tdTomato em células HeLa
- Prepare um tubo de microcentrírígio de 1,5 mL. Para cada tubo, diluir 2 μg de pEB3-tdTomato48 com buffer de transfecção (produto sintético em solução aquosa) para um volume final de 396 μL.
- Adicione 4 μL de reagente de transfecção (não lipídico, contendo polietilenomina) ao primeiro tubo, e vórtice a mistura imediatamente por exatamente 10 s.
- Gire brevemente para baixo o tubo com uma microcentrífuga e incubar à temperatura ambiente (RT) por 10 min.
- Retire as células HeLa da incubadora. Dropwise, adicione 100 μL da mistura de transfecção para cada poço de um coverslip 4 bem com câmara, e retornar as células para a incubadora.
- Após 4 h de incubação a 37 °C e 5% CO2,complemente as células com médio de crescimento fresco e incubam por pelo menos 24 h a 37 °C e 5% CO2.
Nota: É necessário otimizar as condições de transfecção para cada tipo de célula. Os níveis de expressão precisam ser baixos o suficiente para permitir a identificação de fins de crescimento de MT único. Alternativamente, uma linha celular expressando eb3-tdTomato pode ser usado nos experimentos; isso reduziria a variabilidade nos níveis de expressão de EB3-tdTomato entre as preparações e entre as células da mesma preparação49.
3. Sincronização e imagens de células vivas de células HeLa expressando pEB3-tdTomato- expressando células HeLa
- Prepare uma solução de 2,5 μM de dimenastron (DME) em fenol-vermelho livre Dulbecco's Modified Eagle Medium (DMEM) complementada com 10% FCS e 2 mM L-glutamina ou uma oferta alternativa de glutamina.
- Substitua o meio de crescimento no coverslip com câmara com 500 μL do meio de crescimento contendo 2,5 μM DME e incubar as células em 37 °C e 5% CO2.
- Após 3,5 h de incubação com DME, transfira as células para o microscópio, monte o coverslip com câmara em uma câmara ambiental com painéis escuros para imagem a 37 °C e 5% CO2,e incubar ainda mais até que o tempo total de incubação seja de 4 h.
Nota: A manutenção da temperatura a 37 °C sem flutuação é crucial para o experimento. - Realize a imagem de lapso de tempo em um microscópio invertido equipado com um objetivo de imersão de óleo 100x 1.49 N.A., um sistema confocal de disco giratório duplo e um sistema de foco automático confiável para manutenção contínua do plano focal. Defina os parâmetros de imagem da seguinte forma.
Nota: Usamos uma câmera de dispositivo acoplado a carga multiplicadora de elétrons (EM-CCD).- Para a excitação eb3-tdTomato, use uma linha laser de 561 nm com 200 ms tempo de exposição. Colete a luz emitida através de um bandpass quádruplo (405, 488, 561, 640 nm) espelho dicroico e um filtro de emissão de 600/52 nm.
Nota: O poder do laser pode ser ajustado para cada pilha imaged para impedir a saturação da imagem. Em todos os filmes de lapso de tempo dado aqui o poder laser foi definido como 5,3 mW. - Encontre uma célula em profase e foco no z-avião correspondente ao centro do eixo mitomótico monopolar. Adquirir imagens a cada 0,5 s ao longo de um total de 1 min, sem binning e sem iluminação entre as exposições.
- Para a excitação eb3-tdTomato, use uma linha laser de 561 nm com 200 ms tempo de exposição. Colete a luz emitida através de um bandpass quádruplo (405, 488, 561, 640 nm) espelho dicroico e um filtro de emissão de 600/52 nm.
4. Análise da dinâmica MT usando U-Track v2.2.0
- Para analisar a dinâmica mt um software de ambiente de computação numérica é necessário (por exemplo, MATLAB).
Nota: A compreensão básica do software é suficiente para a análise. Material de ajuda abrangente e tutoriais estão disponíveis no site do desenvolvedor (https://uk.mathworks.com/products/matlab/getting-started.html). - Baixe (https://github.com/DanuserLab/u-track) e instale o software u-track v2.2.0 de código aberto seguindo as instruções detalhadas dadas no arquivo "Readme_u-track.pdf"50,51,52.
- Lance o software de análise numérica e adicione a pasta U-Track v2.2.0 com subdobras no caminho de pesquisa de software.
- A partir da chamada janela de comando "filmeSelectorGUI". Isso abre uma janela de diálogo a partir da qual os arquivos brutos gerados pelo software de aquisição de imagem no microscópio podem ser importados(Figura Suplementar 1, Figura 2, Figura 3, Figura 4).
Nota: O software U-Track é compatível com outros formatos de dados de imagem. Ele usa bio-formatos, que reconhece diferentes formatos de dados de ciênciasi vida 53. - O tamanho de cada imagem é lido a partir dos metadados automaticamente. Digite manualmente a abertura numérica do objetivo (neste caso 1,49) e o intervalo de tempo (0,5 s) utilizado para imagem(Figura Suplementar 1B). Além disso, informações sobre o comprimento de onda de excitação, o fluorofofóbico e o tempo de exposição também podem ser fornecidas, mas não são críticas para uma análise mais aprofundada.
- Uma vez que todas as imagens são carregadas, salvar a série de lapso de tempo inscrita como uma lista de filmes, selecionando o "Save As Movie List". No lado direito da janela de diálogo selecione a opção "U-Track" e pressione "Continue" (Figura Suplementar 1C).
Nota: Os valores são otimizados para as células HeLa. Se mudar para uma linha celular diferente, os valores devem ser definidos novamente. Alternativamente, use as configurações recomendadas pelos desenvolvedores de software. A explicação detalhada de cada um dos parâmetros e como eles devem ser definidos pode ser encontrada no relatório técnico fornecido com a versão anterior do software, plusTipTracker50. - A partir da janela pop-up selecionar "Microtubule Plus-Ends" e pressione "Ok" (Figura Suplementar 1C). A nova janela de diálogo permite determinar os parâmetros para as três etapas da análise(Figura Suplementar 1D),que são detecção, rastreamento e análise de rastreamento.
- Na etapa 1 escolha "Configurações" e de um menu suspenso selecione "Detecção de cometa" como um método de detecção(Figura Suplementar 2B).
- A partir da nova janela de diálogo definem os parâmetros para a diferença do filtro gaussians e a segmentação da bacia hidrográfica da seguinte forma(Figura Complementar 2C):Processo de máscara a ser utilizado para a detecção = Nenhum; Desvio padrão gaussiano de passagem baixa = 1 pixel; Desvio padrão gaussiano de alta passagem = 3 pixels; Limite mínimo = 3 desvios padrão; Tamanho da etapa do ponto inicial = 0.25 desvios padrão. Selecione"Aplicar configurações para todos os filmes"e"Aplicar".
- Na etapa 2, os parâmetros para a ligação, fechamento de lacunas, fusão e divisão, e as funções de filtro Kalman são definidos em três etapas, como destacado em rosa, verde e azul, em conformidade(Figura Suplementar 3B). Para essas etapas, selecione a "Microtubule Plus-end Dynamics" e da opção "Configuração" definem os valores indicados na Figura Suplementar 3C-E,respectivamente.
- Para problemas com dimensionalidade, escolha "2" do menu suspenso. Use a diferença máxima para fechar = 5 quadros; Comprimento mínimo de segmentos da trilha da primeira etapa = 3 frames. Como antes, selecione"Aplicar configurações para todos os filmes"e clique em"Aplicar".
- Na etapa 3 da análise, as faixas de MT detectadas são classificadas(Figura Suplementar 4). Como método de análise de pista, escolha "Microtubule Dynamics Classification" e definir os parâmetros através do botão "Configuração" como indicado na Figura Suplementar 4B, C. Depois disso, escolha o"Aplicar Configurações para todos os filmes"caixa e clique em "Aplicar".
- Uma vez que todos os parâmetros são definidos, a partir do "Painel de Controle -U-Track" janela (Figura Suplementar 1D) selecione o "Aplicar Check / Desmarcada para todos os filmes" e "Executar todos os filmes" caixas e imprensa "Run". Isso iniciará a análise mt da série time-lapse.
- Uma vez que o processamento do filme é concluído, uma mensagem "Seu filme (s) foram processados com sucesso" é exibido. Pressione "Ok",então "Salve".
- Agora é seguro sair do software de análise numérica. Os resultados do processamento do filme são armazenados em estruturas de subfolderas como arquivos m na pasta onde os arquivos brutos são armazenados.
5. Análise estatística da dinâmica do MT
- Importe os arquivos m em um programa de análise estatística preferido.
Nota: No nosso caso, primeiro importamos os arquivos em uma planilha padrão para torná-los legíveis. Os arquivos m contêm informações estatísticas (desvio mediano, médio e padrão) em diferentes parâmetros (por exemplo, velocidade de crescimento, dinâmica do MT). A lista detalhada dos parâmetros é dada no relatório técnico fornecido com a versão anterior do software, plusTipTracker50,52. Os arquivos m gerados também podem ser importados para outros softwares de processamento de dados. - Escolha o parâmetro "velocidade de crescimento média" e importá-lo em uma tabela para estatísticas e exibição. Insira as informações em outros parâmetros , (por exemplo, "dinâmica") seja em uma nova tabela ou em uma nova coluna da mesma tabela e enredo agrupados.
Access restricted. Please log in or start a trial to view this content.
Resultados
Seguindo o protocolo dado esboçado na figura 1A,o plasmid do pEB3-tdTomato foi expressado transitonte em pilhas assynchronously crescentes de HeLa. As células foram sincronizadas 48 h após a transfecção na prometaphase através do tratamento de DME (Figura 1B). Esta etapa garantiu que a medição da dinâmica do MT fosse feita sempre na mesma fase do ciclo da pilha. Os filmes de lapso de tempo foram processados e analisados com U-Track v2.2.0 como descrito ...
Access restricted. Please log in or start a trial to view this content.
Discussão
Aqui, descrevemos uma modificação de um método estabelecido pela primeira vez por Ertych et al.44. Junto com várias outras modificações, combinamos esta técnica de análise de dinâmica MT com imagens confocais de disco de fiação dupla. O uso do disco de fiação dupla melhora a resolução de MTs em crescimento, reduzindo a fototoxicidade36. Reduzimos ainda mais o photobleaching e os danos induzidos pela luz laser das células, mudando para um repórter fluorescen...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Agradecemos aos membros da Light Microscopy Facility, Max-Planck Institute of Experimental Medicine, por seus conselhos e apoio especializados.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
Referências
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados