需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
通过旋转磁盘显微镜在单极式斜接主轴中测量微管动力学
摘要
在这里,我们提出了一个强大而详细的微管动力学分析方法,在预元相中同步的细胞使用活细胞旋转磁盘共聚焦显微镜和基于MATLAB的图像处理。
摘要
我们描述了对确定活细胞中微管动力学的既定方法的修改。该协议基于微管正端(EB3标有tdTomato荧光蛋白)的遗传编码标记的表达,以及使用旋转盘共聚焦显微镜的高速、高分辨率活细胞成像。通过抑制线细胞中的中位体分离,实现细胞周期同步和微管密度的增加,并使用开源U-Track软件进行生长分析。使用明亮和红色转移荧光蛋白,结合较低的激光功率和减少曝光时间所需的旋转盘显微镜减少光毒性和光诱导伪影的可能性。这允许在相同的制备中成像更多细胞,同时在标准培养条件下将细胞保持在生长培养基中。由于分析以受监督的自动方式执行,因此结果在统计上是可靠的,可重现。
引言
微管(MTs)是几乎所有真核细胞和某些细菌1中发现的高度动态结构。与蛋白和中间丝一起,他们雕刻细胞骨架2,3。细胞分裂4,分子传输5,鞭拉击6,感觉周围环境通过原发性硅7,听力(基诺西)8,9,胚胎发生10,11,12,入侵和转移13,14,甚至记忆形成15,16,17,18,和许多其他过程主要依赖于MT。如果没有在生长(聚合)和收缩(去聚合)之间快速切换的非凡能力,那么,在所有这些事件中,如果MT的参与是不可能的。此属性被描述为动态不稳定19。MT动态性在许多病理条件下改变20,21,22。因此,确定此属性的性质可以帮助了解疾病机制及其后的治疗。
已经为MT动力学分析开发了一长串的方法,其中大多数都基于成像技术23。最初,宽场光学显微镜用于观察在体外24体中形成图布林聚合物。最终结合(EB)蛋白的发现,在MT加端聚集,并开发荧光标记蛋白质的方法,使得在宽场和共聚焦荧光显微镜25、26、27的活细胞中直接观察MT的行为成为可能。一个EB蛋白是最终结合蛋白3(EB3)28;通过过度表达和跟踪EB3融合到荧光蛋白,MT加端组装速率可以确定29,30。
共聚焦激光扫描荧光显微镜(CLSM)经常用于跟踪MT动力学。然而,这种成像技术带来了光毒性和光漂白的高风险,两个不良的过程活细胞和暗淡样品成像31。为了获得更好的信噪比,激光功率和曝光持续时间应足够高,同时不损坏样品,这需要牺牲分辨率以换取速度。CLSM的一个合适的替代方案是旋转磁盘显微镜32。这种成像模式基于使用Nipkow磁盘33,它包括一个带有一系列针孔的移动磁盘,并且与许多CLS显微镜同时成像同一样品34的工作相同。因此,来自激光的光将同时照亮样品中的多个区域,但会保持共聚焦性质。因此,Nipkow 磁盘允许获取类似于 CLSM 的图像,但速度更快,使用更少的激光功率。横川电机进一步改进了Nipkow圆盘,该盘引入了第二个圆盘,上面装有一系列微透镜,可单独将光线定向到各自的针孔中,进一步降低了光毒性和光漂白35。因此,旋转盘激光扫描显微镜成为活细胞成像的首选方法,并且它使得以高速31、36获得高信噪比的图像成为可能,这对解析来自快速增长的MT端信号至关重要。
MT 动态暂时不同。例如,线粒体MT比相间的37,38更动态。同样,即使在相同的细胞周期阶段,如米细胞炎39,40,也观察到生长速率和收缩的差异。因此,为了避免错误数据收集,MT 动力学的测量应限制在单元周期中的狭窄时间窗口。例如,通过用二甲基苯丙酮(DME)处理细胞,可以测量预元相中的MT动力学,这是一种抑制运动动蛋白Eg541并防止双极线轴42形成的单体模拟物。用Eg5抑制剂DME和其他单星衍生物抑制细胞在预元相不影响MT动力学43,44,45,这使得DME成为研究固定细胞和活细胞44MT动力学的有用工具。
在这里,我们将Ertych等人44描述的prometa相细胞中的MT动力学分析方法与双旋转磁盘成像相结合。该方法允许测量从单个焦平面收集的Prometa相细胞中的MT动力学,成像速率较高,但无光漂白和最小光毒性。此外,作为荧光报告员,我们使用串联二聚体番茄荧光蛋白(tdTomato),与绿色荧光蛋白(EGFP)相比,具有更高的亮度和光稳定性,并且以低能量光46激发。因此,tdTomato 需要较少的激光功率来激发,并且光毒性更少。总之,通过降低光毒性,提高MT动力学分析所需的分辨率和后处理,进一步完善了该方法。此外,我们通过将方法与其他同步技术相结合,为将来修改该方法奠定基础。
Access restricted. Please log in or start a trial to view this content.
研究方案
1. 赫拉细胞的种子
- 在磷酸盐缓冲盐水 (PBS) 中制备 2 mL 的 5 μg/mL 纤维化溶液,并将其加入 4 口井盖玻片 (#1.5) 的每个井中。在37°C和5%CO2下孵育幻灯片15分钟。
- 用Dulbecco的磷酸盐缓冲盐水(DPBS)冲洗异步生长的HeLa细胞,并在37°C下孵育胰蛋白酶-EDTA(0.05%:0.02%;w:v)。5分钟。停止酶反应添加罗斯威尔公园纪念研究所(RPMI)1640介质辅以10%热灭活胎儿小牛血清(FCS)在3:1 (v:v)比例添加胰蛋白酶-EDTA。
注:HeLa细胞在RPMI 1640介质中保持,在37°C和5%CO2下辅以10%热灭活FCS,一旦达到80-90%的汇合,则按照上述方式定期通过。 - 使用 Neubauer 腔室确定细胞浓度。以 1:1 (v:v) 比率将细胞悬浮液的 50 μL 等分与锥蓝色试孔混合,重新悬浮,并将悬浮液的 10 μL 转移到腔室中。只计算四个大方块内的锥形蓝负细胞(详情见Phelan等人47)。使用以下公式从计数的细胞数中派生细胞浓度:
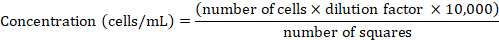
- 在300 x g下离心将细胞压离2分钟,用新鲜的RPMI 1640重新悬浮,以获得1 x 106细胞/mL。
- 从密室盖玻片中取出纤维,用DPBS洗井两次,每口井播种50,000个细胞。
- 将带细胞的室盖玻片返回培养箱,在37°C和5%CO2下生长24小时。
2. HeLa细胞中pEB3-tdtomato的表达
- 准备一个1.5 mL微离心管。对于每管,用转染缓冲液(水溶液中的合成产物)将2μg的pEB3-tdTomato48稀释至396μL的最终体积。
- 将4μL转染试剂(非脂质,含有聚乙烯酰胺)加入第一管,并立即涡旋混合物正好10s。
- 用微离心机短暂地旋转管,并在室温 (RT) 下孵育 10 分钟。
- 从培养箱中取出 HeLa 细胞。顺流,将100μL的转染混合物加入4个井室盖玻片的每个井中,并将细胞返回到孵化器。
- 在37°C和5%CO2孵育4小时后,用新鲜生长培养基补充细胞,并在37°C和5%CO2下孵育至少24小时。
注:有必要优化每种细胞类型的转染条件。表达级别需要足够低,以便识别单个 MT 增长端。或者,在实验中可以使用一条能稳定表达EB3-tdTomato的细胞系;这将减少来自同一制剂的细胞之间和细胞之间的EB3-tdTomato表达水平的变异性49。
3. pEB3-tdtomato_表达HeLa细胞的同步和活细胞成像
- 在不含苯酚红色的Dulbeco改性鹰介质(DMEM)中制备2.5 μM二甲基苯丙胺(DME)溶液,辅以10%FCS和2 mM L-谷氨酰胺或替代谷氨酰胺供应。
- 用含有2.5μM DME的生长培养基500μL替换室盖玻片中的生长培养基,并在37°C和5%CO2下孵育细胞。
- 使用DME孵育3.5小时后,将细胞转移到显微镜上,将室盖玻片安装到带有深色面板的环境室中,在37°C和5%CO2下进行成像,并进一步孵育,直到总孵育时间为4小时。
注:保持37°C的温度无波动对实验至关重要。 - 在配备 100x 1.49 N.A. 油浸物、双旋转盘共焦系统和可靠自动对焦系统的倒置显微镜上执行延时成像,以便持续维护焦平面。定义成像参数,如下所示。
注:我们使用电子倍增电荷耦合器件相机(EM-CCD)。- 对于 EB3-tdTomato 激发,请使用 561 nm 激光线,曝光时间为 200 ms。通过四联带通(405、488、561、640 nm)二色镜和 600/52 nm 发射滤波器收集发射光。
注:可以为每个成像单元调整激光功率,以防止图像饱和。在此处给出的所有延时电影中,激光功率设置为 5.3 mW。 - 在与单极线轴中心相对应的 Z 平面中查找预相和焦点的细胞。每 0.5 秒获取一次图像,总共 1 分钟,曝光之间没有分箱和照明。
- 对于 EB3-tdTomato 激发,请使用 561 nm 激光线,曝光时间为 200 ms。通过四联带通(405、488、561、640 nm)二色镜和 600/52 nm 发射滤波器收集发射光。
4. 使用 U-Track v2.2.0 分析 MT 动力学
- 要分析 MT 动力学,需要一个数字计算环境软件(例如 MATLAB)。
注:对软件的基本了解足以进行分析。全面的帮助材料和教程可在开发人员的网站 (https://uk.mathworks.com/products/matlab/getting-started.html) 上找到。 - 下载 (https://github.com/DanuserLab/u-track) 并安装开源 U-Track v2.2.0 软件,按照"Readme_u-track.pdf"文件中给出的详细说明进行操作。
- 启动数值分析软件,并将带有子文件夹的 U-Track v2.2.0 文件夹添加到软件搜索路径中。
- 从命令窗口调用"电影选择GUI"。这将打开一个对话框窗口,从中可以导入显微镜上图像采集软件生成的原始文件(补充图1,图2,图3,图4)。
注:U-Track 软件与其他图像数据格式兼容。它使用生物格式,它识别不同的生命科学数据格式53。 - 从元数据自动读取每个图像的大小。手动输入目标的数字孔径(本例中为 1.49)和用于成像的时间间隔 (0.5 s)(补充图 1B)。此外,还可以提供有关激发波长、荧光和暴露时间的信息,但它们对于进一步分析并不重要。
- 加载所有图像后,通过选择"保存为影片列表",将输入的延时序列另存为影片列表。在对话框的右侧选择"U-Track"选项,然后按"继续"(补充图1C)。
注:这些值针对 HeLa 细胞进行了优化。如果切换到其他单元格线,应再次定义值。或者,使用软件开发人员建议的设置。每个参数的详细说明以及如何定义它们,请参阅与该软件的早期版本一起提供的技术报告,以及TipTracker50。 - 从弹出窗口中选择"微管加端",然后按"确定"(补充图1C)。新的对话窗口允许确定分析的三个步骤的参数(补充图 1D),即检测、跟踪和跟踪分析。
- 在步骤 1 中选择"设置",从下拉菜单中选择"彗星探测"作为检测方法(补充图 2B)。
- 在新的对话窗口中定义高斯滤波器差异的参数和分水岭分割如下(补充图2C):用于检测的掩码过程= 无;低通高斯标准差 = 1 像素;高通高斯标准差 = 3 像素;最小阈值 = 3 个标准偏差;阈值步长大小 = 0.25 标准偏差。选择"对所有影片应用设置"和"应用"。
- 在步骤 2 中,链接、间隙闭合、合并和拆分以及 Kalman 筛选器函数的参数以三个步骤定义,相应地以粉红色、绿色和蓝色突出显示(补充图 3B)。对于这些步骤,选择"微管加端动力学"和"设置"选项分别定义补充图 3C+E中所示的值。
- 对于尺寸问题,请从下拉菜单中选择"2"。使用最大间隙关闭 = 5 帧;从第一步开始的轨道段最小长度 = 3 帧。与之前一样,选择"对所有影片应用设置",然后单击"应用"。
- 在分析的第 3 步中,检测到的 MT 磁道被分类 (补充图 4)。作为跟踪分析方法,选择"微管动力学分类",并通过补充图4B,C所示的"设置"按钮定义参数。之后,选择"对所有影片应用设置"框,然后单击"应用"。
- 定义所有参数后,从"控制面板+U-Track"窗口(补充图 1D)中选择"对所有影片应用检查/取消选中"和"运行所有影片"框并按"运行"。这将启动延时系列的 MT 分析。
- 完成影片处理后,将显示一条消息"您的影片已成功处理"。按"确定",然后"保存"。
- 现在,可以安全地退出数值分析软件。影片处理的结果存储在子文件夹结构中,作为 m 文件存储在存储原始文件的文件夹中。
5. MT动力学的统计分析
- 将 m 文件导入首选统计分析程序。
注:在我们的案例中,我们首先在标准电子表格中导入文件,使其可读。m-file 包含不同参数(例如,增长速度、MT 动态性)的统计信息(中位数、均值和标准偏差)。参数的详细列表在该软件的早期版本提供的技术报告中给出,加上TipTracker50,52。生成的 m 文件也可以导入到其他数据处理软件中。 - 选择"增长速度平均值"参数并将其导入表进行统计和显示。在新表或同一分组表和绘图的新列中输入有关其他参数的信息(例如,"动态性")。
Access restricted. Please log in or start a trial to view this content.
结果
按照图1A中概述的给定协议,pEB3-tdTomato质粒在异步生长的HeLa细胞中瞬时表达。通过DME治疗在临元相转染后48小时与细胞同步(图1B)。此步骤可确保 MT 动力学的测量始终在细胞周期的同一阶段进行。延时电影被进一步处理和分析与U-Track v2.2.0,如其补充文件50,51,52所述。虽然加端?...
Access restricted. Please log in or start a trial to view this content.
讨论
在这里,我们描述了Ertych等人首先建立的方法的修改。除了其他几种修改之外,我们还将这一MT动力学分析技术与双旋转磁盘共聚焦成像相结合。使用双纺盘提高了增长的MT的分辨率,同时降低了光毒性36。通过切换到长波长荧光报告器,我们进一步减少了光漂白和激光引起的细胞损伤。与EGFP46相比,tdTomato荧光蛋白具有更高的光稳定性和亮度?...
Access restricted. Please log in or start a trial to view this content.
披露声明
作者没有什么可透露的。
致谢
我们感谢马克斯-普朗克实验医学研究所光显微镜设施的成员,感谢他们的专家建议和支持。
Access restricted. Please log in or start a trial to view this content.
材料
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
参考文献
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。