Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Medición de microtúbulos dinámicos mediante microscopía de disco giratorio en husillos mitoticos Monopolar
En este artículo
Resumen
Aquí presentamos un método robusto y detallado de análisis de dinámica de microtúbulos en células sincronizadas en prometiafase utilizando microscopía confocal de disco giratorio de células vivas y procesamiento de imágenes basado en MATLAB.
Resumen
Describimos una modificación de un método establecido para determinar la dinámica de microtúbulos en células vivas. El protocolo se basa en la expresión de un marcador codificado genéticamente para los extremos positivos de los microtúbulos (EB3 etiquetado con proteína fluorescente tdTomato) y la imagen de células vivas de alta velocidad y alta resolución mediante microscopía confocal de disco giratorio. La sincronización del ciclo celular y el aumento de la densidad de los microtúbulos se logran mediante la inhibición de la separación centrosómica en células mitoticas, y el análisis del crecimiento se realiza utilizando el software U-Track de código abierto. El uso de una proteína fluorescente brillante y de cambio de rojo, en combinación con la menor potencia láser y el tiempo de exposición reducido necesario para la microscopía de disco giratorio reducen la fototoxicidad y la probabilidad de artefactos inducidos por la luz. Esto permite tomar imágenes de un mayor número de células en la misma preparación mientras se mantienen las células en un medio de crecimiento bajo condiciones de cultivo estándar. Debido a que el análisis se realiza de forma automática supervisada, los resultados son estadísticamente robustos y reproducibles.
Introducción
Los microtúbulos (MT) son estructuras altamente dinámicas que se encuentran en prácticamente todas las células eucariotas y en algunas bacterias1. Junto con la actina y los filamentos intermedios, esculpen el citoesqueleto2,3. División celular4, transporte de moléculas5, flagelante golpeando6, la sensación del entorno circundante a través del cilium primario7, audición (kinocilium)8,9, embriogénesis10,11,12, invasión y metástasis13,14, e incluso la formación de la memoria15,16,17,18, y muchos otros procesos se basan principalmente en los TM. La participación de los MT en todos estos eventos sería imposible sin su notable capacidad de cambiar rápidamente entre el crecimiento (polimerización) y la contracción (despolimerización). Esta propiedad se describe como inestabilidad dinámica19. La dinámica MT se altera en muchas condiciones patológicas20,21,22. Por lo tanto, determinar la naturaleza de esta propiedad puede ayudar a entender los mecanismos de la enfermedad y posteriormente su tratamiento.
Se ha desarrollado una larga lista de métodos para el análisis de dinámica MT, la mayoría de los cuales se basan en técnicas de imagen23. Inicialmente, se utilizaron microscopios de luz de campo amplio para observar la formación de polímeros de tubulina in vitro24. El descubrimiento de proteínas de unión final (EB) que se acumulan en MT plus-ends y el desarrollo de métodos para etiquetar fluorescentes proteínas hicieron posible observar el comportamiento de los MT directamente en células vivas con microscopios de fluorescencia confocal esdecir de campo ancho y confocal25,26,27. Una proteína EB es proteína de unión final 3 (EB3)28; mediante la sobreexpresión y el seguimiento de EB3 fusionado con una proteína fluorescente, MT plus-end tasas de montaje se pueden determinar29,30.
La microscopía de fluorescencia de escaneo láser confocal (CLSM) se utiliza con frecuencia para seguir la dinámica de MT. Sin embargo, esta técnica de diagnóstico por imágenes supone un alto riesgo de fototoxicidad y fotoblanqueo, dos procesos indeseables para las imágenes de células vivas y muestras tenues31. Con el fin de obtener una mejor relación señal-ruido, la potencia del láser y la duración de la exposición deben ser lo suficientemente altas mientras que no dañan las muestras, y esto requiere sacrificar la resolución a cambio de la velocidad. Una alternativa adecuada a CLSM es la microscopía de disco giratorio32. Esta modalidad de imagen se basa en el uso de un disco Nipkow33,que consiste en un disco en movimiento que lleva una serie de agujeros, y funciona de forma equivalente a muchos microscopios CLS que toma imágenes de la misma muestra simultáneamente34. Por lo tanto, la luz del láser iluminará varias regiones de la muestra simultáneamente, pero conservará la naturaleza confocal. El disco Nipkow, por lo tanto, permite obtener imágenes similares a CLSM pero más rápido y utilizando menos potencia láser. El disco nipkow fue mejorado aún más por Yokogawa Electric, que introdujo un segundo disco con una serie de microlentes que dirigen individualmente la luz en un agujero respectivo, reduciendo aún más la fototoxicidad y el fotoblanqueo35. Por lo tanto, la microscopía de escaneo láser de disco giratorio se convirtió en un método de elección para la creación de imágenes de células en vivo, y permite obtener imágenes con alta relación señal-ruido a una alta velocidad31,36, que es crucial para resolver señales como las de los extremos MT de rápido crecimiento.
La dinámica de MT difiere temporalmente. Por ejemplo, los MT mitoticos son más dinámicos que los interfásicos37,38. Del mismo modo, se han observado diferencias en la tasa de crecimiento y la contracción incluso dentro de la misma fase del ciclo celular, como la mitosis39,40. Por lo tanto, para evitar la recopilación de datos falsos, la medición de la dinámica de MT debe limitarse a una ventana de tiempo estrecha durante el ciclo de celda. Por ejemplo, la medición de la dinámica MT en prometafase se puede lograr mediante el tratamiento de las células con dimetilenasnía (DME), un análogo monastrol que inhibe la quinesina motora Eg541 y evita la formación del husillo mitotico bipolar42. La inhibición de células en prometafase con inhibidor de Eg5 DME y otros derivados monastrolno no afecta a la dinámica MT43,44,45, lo que hace de DME una herramienta útil para el estudio de la dinámica MT tanto en células fijas como vivas44.
Aquí combinamos el método de análisis de dinámica MT en células prometifásicas descrito por Ertych et al.44 con imágenes de disco giratorios duales. Este método permite medir la dinámica de MT en células de prometafase recogidas de un solo plano focal con una mayor tasa de diagnóstico por imágenes, pero sin fotoblanqueo y fototoxicidad mínima. Además, como reportero fluorescente, utilizamos dimer tándem Proteína fluorescente de tomate (tdTomato) que ha mejorado el brillo y la fotoestabilidad en comparación con la proteína fluorescente verde (EGFP) y se excita con menor luz de energía46. Por lo tanto, tdTomato requiere menos potencia láser para la excitación y es menos fototóxico. En conjunto, mejoramos aún más el método reduciendo la fototoxicidad y mejorando la resolución y el postprocesamiento necesarios para el análisis de dinámica MT. Además, creamos una base para futuras modificaciones del método combinándolo con otras técnicas de sincronización.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Sembrado de células helancos
- Preparar 2 ml de solución de fibronectina de 5 g/ml en solución salina tamponada de fosfato (PBS) y añadir 450 ml de ella en cada pocal de un cubreobjetos de 4 cámaras bien (#1.5). Incubar el tobogán durante 15 min a 37oC y 5%CO2.
- Enjuague las células de HeLa de crecimiento asíncrono con solución salina tamponada de fosfato de Dulbecco (DPBS) e incubar con trippsina-EDTA (0,05%: 0,02%; w:v) durante 5 min a 37 oC. Detener la reacción enzimática mediante la adición de Roswell Park Memorial Institute (RPMI) 1640 medio complementado con 10% suero de becerro fetal inactivado por calor (FCS) en 3:1 (v:v) relación de trippsin-EDTA añadido.
NOTA: Las células HeLa se mantuvieron en el medio RPMI 1640 complementado con 10% de FCS inactivado por calor a 37 oC y 5% de CO2 y se pasaron rutinariamente una vez que alcanzaron una confluencia del 80-90% como se describió anteriormente. - Determine la concentración celular utilizando una cámara Neubauer. Mezcle una alícuota de 50 l de la suspensión celular con el azul trypan a una relación de 1:1 (v:v), vuelva a suspender y transfiera 10 l de la suspensión a la cámara. Cuente sólo las celdas de trypan azul-negativas dentro de los cuatro cuadrados grandes (para más detalles ver Phelan et al.47). Derive la concentración de celdas del número de celda contada utilizando la siguiente fórmula:
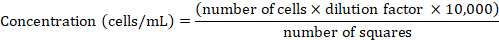
- Resuspender las células por centrifugación a 300 x g durante 2 min. Resuspender con RPMI 1640 fresco para obtener 1 x 106 células/ml.
- Retire la fibronectina del cubreobjetos con cámara, lave los pozos dos veces con DPBS y la semilla 50.000 células por poca.
- Devolver la cubierta con cámara con las células a la incubadora y hacerlas crecer durante 24 h a 37oC y 5%CO2.
2. Expresión de pEB3-tdTomato en células HeLa
- Prepare un tubo de microcentrífuga de 1,5 ml. Para cada tubo, diluir 2 g de pEB3-tdTomato48 con tampón de transfección (producto sintético en solución acuosa) a un volumen final de 396 l.
- Añadir 4 l de reactivo de transfección (no lipídico, que contiene polietilenimina) al primer tubo, y vórtice la mezcla inmediatamente durante exactamente 10 s.
- Gire brevemente por el tubo con una microcentrífuga e incubar a temperatura ambiente (RT) durante 10 minutos.
- Retire las células de HeLa de la incubadora. En el sentido de gota, añadir 100 l de la mezcla de transfección a cada pocal de un cubreobjetos de 4 bien encámaras, y devolver las células a la incubadora.
- Después de 4 h de incubación a 37oC y 5% CO2,complementar las células con medio de crecimiento fresco e incubar durante al menos 24 h a 37oC y 5%CO2.
NOTA: Es necesario optimizar las condiciones de transfección para cada tipo de celda. Los niveles de expresión deben ser lo suficientemente bajos como para permitir la identificación de extremos de crecimiento MT únicos. Alternativamente, una línea celular que expresa establemente EB3-tdTomato se puede utilizar en los experimentos; esto reduciría la variabilidad en los niveles de expresión de EB3-tdTomato entre preparaciones y entre células de la misma preparación49.
3. Sincronización e imágenes de células vivas de células Demitovares pEB3-tdTomato-que expresan
- Preparar una solución de 2,5 m de dimetilenasonión (DME) en el medio de águila modificado (DMEM) libre de fenol-rojo complementado con 10% FCS y 2 mM de L-glutamina o un suministro alternativo de glutamina.
- Sustituya el medio de crecimiento en la cubierta con cámara por 500 ml del medio de crecimiento que contiene 2,5 M DME e incubar las células a 37 oC y 5% deCO2.
- Después de 3,5 h de incubación con DME, transferir las células al microscopio, montar la cubierta de cámara en una cámara ambiental con paneles oscuros para la toma de imágenes a 37 oC y 5% de CO2,e incubar aún más hasta que el tiempo total de incubación sea de 4 h.
NOTA: El mantenimiento de la temperatura a 37 oC sin fluctuaciones es crucial para el experimento. - Realice las imágenes de lapso de tiempo en un microscopio invertido equipado con un objetivo de inmersión de aceite de 100x 1.49 N.A., un sistema confocal de disco giratorio dual y un sistema de enfoque automático confiable para el mantenimiento continuo del plano focal. Defina los parámetros de imagen de la siguiente manera.
NOTA: Utilizamos una cámara de dispositivo acoplado de carga de multiplicación electrónica (EM-CCD).- Para la excitación EB3-tdTomato, utilice una línea láser de 561 nm con un tiempo de exposición de 200 ms. Recoja la luz emitida a través de un faro cuádruple (405, 488, 561, 640 nm) espejo dichroico y un filtro de emisión de 600/52 nm.
NOTA: La potencia láser se puede ajustar para cada celda con imágenes para evitar la saturación de la imagen. En todas las películas de lapso de tiempo dadas aquí la potencia del láser se estableció en 5,3 mW. - Encuentre una celda en la fase y el foco en el plano Z correspondiente al centro del husillo mitético monopolar. Adquiere imágenes cada 0,5 s durante un total de 1 min sin binning y sin iluminación entre las exposiciones.
- Para la excitación EB3-tdTomato, utilice una línea láser de 561 nm con un tiempo de exposición de 200 ms. Recoja la luz emitida a través de un faro cuádruple (405, 488, 561, 640 nm) espejo dichroico y un filtro de emisión de 600/52 nm.
4. Análisis de LA dinámica MT usando U-Track v2.2.0
- Para analizar la dinámica MT se requiere un software de entorno de computación numérica (por ejemplo, MATLAB).
NOTA: La comprensión básica del software es suficiente para el análisis. El material de ayuda completo y los tutoriales están disponibles en el sitio web del desarrollador (https://uk.mathworks.com/products/matlab/getting-started.html). - Descargue (https://github.com/DanuserLab/u-track) e instale el software de código abierto U-Track v2.2.0 siguiendo las instrucciones detalladas dadas en el archivo "Readme_u-track.pdf"50,51,52.
- Inicie el software de análisis numérico y agregue la carpeta U-Track v2.2.0 con subcarpetas en la ruta de búsqueda de software.
- Desde la ventana de comandos llame a "movieSelectorGUI". Esto abre una ventana de diálogo desde la que se pueden importar los archivos sin procesar generados por el software de adquisición de imágenes en el microscopio (Figura Suplementaria 1, Figura 2, Figura 3, Figura 4).
NOTA: El software U-Track es compatible con otros formatos de datos de imagen. Utiliza Bio-Formats, que reconoce diferentes formatos de datos de ciencias de la vida53. - El tamaño de cada imagen se lee automáticamente de los metadatos. Introduzca manualmente la apertura numérica del objetivo (en este caso 1,49) y el intervalo de tiempo (0,5 s) utilizado para la toma deimágenes (Figura suplementaria 1B). Además, también se puede proporcionar información sobre la longitud de onda de excitación, el fluoróforo y el tiempo de exposición, pero no son críticos para un análisis posterior.
- Una vez cargadas todas las imágenes, guarde la serie de lapso de tiempo introducida como una lista de películas seleccionando la"Guardar como listade películas". En el lado derecho de la ventana de diálogo seleccione la opción "U-Track" y pulse "Continue" (Supplementary Figure 1C).
NOTA: Los valores están optimizados para las celdas HeLa. Si cambia a una línea de celda diferente, los valores deben definirse de nuevo. Como alternativa, utilice la configuración recomendada por los desarrolladores de software. La explicación detallada de cada uno de los parámetros y cómo deben definirse se puede encontrar en el informe técnico proporcionado con la versión anterior del software, plusTipTracker50. - En la ventana emergente seleccione "Microtubule Plus-Ends" y pulse "Ok" (Figura complementaria 1C). La nueva ventana de diálogo permite determinar los parámetros para los tres pasos del análisis(Figura suplementaria 1D),que son la detección, el seguimiento y el análisis de seguimiento.
- En el paso 1 elija "Configuración" y en un menú desplegable seleccione "Detección de cometas" como método de detección(Figura suplementaria 2B).
- A partir de la nueva ventana de diálogo definir los parámetros para la diferencia del filtro gaussiano y la segmentación de cuenca hidrográfica de la siguiente manera (Figura suplementaria 2C): Proceso de máscara que se utilizará para la detección - Ninguno; Desviación estándar gaussiana de paso bajo: 1 píxel; Desviación estándar gaussiana de paso alto: 3 píxeles; Umbral mínimo: 3 desviaciones estándar; Tamaño del paso de umbral: 0,25 desviaciones estándar. Seleccione"Aplicar configuración a todas las películas"y"Aplicar".
- En el paso 2, los parámetros para vincular, cerrar brechas, fusionar y dividir y las funciones de filtro Kalman se definen en tres pasos resaltados en rosa, verde y azul, en consecuencia (Figura complementaria 3B). Para estos pasos, seleccione la opción "Microtubule Plus-end Dynamics" y en la opción "Setting" (Configuración) define los valores como se indica en la Figura suplementaria 3C–E, respectivamente.
- Para problemas con la dimensionalidad, elija "2" en el menú desplegable. Utilice La brecha máxima para cerrar 5 fotogramas; Longitud mínima de los segmentos de pista desde el primer paso 3 fotogramas. Como antes, seleccione"Aplicar configuración a todas las películas"y haga clic en"Aplicar".
- En el paso 3 del análisis, se clasifican las pistas MT detectadas(Figura Suplementaria 4). Como método de análisis de pista, elija "Clasificación dinámica demicrotúbulos"y defina los parámetros a través del botón "Configuración"como se indica en la figura complementaria 4B,C. Después de eso, elija el cuadro"Aplicar configuración a todas las películas"y haga clic en"Aplicar".
- Una vez definidos todos los parámetros, desde la ventana " Panel decontrol–U-Track"(Figura suplementaria 1D)seleccione las casillas"Aplicar marca/desmarque a todas las películas"y"Ejecutar todas las películas"y pulse "Ejecutar". Esto iniciará el análisis MT de la serie de lapso de tiempo.
- Una vez completado el procesamiento de la película, se muestra el mensaje "Sus películas se han procesado correctamente". Pulse "Ok", luego "Guardar".
- Ahora es seguro salir del software de análisis numérico. Los resultados del procesamiento de películas se almacenan en estructuras de subcarpetas como archivos m en la carpeta donde se almacenan los archivos sin procesar.
5. Análisis estadístico de la dinámica MT
- Importe los archivos m a un programa de análisis estadístico preferido.
NOTA: En nuestro caso, primero importamos los archivos en una hoja de cálculo estándar para que sean legibles. Los archivos m contienen información estadística (mediana, media y desviación estándar) sobre diferentes parámetros (por ejemplo, velocidad de crecimiento, dinámica MT). La lista detallada de los parámetros se da en el informe técnico proporcionado con la versión anterior del software, plusTipTracker50,52. Los archivos m generados también se pueden importar a otro software de procesamiento de datos. - Elija el parámetro "media de velocidad de crecimiento" e impórtelo en una tabla para estadísticas y visualización. Introduzca la información sobre otros parámetros (por ejemplo, "dinámica") en una tabla nueva o en una nueva columna de la misma tabla y trazado agrupados.
Access restricted. Please log in or start a trial to view this content.
Resultados
Siguiendo el protocolo dado descrito en la Figura 1A, el plásmido pEB3-tdTomato se expresó transitoriamente en células HeLa de crecimiento asíncrono. Las células se sincronizaron 48 horas después de la transfección en prometafase a través del tratamiento DME(Figura 1B). Este paso aseguró que la medición de la dinámica MT siempre se realizaba en la misma fase del ciclo celular. Las películas de lapso de tiempo se procesaron y analizaron con U-Track v2...
Access restricted. Please log in or start a trial to view this content.
Discusión
Aquí, describimos una modificación de un método establecido por primera vez por Ertych et al.44. Junto con varias otras modificaciones, combinamos esta técnica de análisis de dinámica MT con imágenes confocales de disco giratorio dual. El uso del disco giratorio dual mejora la resolución de los TM en crecimiento al tiempo que reduce la fototoxicidad36. Reducimos aún más el fotoblanqueo y el daño inducido por la luz láser de las células cambiando a un reportero ...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a los miembros del Light Microscopy Facility, Instituto Max-Planck de Medicina Experimental, por su asesoramiento y apoyo experto.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Dimethylenastron | Merck | 324622 | |
| DMEM w/o phenol red | Gibco | 31053-28 | |
| DPBS | Gibco | 14190-094 | |
| Fetal bovine serum | Biochrom | S0415 | |
| Fibronectin Bovine Plasma | Merck | F4759 | Sterile powder |
| GlutaMAX | Gibco | 35050-038 | Stable glutamine substitutive |
| jetPRIME | Polyplus | 114-15 | |
| EB3-TdTomato | Addgene | plasmid #50708 | |
| RPMI 1640 | Gibco | 61870-010 | |
| Trypan Blue | Merck | T8154-20ML | |
| Trypsin/EDTA solution | Biochrom | L2143 | 0.05%/0.02 % w/o calcium and magnesium |
| µ-slide | Ibidi | 80426 | 4-well slide with #1.5 coverslip |
| Eclipse Ti Inverted microscope | Nikon | NA | |
| Objective | Nikon | MRD01991 | CFI Apo TIRF 100xC Oil |
| ACAL Laser Excahnger | Nikon | Laser box. 405, 458, 488, 514, 561 and 647 nm | |
| Spinning disk module | Andor | CSU-W | |
| Camera | Andor | iXon Ultra 888 | |
| Environmental Chamber | Okolab | Dark chamber equipped with CO2 supply, temperature control and humidifier | |
| HeLa Cells | DSMZ | ACC-57 | |
| NIS Elements v4 | Nikon | Spinning disk microscope. Acquisition Software | |
| MATLAB | Mathworks | Computing environment | |
| Prism 8 | GraphPad | Statistical analysis and display software |
Referencias
- Erickson, H. P. Evolution of the cytoskeleton. Bioessays. 29 (7), 668-677 (2007).
- Pollard, T. D., Goldman, R. D. Overview of the Cytoskeleton from an Evolutionary Perspective. Cold Spring Harbor Perspectives in Biology. 10 (7), (2018).
- Wade, R. H. On and around microtubules: an overview. Molecular Biotechnology. 43 (2), 177-191 (2009).
- Forth, S., Kapoor, T. M. The mechanics of microtubule networks in cell division. Journal of Cell Biology. 216 (6), 1525-1531 (2017).
- Franker, M. A., Hoogenraad, C. C. Microtubule-based transport - basic mechanisms, traffic rules and role in neurological pathogenesis. Journal of Cell Science. 126, Pt 11 2319-2329 (2013).
- Lindemann, C. B., Lesich, K. A. Flagellar and ciliary beating: the proven and the possible. Journal of Cell Science. 123, Pt 4 519-528 (2010).
- Wheway, G., Nazlamova, L., Hancock, J. T. Signaling through the Primary Cilium. Frontiers in Cell and Developmental Biology. 6, 8(2018).
- Falk, N., Losl, M., Schroder, N., Giessl, A. Specialized Cilia in Mammalian Sensory Systems. Cells. 4 (3), 500-519 (2015).
- Spoon, C., Grant, W. Biomechanical measurement of kinocilium. Methods in Enzymology. 525, 21-43 (2013).
- Zenker, J., et al. A microtubule-organizing center directing intracellular transport in the early mouse embryo. Science. 357 (6354), 925-928 (2017).
- Goldstein, B. Embryonic polarity: a role for microtubules. Current Biology. 10 (22), 820-822 (2000).
- Uchida, S., Shumyatsky, G. P. Deceivingly dynamic: Learning-dependent changes in stathmin and microtubules. Neurobiology of Learning and Memory. 124, 52-61 (2015).
- Fife, C. M., McCarroll, J. A., Kavallaris, M. Movers and shakers: cell cytoskeleton in cancer metastasis. British Journal of Pharmacology. 171 (24), 5507-5523 (2014).
- Bouchet, B. P., Akhmanova, A. Microtubules in 3D cell motility. Journal of Cell Science. 130 (1), 39-50 (2017).
- Dent, E. W. Of microtubules and memory: implications for microtubule dynamics in dendrites and spines. Molecular Biology of the Cell. 28 (1), 1-8 (2017).
- Craddock, T. J., Tuszynski, J. A., Hameroff, S. Cytoskeletal signaling: is memory encoded in microtubule lattices by CaMKII phosphorylation. PLOS Computational Biology. 8 (3), (2012).
- Smythies, J. Off the beaten track: the molecular structure of long-term memory: three novel hypotheses-electrical, chemical and anatomical (allosteric). Frontiers in Integrative Neuroscience. 9, 4(2015).
- Kaganovsky, K., Wang, C. Y. How Do Microtubule Dynamics Relate to the Hallmarks of Learning and Memory. Journal of Neuroscience. 36 (22), 5911-5913 (2016).
- Mitchison, T., Kirschner, M. Dynamic instability of microtubule growth. Nature. 312 (5991), 237-242 (1984).
- Dubey, J., Ratnakaran, N., Koushika, S. P. Neurodegeneration and microtubule dynamics: death by a thousand cuts. Frontiers in Cellular Neuroscience. 9, 343(2015).
- Parker, A. L., Kavallaris, M., McCarroll, J. A. Microtubules and their role in cellular stress in cancer. Frontiers in Oncology. 4, 153(2014).
- Honore, S., Pasquier, E., Braguer, D. Understanding microtubule dynamics for improved cancer therapy. Cell and Molecular Life Sciences. 62 (24), 3039-3056 (2005).
- Straube, A. Methods in Molecular Biology. , Humana Press. Totowa, NJ. (2011).
- Budde, P. P., Desai, A., Heald, R. Analysis of microtubule polymerization in vitro and during the cell cycle in Xenopus egg extracts. Methods. 38 (1), 29-34 (2006).
- Gierke, S., Kumar, P., Wittmann, T. Analysis of microtubule polymerization dynamics in live cells. Methods in Cell Biology. 97, 15-33 (2010).
- Matov, A., et al. Analysis of microtubule dynamic instability using a plus-end growth marker. Nature Methods. 7 (9), 761-768 (2010).
- Bailey, M., Conway, L., Gramlich, M. W., Hawkins, T. L., Ross, J. L. Modern methods to interrogate microtubule dynamics. Integrative Biology (Camb). 5 (11), 1324-1333 (2013).
- Galjart, N. Plus-end-tracking proteins and their interactions at microtubule ends. Current Biology. 20 (12), 528-537 (2010).
- Stepanova, T., et al. Visualization of microtubule growth in cultured neurons via the use of EB3-GFP (end-binding protein 3-green fluorescent protein). Journal of Neuroscience. 23 (7), 2655-2664 (2003).
- Zwetsloot, A. J., Tut, G., Straube, A. Measuring microtubule dynamics. Essays in Biochemistry. 62 (6), 725-735 (2018).
- Bayguinov, P. O., et al. Modern Laser Scanning Confocal Microscopy. Current Protocols in Cytometry. 85 (1), 39(2018).
- Nakano, A. Spinning-disk confocal microscopy -- a cutting-edge tool for imaging of membrane traffic. Cell Structure and Function. 27 (5), 349-355 (2002).
- Elektrisches teleskop. Germany patent. , (1884).
- Yin, S., Lu, G., Zhang, J., Yu, F. T., Mait, J. N. Kinoform-based Nipkow disk for a confocal microscope. Applied Optics. 34 (25), 5695-5698 (1995).
- Nipkow disk for confocal optical scanner. European patent application. , EP92114750A (1992).
- Oreopoulos, J., Berman, R., Browne, M. Spinning-disk confocal microscopy: present technology and future trends. Methods in Cell Biology. 123, 153-175 (2014).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. M., Wadsworth, P. Cell cycle-dependent changes in microtubule dynamics in living cells expressing green fluorescent protein-alpha tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Rusan, N. M., Fagerstrom, C. J., Yvon, A. -M. C., Wadsworth, P. Cell Cycle-Dependent Changes in Microtubule Dynamics in Living Cells Expressing Green Fluorescent Protein-α Tubulin. Molecular Biology of the Cell. 12 (4), 971-980 (2001).
- Liu, D., Davydenko, O., Lampson, M. A. Polo-like kinase-1 regulates kinetochore-microtubule dynamics and spindle checkpoint silencing. Journal of Cell Biology. 198 (4), 491-499 (2012).
- Maiato, H., Sunkel, C. E. Kinetochore-microtubule interactions during cell division. Chromosome Research. 12 (6), 585-597 (2004).
- Muller, C., et al. Inhibitors of kinesin Eg5: antiproliferative activity of monastrol analogues against human glioblastoma cells. Cancer Chemotherrapy and Pharmacology. 59 (2), 157-164 (2007).
- Mayer, T. U., et al. Small molecule inhibitor of mitotic spindle bipolarity identified in a phenotype-based screen. Science. 286 (5441), 971-974 (1999).
- Kapoor, T. M., Mayer, T. U., Coughlin, M. L., Mitchison, T. J. Probing spindle assembly mechanisms with monastrol, a small molecule inhibitor of the mitotic kinesin Eg5. The Journal of Cell Biology. 150 (5), 975-988 (2000).
- Ertych, N., et al. Increased microtubule assembly rates influence chromosomal instability in colorectal cancer cells. Nature Cell Biology. 16 (8), 779-791 (2014).
- Brito, D. A., Yang, Z., Rieder, C. L. Microtubules do not promote mitotic slippage when the spindle assembly checkpoint cannot be satisfied. The Journal of Cell Biology. 182 (4), 623-629 (2008).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. Journal of Cell Science. 120 (24), 4247-4260 (2007).
- Phelan, M. C., Lawler, G. Cell Counting. Current Protocols in Cytometry. 00 (1), 3(1997).
- Merriam, E. B., et al. Synaptic regulation of microtubule dynamics in dendritic spines by calcium, F-actin, and drebrin. Journal of Neuroscience. 33 (42), 16471-16482 (2013).
- Samora, C. P., et al. MAP4 and CLASP1 operate as a safety mechanism to maintain a stable spindle position in mitosis. Nature Cell Biology. 13 (9), 1040-1050 (2011).
- Applegate, K. T., et al. plusTipTracker: Quantitative image analysis software for the measurement of microtubule dynamics. Journal of Structural Biology. 176 (2), 168-184 (2011).
- Jaqaman, K., et al. Robust single-particle tracking in live-cell time-lapse sequences. Nature Methods. 5 (8), 695-702 (2008).
- Stout, A., D'Amico, S., Enzenbacher, T., Ebbert, P., Lowery, L. A. Using plusTipTracker Software to Measure Microtubule Dynamics in Xenopus laevis Growth Cones. Journal of Visualized Experiments. , e52138(2014).
- Linkert, M., et al. Metadata matters: access to image data in the real world. The Journal of Cell Biology. 189 (5), 777-782 (2010).
- Brouhard, G. J. Dynamic instability 30 years later: complexities in microtubule growth and catastrophe. Molecular Biology of the Cell. 26 (7), 1207-1210 (2015).
- Burbank, K. S., Mitchison, T. J. Microtubule dynamic instability. Current Biology : CB. 16 (14), 516-517 (2006).
- Caplow, M., Shanks, J., Ruhlen, R. L. Temperature-jump studies of microtubule dynamic instability. Journal of Biological Chemistry. 263 (21), 10344-10352 (1988).
- Prasad, V., Jordan, M. A., Luduena, R. F. Temperature sensitivity of vinblastine-induced tubulin polymerization in the presence of microtubule-associated proteins. Journal of Protein Chemistry. 11 (5), 509-515 (1992).
- Wasteneys, G. O. Microtubules Show their Sensitive Nature. Plant and Cell Physiology. 44 (7), 653-654 (2003).
- Turi, A., Lu, R. C., Lin, P. -S. Effect of heat on the microtubule disassembly and its relationship to body temperatures. Biochemical and Biophysical Research Communications. 100 (2), 584-590 (1981).
- Safinya, C. R., et al. The effect of multivalent cations and Tau on paclitaxel-stabilized microtubule assembly, disassembly, and structure. Advances in Colloid and Interface Science. 232, 9-16 (2016).
- Sandoval, I. V., Weber, K. Calcium-Induced Inactivation of Microtubule Formation in Brain Extracts. European Journal of Biochemistry. 92 (2), 463-470 (1978).
- Vater, W., Böhm, K. J., Unger, E. Tubulin assembly in the presence of calcium ions and taxol: Microtubule bundling and formation of macrotubule-ring complexes. Cell Motility. 36 (1), 76-83 (1997).
- Yamashita, N., et al. Three-dimensional tracking of plus-tips by lattice light-sheet microscopy permits the quantification of microtubule growth trajectories within the mitotic apparatus. Journal of Biomedical Optics. 20 (10), 1-18 (2015).
- Pamula, M. C., et al. High-resolution imaging reveals how the spindle midzone impacts chromosome movement. Journal of Cell Biology. 218 (8), 2529-2544 (2019).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados