A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
القياس الكمي لآثار مضادات الميكروبات على بنية الأغشية الحيوية في المختبر باستخدام برمجيات COMSTAT
In This Article
Summary
تختلف التغيرات التي تسببها مضادات الميكروبات في بنية الأغشية الحيوية Pseudomonas aeruginosa بين العزلات السريرية المستزرعة من المرضى الذين يعانون من التليف الكيسي والعدوى الرئوية المزمنة. بعد الفحص المجهري متحد البؤر ، يمكن استخدام برنامج COMSTAT لتحديد الاختلافات في بنية الأغشية الحيوية (على سبيل المثال ، مساحة السطح ، السمك ، الكتلة الحيوية) للعزلات الفردية لتقييم فعالية العوامل المضادة للعدوى.
Abstract
الأغشية الحيوية هي مجاميع من الكائنات الحية الدقيقة التي تعتمد على مصفوفة منتجة ذاتيا من مادة بوليمرية خارج الخلية للحماية والسلامة الهيكلية. من المعروف أن العامل الممرض في المستشفيات ، Pseudomonas aeruginosa ، يتبنى وضع نمو الأغشية الحيوية ، مما يسبب عدوى رئوية مزمنة في مرضى التليف الكيسي (CF). والبرنامج الحاسوبي COMSTAT أداة مفيدة لقياس التغيرات التي تسببها مضادات الميكروبات في بنية الأغشية الحيوية P. aeruginosa عن طريق استخراج البيانات من الصور ثلاثية البؤر البؤرية. ومع ذلك ، فإن التشغيل الموحد للبرنامج أقل شيوعا ، وهو أمر مهم للإبلاغ الأمثل عن سلوك الأغشية الحيوية والمقارنة عبر المراكز. وبالتالي ، فإن الهدف من هذا البروتوكول هو توفير إطار بسيط وقابل للتكرار لقياس هياكل الأغشية الحيوية في المختبر في ظل ظروف مختلفة مضادة للميكروبات عبر COMSTAT. تم تصميم هذه التقنية باستخدام عزل CF P. aeruginosa ، الذي ينمو في شكل نسخ بيوفيلم مكرر ، ويتعرض للتوبراميسين والجسم المضاد أحادي النسيلة المضاد ل Psl ، Psl0096. يهدف النهج التدريجي إلى تقليل غموض المستخدم وتقليل فرصة تجاهل خطوات معالجة الصور الحاسمة. وعلى وجه التحديد، يشدد البروتوكول على إزالة الاختلافات الذاتية المرتبطة بالتشغيل اليدوي للقاعدة الإحصائية للقاعدة الإحصائية (COMSTAT)، بما في ذلك تجزئة الصور واختيار وظائف التحليل الكمي المناسبة. على الرغم من أن هذه الطريقة تتطلب من المستخدمين قضاء وقت إضافي في معالجة الصور متحدة البؤر قبل تشغيل COMSTAT ، إلا أنها تساعد على تقليل عدم تجانس الأغشية الحيوية المشوهة في المخرجات الآلية.
Introduction
الأغشية الحيوية هي مجاميع من الكائنات الحية الدقيقة الموجهة في مصفوفة من المواد البوليمرية خارج الخلية المنتجة ذاتيا (EPS). مصفوفة EPS معقدة للغاية ، وتتكون بشكل أساسي من الخلايا البكتيرية والماء والبروتينات والسكريات والدهون والأحماض النووية1 ، وكلها تجعل الأغشية الحيوية مختلفة بشكل واضح عن خلايا العوالق الحية الحرة. Biofilm EPS ملتصقة ببعضها البعض والأسطح المختلفة. تتميز مصفوفة EPS بخصائص تتوسط التبادل من خلية إلى خلية للمستقلبات والمواد الوراثية والمركبات المستخدمة في الإشارات والدفاع بين الخلايا2. توفر هذه الخصائص مجتمعة السلامة الهيكلية للأغشية الحيوية والحماية ضد الضغوطات الخارجية ، مما يساهم في التهرب المناعي ومقاومة مضادات الميكروبات3.
Pseudomonas aeruginosa هو أحد مسببات الأمراض المعترف بها جيدا في المستشفيات ، والمعروف أنه يتبنى استراتيجية نمو الأغشية الحيوية المراوغة استجابة لمضادات الميكروبات. مثال رئيسي على ذلك يحدث في المرضى الذين يعانون من الاضطراب الوراثي المتنحي ، التليف الكيسي (CF). تلعب الأغشية الحيوية دورا محوريا في تطوير P. aeruginosa4 المقاوم لمضادات الميكروبات وتسمح بإنشاء عدوى رئوية مزمنة في مرضى التليف الكيسي ، مما يتسبب في انخفاض متسارع في وظائف الرئة والوفيات المبكرة5. ومن ثم ، يتم إجراء دراسات الأغشية الحيوية في المختبر لاختبار فعالية المضادات الحيوية والعوامل الجديدة المضادة للعدوى ضد عزلات P. aeruginosa التي تم الحصول عليها من المرضى الذين يعانون من CF 6,7. بعد تكوين الأغشية الحيوية الرقيقة ، يتم تطبيق مضادات الميكروبات خارجيا على الهيكل ، ويتم استخدام مجهر المسح بالليزر متحد البؤر (CLSM) لتوليد عمليات إعادة بناء عالية الدقة وثلاثية الأبعاد لشرائح الأغشية الحيوية. من الممارسات الشائعة استخدام برنامج الكمبيوتر ، COMSTAT ، كمكون إضافي ل ImageJ ، لتحديد التغييرات في بنية الأغشية الحيوية8،9،10،11.
وعلى الرغم من أن الكومستات مفيد في القياس الكمي لبنية الأغشية الحيوية، فإن قابلية استنساخ تحليل الصور وتوحيدها أقل شيوعا. على سبيل المثال ، إجراء معالجة الصور ، الذي تم إجراؤه قبل تشغيل COMSTAT ، موضوعي ، ولكنه يحتوي على عنصر ذاتي عند تعيين عتبات الصورة12,13. بطريقة مماثلة ، يسمح برنامج COMSTAT للمشغل بالاختيار من بين الشروط والمعلمات الأساسية إلى المتقدمة لتجزئة الصورة بالإضافة إلى عشر وظائف تحليل كمي (على سبيل المثال ، توزيع السمك ، مساحة السطح ، الكتلة الحيوية ، معامل الخشونة بدون أبعاد). قد يؤدي تعدد خيارات المستخدم ، بالإضافة إلى اختلاف مستويات خبرة المشغلين ، إلى الإبلاغ المضلل عن سلوك الأغشية الحيوية.
وبالتالي ، فإن الهدف من هذا البروتوكول هو تقديم طريقة بسيطة نسبيا للمقارنة الكمية لهياكل الأغشية الحيوية في المختبر باستخدام COMSTAT. هنا ، يتم التقاط صور ثلاثية الأبعاد لمقاطع الأغشية الحيوية من عزل CF P. aeruginosa عبر CLSM باستخدام نموذج غطاء زجاجي حجري14 - وهي تقنية راسخة تستخدم لإجراء تجارب الأغشية الحيوية القابلة للتكرار في المختبر . باستخدام COMSTAT كمكون إضافي ل ImageJ ، تسمح هذه الطريقة للباحثين بتحديد التغيرات في بنية الأغشية الحيوية كميا في وجود مضادات الميكروبات في ظل ظروف مختلفة. وبشكل عام، تهدف هذه الطريقة إلى القضاء على الاختلافات الذاتية المرتبطة بالتشغيل اليدوي للقاعدة العامة للإحصاء (COMSTAT)، وبالتالي تسهيل توحيد البروتوكولات عبر المراكز.
Protocol
1. جمع العزلة البكتيرية
- الحصول على عزلات P. aeruginosa من مجموعة من مرضى الأطفال المصابين بالتليف الكيسي الذين يخضعون لعلاج الاستئصال باستخدام توبراميسين المستنشق في SickKids (تورنتو). تجميد العزلات عند -80 درجة مئوية في سترات الجلسرين والمزارع الفرعية ثلاث مرات على الأقل قبل الاستخدام.
2. تشكيل الأغشية الحيوية في المختبر
ملاحظة: استخدم طريقة غطاء زجاجي1 لتشكيل الأغشية الحيوية في المختبر مع التعديلات. يظهر سير العمل العام لهذا النموذج في الشكل 1.
- تنمو P. aeruginosa معزولة طوال الليل عند 37 درجة مئوية على أجار الدم المحضر بأجار الصويا التربتي و 5٪ دم الأغنام (انظر جدول المواد).
- تلقيح 1-2 مستعمرات بكتيرية من أجار الدم إلى 4 مل من مرق الليزوجيني (LB). تنمو بين عشية وضحاها عند 37 درجة مئوية على شاكر مضبوط على 225 دورة في الدقيقة.
- قم بإعداد تخفيف 1: 100 من اللقاح الليلي عن طريق إضافة 40 ميكرولتر من المستنبت في 4 مل من LB الطازج. تنمو لمدة 3-4 ساعات عند 37 درجة مئوية على شاكر مضبوط على 225 دورة في الدقيقة ، لتحقيق كثافة بصرية تبلغ حوالي 0.1 عند 600 نانومتر (OD600) (مرحلة السجل المبكر).
- انقل 220 ميكرولتر من اللقاح إلى كل بئر من شريحة زجاجية ذات 8 غرف. احتضان دون عائق عند 37 درجة مئوية لمدة 24 ساعة.
- قم بإزالة الوسط ببطء من كل بئر لمنع الأغشية الحيوية من الانفصال عند القاعدة.
ملاحظة: قم بإمالة الشريحة للأمام بزاوية 45 درجة وقم بشفط الوسط من الزوايا السفلية لكل بئر حجرة دون لمس القاعدة بطرف ماصة. - تحضير وإضافة 100 ميكرولتر ببطء من 56 ميكروغرام / مل من الجسم المضاد أحادي النسيلة المسمى بالفلورسنت (mAb) (انظر جدول المواد) إلى جانب الآبار المخصصة للغرفة. احتضان في درجة حرارة الغرفة (RT) لمدة 1 ساعة للسماح لارتباط الجسم المضاد بخاتمة المستضد البكتيري.
ملاحظة: قبل الاستخدام، قم بتخفيف الفلورسنت المسمى (الأحمر) mAb، Psl0096، في رطل للحصول على تركيز نهائي قدره 56 ميكروغرام/مل. Psl0096 هو مضاد ل Psl mAb (مشتق تقارب محسن من Cam003) ، والذي يرتبط بخاتمة Psl من الفئة الأولى - وهو مكون رئيسي لمصفوفة EPS للأغشية الحيوية P. aeruginosa المشاركة في الارتباط الأولي للخلية والسلامة الهيكلية15. - قم بإعداد وإضافة 100 ميكرولتر ببطء من محلول مضاد حيوي 1000 ميكروغرام / مل (انظر جدول المواد) إلى جانب الآبار المخصصة للغرفة. احتضان دون عائق عند 37 درجة مئوية لمدة 24 ساعة.
ملاحظة: قبل الاستخدام، يتم تخفيف مخزون 50 ملغ/مل من مضاد توبراميسين الحيوي في رطل للحصول على تركيز نهائي قدره 1000 ميكروغرام/مل.
3. تلطيخ الفلورسنت بيوفيلم
- تحضير محلول 0.01 mM من صبغة فلورية ملطخة بالخلايا الحية. قم بإزالة الوسط ببطء من الآبار الحجرية وأضف 200 ميكرولتر من خليط الصبغة إلى كل بئر. احتضان في RT في الظلام لمدة 45 دقيقة.
ملاحظة: قبل الاستخدام ، قم بإعداد صبغة الفلورسنت الملطخة بالخلايا الحية (الخضراء) بإضافة 4 ميكرولتر من مخزون 5 مللي مول إلى 2 مل من LB. - قم بإزالة الوسط ببطء من كل بئر ، واغسل 2x ب 200 ميكرولتر من LB الطازج.
- أضف 200 ميكرولتر من LB الطازج إلى كل بئر وانتقل إلى الفحص عبر الفحص المجهري متحد البؤر.
4. الحصول على الصور عن طريق المجهر متحد البؤر
ملاحظة: يرد في الشكل 2 إجراء معالجة الصور وتحليل COMSTAT. الحصول على صور للآبار في نفس يوم تلطيخ الأغشية الحيوية. إذا تجاوز التأخير في التصور 1 ساعة ، فقم بتبريد غطاء الغرفة في الظلام حتى تتم المعالجة الإضافية.
- الحصول على صور للآبار باستخدام نظام مجهر متحد البؤر (انظر جدول المواد) مع أطوال موجية مناسبة للإثارة بالليزر ومجموعات مرشحات للاقتناء.
ملاحظة: هنا ، قم بإثارة صبغة mAb الفلورية (الحمراء) والخلية الحية (الخضراء) باستخدام الأطوال الموجية للإثارة 561 و 491 نانومتر ، على التوالي. - التقط صورا ذات طبقات من z-stack (من الطبقة التحتية إلى أعلى كل جزء من الأغشية الحيوية) بزيادات قدرها 0.3 ميكرومتر باستخدام عدسة غمر بالماء بمعدل 20-25 ضعفا. خذ ما لا يقل عن 6 مكدسات صور لكل بئر.
ملاحظة: هنا ، تصور الصور باستخدام كاميرا عالية الدقة مع عدسة غمر بالماء 25x ومعالجتها باستخدام برنامج تحليل الصور (انظر جدول المواد). حافظ على إعداد البرامج ومعلمات التصوير الرقمي (أي السطوع والحساسية) ثابتة لجميع عمليات الاستحواذ في تجربة واحدة. - احفظ الصور بتنسيق OME-TIFFs لتحليل COMSTAT.
ملاحظة: تأكد من حفظ OME-TIFFs بشكل منفصل لكل قناة (أي الأحمر والأخضر). تختلف هذه الخطوة وفقا لبرنامج تحليل الصور المستخدم. - كرر الخطوات 2.1-4.3 لالتقاط صور من إجمالي 3 مكررات بيولوجية (أي 3 تجارب مستقلة) لكل عزل بكتيري.
5. تحليل الكومستات
ملاحظة: تحليل الصور كميا باستخدام برنامج الكمبيوتر المتاح مجانا ، COMSTAT16,17 ، المعاد كتابته كمكون إضافي (Comstat2) إلى ImageJ. اقرأ التعليمات العامة لتحليل مكدسات صور الأغشية الحيوية داخل الحزمة التي تم تنزيلها. توفر هذه المساهمة بروتوكولا موجزا ، مع خطوات معالجة مختارة ل ImageJ وميزات COMSTAT الموصى بها لتحديد آثار مضادات الميكروبات على تكوين الأغشية الحيوية.
- قم بتنزيل حزمة Comstat2 من http://www.comstat.dk/. داخل المجلد المثبت ، حدد موقع ImageJ وقم بتشغيله.
- قم بإنشاء مجلد مصدر على سطح المكتب وأضف OME-TIFF واحدا إلى المجلد.
- افتح OME-TIFF من المجلد المصدر ، واحذف أي طبقات فارغة لا تحتوي على كتلة حيوية. ستكون هذه الطبقات إما الطبقات القليلة الأولى أو الأخيرة من مكدس z.
ملاحظة: أحيانا ما يبالغ المستخدمون في تقدير حدود مكدس z المعرفة بالمجهر. يؤدي حذف هذه الطبقات الفارغة إلى إنشاء حدود مكدس z أكثر دقة لتحليل COMSTAT. - استيراد OME-TIFF في ImageJ عن طريق تحديد ملف | استيراد | تسلسل الصور. حدد موقع المجلد المصدر، وقم بتمييزه دون فتحه، وانقر فوق تحديد. ستظهر نافذة "خيارات التسلسل". حدد موافق.
ملاحظة: لاستيراد صور إضافية في ImageJ، قم أولا بإزالة OME-TIFF السابق من المجلد المصدر، ثم أضف OME-TIFF الجديد إلى المجلد، وكرر الخطوتين 5.3 و5.4. - اقلب اتجاه الغشاء الحيوي الرقيق بتحديد صورة | تحويل | اقلب Z لوضع الطبقة التحتية كأول مكدس (أعلى).
ملاحظة: تقرأ خوارزميات COMSTAT الأغشية الحيوية في الاتجاه z من الأعلى (المكدس 1) إلى الأسفل. اعتمادا على نظام المجهر متحد البؤر المستخدم ، يمكن عكس إخراج OME-TIFF. وبالتالي ، من المهم عكس ترتيب الشرائح عن طريق وضع الطبقة التحتية كمكدس صور 1 لمنع بيانات الإخراج من أن تصبح معيبة. - تحديد خصائص الصورة عن طريق تحديد صورة | الخصائص. ستظهر نافذة "المصدر".
- حدد "وحدة الطول" ك "ميكرون".
- حدد رياضيا "عرض البكسل" و "ارتفاع البكسل" باستخدام المعادلة التالية:
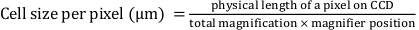 (1)
(1)
هنا ، يتم تعريف "عرض البكسل" و "ارتفاع البكسل" على أنه 0.427 ، حيث يكون الطول الفعلي للبكسل على الجهاز المقترن بالشحن (CCD) 16 ميكرومتر ؛ التكبير الكلي هو 25x ؛ وموضع المكبر هو 1.5x.
ملاحظة: قد تختلف المعادلة المستخدمة لحساب حجم الخلية لكل بكسل وفقا للشركة المصنعة لكاميرا المجهر. بدلا من ذلك ، يمكن تعريف "عرض البكسل" و "ارتفاع البكسل" من خلال المعايرة المكانية (راجع https://imagej.net/Spatial_Calibration). - حدد "عمق Voxel" على أنه 0.3 (أي مسافة تزايدية بين كل طبقة z-stack). حدد موافق في نافذة "المصدر".
- اضبط حد الصورة عن طريق تحديد صورة | ضبط | العتبة. ستظهر نافذة "العتبة".
ملاحظة: ستظهر الكائنات الموجودة في الصورة باللون الأحمر مع خلفية رمادية. بدلا من ذلك ، يمكن تعديل العتبة بالأبيض والأسود ، باستخدام القائمة المنسدلة في نافذة "العتبة" لتغيير "الأحمر" إلى "B&W".- في نافذة الصورة، اضبط شريط التمرير إلى أقصى اليمين (أي الطبقة العليا من الغشاء الحيوي). لإزالة ضوضاء الخلفية ، استخدم نافذة "العتبة" ، التي تعرض مدرجا تكراريا للصورة ، لتعيين قيم الحد الأقصى والحد الأدنى يدويا. أولا ، قم بتعيين الحد الأقصى لقيمة العتبة عن طريق ضبط شريط التمرير السفلي في أقصى اليمين قدر الإمكان. ثانيا ، استخدم شريط التمرير العلوي لضبط الحد الأدنى لقيمة العتبة ، والذي يقسم الصورة إلى مرحلتين منفصلتين: الكتلة الحيوية الحمراء والخلفية ذات التدرج الرمادي (الشكل 3).
ملاحظة: يقوم إجراء تكوين الأغشية الحيوية الرقيقة في المختبر وإجراء الفحص المجهري الفلوري الموصوف هنا بإنشاء OME-TIFFs في الحالة المثالية ، مما يسمح بتقسيم الصور إلى مرحلتين متميزتين عن طريق طريقة عتبة الرسم البياني البسيطة. ومع ذلك ، في بعض الحالات ، لا يكون التمييز التكراري بين المراحل المختلفة واضحا. يمكن أن يكون هذا بسبب وجود ضوضاء خلفية واسعة النطاق ، أو شدة خلفية متفاوتة ، أو تباين منخفض الكثافة بين الكتلة الحيوية والخلفية. في مثل هذه الحالات ، يجب على المستخدمين اعتماد إجراء تجزئة محسن 18,19.- بدلا من ذلك ، اضبط الحدود خوارزميا للصور الفردية باستخدام القائمة المنسدلة اليسرى التي تم تعيينها على أنها "افتراضية" في نافذة "العتبة". توفر هذه الميزة 17 خيارا مختلفا لعتبة خوارزمية للاختيار من بينها (راجع https://imagej.net/Auto_Threshold). حدد الخيار الأكثر قابلية للتطبيق ، ثم "تلقائي" لتعيين العتبة.
- عند ضبط قيم الحد، استخدم المنزلق في نافذة الصورة للتمرير خلال كل طبقة لضمان إزالة ضوضاء الخلفية بشكل كاف طوال الوقت.
- حدد تعيين في نافذة "العتبة" لإصلاح قيمة الحد الأدنى أولا. ستظهر نافذة "تعيين مستويات العتبة". حدد موافق. حدد تعيين مرة أخرى وكرر هذه الخطوة لإصلاح الحد الأقصى لقيمة العتبة.
ملاحظة: في كل مرة يتم فيها تحديد "ضبط" ، قد يتم ضبط شريط التمرير السفلي تلقائيا. في مثل هذه الحالات ، أعد ضبط شريط التمرير يدويا (أو حدد "تلقائي" في حالة استخدام أحد الحدود الخوارزمية) وكرر الخطوات 5.6.2 - 5.6.3. الفكرة الرئيسية هي أنه كلما تم إعادة ضبط أشرطة التمرير تلقائيا ، يجب تحديد "تعيين" مرتين إضافيتين بعد ذلك لضمان إصلاح كل من العتبات العلوية والسفلية. - حدد تطبيق وستظهر نافذة "تحويل المكدس إلى ثنائي". حدد موافق ، ثم اخرج من نافذة "العتبة".
- احفظ OME-TIFF المعدل حديثا عن طريق تحديد المكونات الإضافية | التنسيقات الحيوية | مصدر التنسيقات الحيوية |. أدخل اسم ملف جديد واحفظه ك OME-TIFF في المجلد المصدر. ستظهر نافذة "مصدر التنسيقات الحيوية - ملفات متعددة". حدد موافق. ستظهر نافذة "خيارات مصدر التنسيقات الحيوية". حدد موافق.
ملاحظة: تأكد من حفظ OME-TIFF(s) المعدلة حديثا بالأبيض والأسود فقط في المجلد المصدر قبل المتابعة إلى تحليل COMSTAT. قم بإزالة كافة ملفات OME-TIFF الأصلية من المجلد.
- في نافذة الصورة، اضبط شريط التمرير إلى أقصى اليمين (أي الطبقة العليا من الغشاء الحيوي). لإزالة ضوضاء الخلفية ، استخدم نافذة "العتبة" ، التي تعرض مدرجا تكراريا للصورة ، لتعيين قيم الحد الأقصى والحد الأدنى يدويا. أولا ، قم بتعيين الحد الأقصى لقيمة العتبة عن طريق ضبط شريط التمرير السفلي في أقصى اليمين قدر الإمكان. ثانيا ، استخدم شريط التمرير العلوي لضبط الحد الأدنى لقيمة العتبة ، والذي يقسم الصورة إلى مرحلتين منفصلتين: الكتلة الحيوية الحمراء والخلفية ذات التدرج الرمادي (الشكل 3).
- قم بتشغيل COMSTAT عن طريق تحديد المكونات الإضافية | كومستات 2 |. ستظهر نافذة "حول". حدد موافق. ستظهر ثلاث نوافذ.
- في نافذة "الدلائل المرصودة" (أعلى يسار الصفحة)، اختر إضافة. حدد موقع المجلد المصدر، وقم بتمييزه دون فتحه، وحدد اختيار. ستظهر نافذة "الصور في الدلائل" (أعلى اليسار) التي تسرد OME-TIFFs ليتم تحليلها عبر COMSTAT.
- في نافذة "Comstat 2.1" (أسفل اليمين) ، قم بإلغاء تحديد "العتبة التلقائية (طريقة Otsu)" للتأكد من أن البرنامج يستخدم قيم العتبة التي تم إعدادها مسبقا ل OME-TIFFs الفردية. أيضا ، قم بإلغاء تحديد مرشح وحدة التخزين المتصلg (CVF) لضمان تضمين الأجزاء الرقيقة جدا من الأغشية الحيوية وكذلك الخلايا العائمة الحرة أو الكتلة الحيوية الموجودة داخل فراغات بنية الأغشية الحيوية في التحليل.
ملاحظة: هنا ، قم بإلغاء تحديد CVF لأن تحليل COMSTAT يتم إجراؤه على الأغشية الحيوية المبكرة جدا (النمو الأولي لمدة 24 ساعة) والخلايا / المستعمرات العوالق المتبقية بعد العلاج بمضادات الميكروبات. بالنسبة للأغشية الحيوية الناضجة، حدد CVF للتأكد من أن الكتلة الحيوية المتصلة بهيكل الأغشية الحيوية فقط هي التي يتم قياسها كميا. - في نافذة "Comstat 2.1" (أسفل اليمين) ، حدد الميزات المطلوبة للتحليل الكمي. هنا ، حدد الكتلة الحيوية وتوزيع السماكة ومساحة السطح. حدد Go لتشغيل البرنامج. في نافذة "السجل" (أسفل اليسار) ، تظهر بيانات الإخراج وهي تعالج حتى تظهر "تم باستخدام الوظائف / الصور المحددة!". سجل قياسات COMSTAT. يتم أيضا حفظ هذه القياسات تلقائيا كملفات TXT في المجلد المصدر.
النتائج
يتم استخدام عزل P. aeruginosa المستزرع من مريض مصاب بالتليف الكيسي لإثبات نقاط القوة في هذا النهج في القياس الكمي الدقيق للتغيرات التي تسببها مضادات الميكروبات في بنية الأغشية الحيوية في المختبر . يتم تمثيل سير العمل العام لهذا النموذج في الشكل 1. يظهر إجراء معالجة الص?...
Discussion
لا توجد طريقة محددة للمقارنة الكمية للصور ثلاثية الأبعاد لهياكل الأغشية الحيوية في المختبر ، وغالبا ما يصعب توحيد الإجراءات الموصوفة في هذا السياق بسبب التباين بين المشغلين20. وهكذا، يوفر هذا البروتوكول إطارا بسيطا وقابلا للتكرار لتطبيقات الكومستات التي تسعى إلى قياس ا...
Disclosures
اي
Acknowledgements
يود المؤلفون أن يشكروا مؤسسة التليف الكيسي لتوفير التمويل لهذا البحث.
Materials
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
References
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved