È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Quantificazione degli effetti degli antimicrobici sull'architettura del biofilm in vitro utilizzando il software COMSTAT
In questo articolo
Riepilogo
Le alterazioni indotte da antimicrobici dell'architettura del biofilm di Pseudomonas aeruginosa differiscono tra gli isolati clinici coltivati da pazienti con fibrosi cistica e infezione polmonare cronica. Dopo la microscopia confocale, il software COMSTAT può essere utilizzato per quantificare le variazioni nell'architettura del biofilm (ad esempio, area superficiale, spessore, biomassa) per i singoli isolati per valutare l'efficacia degli agenti anti-infettivi.
Abstract
I biofilm sono aggregati di microrganismi che si basano su una matrice autoprodotta di sostanza polimerica extracellulare per la protezione e l'integrità strutturale. Il patogeno nosocomiale, Pseudomonas aeruginosa, è noto per adottare una modalità di crescita del biofilm, causando infezione polmonare cronica nei pazienti con fibrosi cistica (FC). Il programma informatico, COMSTAT, è uno strumento utile per quantificare i cambiamenti indotti da antimicrobici nell'architettura del biofilm di P. aeruginosa estraendo dati da immagini confocali tridimensionali. Tuttavia, il funzionamento standardizzato del software è meno comunemente affrontato, il che è importante per la segnalazione ottimale del comportamento del biofilm e il confronto tra i centri. Pertanto, l'obiettivo di questo protocollo è quello di fornire un quadro semplice e riproducibile per quantificare le strutture del biofilm in vitro in condizioni antimicrobiche variabili tramite COMSTAT. La tecnica è modellata utilizzando un isolato di CF P. aeruginosa , coltivato sotto forma di repliche di biofilm ed esposto alla tobramicina e all'anticorpo monoclonale anti-Psl, Psl0096. L'approccio graduale mira a ridurre l'ambiguità dell'utente e a minimizzare la possibilità di trascurare le fasi cruciali dell'elaborazione delle immagini. In particolare, il protocollo enfatizza l'eliminazione delle variazioni soggettive associate al funzionamento manuale di COMSTAT, tra cui la segmentazione delle immagini e la selezione di appropriate funzioni di analisi quantitativa. Sebbene questo metodo richieda agli utenti di dedicare più tempo all'elaborazione di immagini confocali prima di eseguire COMSTAT, aiuta a ridurre al minimo l'eterogenicità del biofilm travisato negli output automatizzati.
Introduzione
I biofilm sono aggregati di microrganismi orientati in una matrice di sostanze polimeriche extracellulari (EPS) autoprodotte. La matrice EPS è molto complessa, costituita principalmente da cellule batteriche, acqua, proteine, polisaccaridi, lipidi e acidi nucleici1, che rendono i biofilm nettamente diversi dalle cellule planctoniche a vita libera. I Biofilm EPS sono aderenti l'uno all'altro e alle varie superfici. La matrice EPS ha proprietà che mediano lo scambio cellula-cellula di metaboliti, materiale genetico e composti utilizzati per la segnalazione e la difesa intercellulare2. Queste proprietà forniscono collettivamente integrità strutturale dei biofilm e protezione contro i fattori di stress esterni, contribuendo all'evasione immunitaria e alla resistenza antimicrobica3.
Pseudomonas aeruginosa è un patogeno nosocomiale ben noto, noto per adottare una strategia evasiva di crescita del biofilm in risposta agli antimicrobici. Un primo esempio di ciò si verifica nei pazienti con la malattia genetica recessiva, la fibrosi cistica (FC). I biofilm svolgono un ruolo fondamentale nello sviluppo di P. aeruginosa 4 resistente agli antimicrobici e consentono l'instaurarsi di un'infezione polmonare cronica nei pazienti con FC, causando un declino accelerato della funzione polmonare e una mortalità prematura5. Pertanto, vengono eseguiti studi in vitro sul biofilm per testare l'efficacia di antibiotici e nuovi agenti antinfettivi contro gli isolati di P. aeruginosa ottenuti da pazienti con FC 6,7. Dopo la formazione del biofilm, gli antimicrobici vengono applicati esternamente alla struttura e la microscopia a scansione laser confocale (CLSM) viene utilizzata per generare ricostruzioni tridimensionali ad alta risoluzione di segmenti di biofilm. È pratica comune utilizzare il software per computer, COMSTAT, come plug-in di ImageJ, per quantificare i cambiamenti nell'architettura del biofilm 8,9,10,11.
Sebbene COMSTAT sia utile per quantificare la struttura del biofilm, la riproducibilità e la standardizzazione dell'analisi delle immagini sono meno comunemente affrontate. Ad esempio, la procedura di elaborazione delle immagini, eseguita prima dell'esecuzione di COMSTAT, è oggettiva, ma contiene un elemento di soggettività quando si impostano le soglie delle immagini12,13. Allo stesso modo, il programma COMSTAT consente all'operatore di scegliere tra condizioni e parametri di base e avanzati per la segmentazione delle immagini, nonché dieci funzioni di analisi quantitativa (ad esempio, distribuzione dello spessore, area superficiale, biomassa, coefficiente di rugosità adimensionale). La moltitudine di opzioni per l'utente, combinate con i diversi livelli di esperienza dell'operatore, può portare a una segnalazione errata del comportamento del biofilm.
Pertanto, l'obiettivo di questo protocollo è quello di presentare un metodo relativamente semplice per il confronto quantitativo di strutture di biofilm in vitro utilizzando COMSTAT. In questo articolo, le immagini tridimensionali dei segmenti di biofilm di un isolato di CF P. aeruginosa vengono acquisite tramite CLSM utilizzando il modello14 di vetro di copertura a camera, una tecnica consolidata utilizzata per eseguire esperimenti di biofilm in vitro riproducibili. Utilizzando COMSTAT come plug-in di ImageJ, questo metodo consente ai ricercatori di identificare quantitativamente i cambiamenti nell'architettura del biofilm in presenza di antimicrobici in condizioni variabili. Nel complesso, questo metodo mira a eliminare le variazioni soggettive associate al funzionamento manuale di COMSTAT, facilitando così la standardizzazione dei protocolli tra i centri.
Protocollo
1. Raccolta di isolati batterici
- Ottenere isolati di P. aeruginosa da una coorte di pazienti pediatrici con FC sottoposti a trattamento di eradicazione con tobramicina per via inalatoria presso SickKids (Toronto). Congelare gli isolati a -80 °C in glicerolo citrato e sottocoltura almeno tre volte prima dell'uso.
2. Formazione di biofilm in vitro
NOTA: Utilizzare un metodo di coverglass a camera1 per la formazione di biofilm in vitro con modifiche. Il flusso di lavoro complessivo di questo modello è illustrato nella Figura 1.
- Coltivare l'isolato di P. aeruginosa per una notte a 37 °C su agar sanguigno preparato con agar di soia triptica e sangue di pecora al 5% (vedi Tabella dei materiali).
- Inoculare 1-2 colonie batteriche dall'agar sanguigno in 4 ml di brodo di lisogenia (LB). Coltivare per una notte a 37 °C su uno shaker impostato a 225 giri/min.
- Preparare una diluizione 1:100 dell'inoculo notturno aggiungendo 40 μL di coltura in 4 mL di LB fresco. Coltivare per 3-4 ore a 37 °C su uno shaker impostato a 225 giri/min, per ottenere una densità ottica di circa 0,1 a 600 nm (OD600) (fase logaritmica iniziale).
- Trasferire 220 μl di inoculo in ciascun pozzetto di un vetrino di copertura a 8 camere. Incubare indisturbato a 37 °C per 24 ore.
- Rimuovere lentamente il terreno da ogni pozzetto per evitare che i biofilm si stacchino alla base.
NOTA: Inclinare il carrello in avanti con un angolo di 45° e aspirare il fluido dagli angoli inferiori di ciascun pozzetto della camera senza toccare la base con la punta della pipetta. - Preparare e aggiungere lentamente 100 μL di anticorpo monoclonale (mAb) marcato con fluorescenza da 56 μg/mL (vedere la Tabella dei materiali) a lato dei pozzetti a camera designati. Incubare a temperatura ambiente (RT) per 1 ora per consentire l'adesione degli anticorpi all'epitopo dell'antigene batterico.
NOTA: Prima dell'uso, diluire l'anticorpo monoclonale fluorescente marcato (rosso), Psl0096, in LB per ottenere una concentrazione finale di 56 μg/mL. Psl0096 è un anticorpo monoclonale anti-Psl (derivato di affinità ottimizzato di Cam003), che si lega all'epitopo Psl di classe I, un componente chiave della matrice EPS dei biofilm di P. aeruginosa coinvolti nell'adesione iniziale alle cellule e nell'integrità strutturale15. - Preparare e aggiungere lentamente 100 μL di una soluzione antibiotica da 1000 μg/mL (vedere la Tabella dei materiali) sul lato dei pozzetti a camera designati. Incubare indisturbato a 37 °C per 24 ore.
NOTA: Prima dell'uso, diluire una dose di 50 mg/mL di antibiotico tobramicina in LB per ottenere una concentrazione finale di 1000 μg/mL.
3. Colorazione fluorescente del biofilm
- Preparare una soluzione 0,01 mM di un colorante fluorescente colorante con cellule vive. Rimuovere lentamente il terreno dai pozzetti della camera e aggiungere 200 μl della miscela di colorante a ciascun pozzetto. Incubare a RT al buio per 45 min.
NOTA: Prima dell'uso, preparare il colorante fluorescente per la colorazione delle cellule vive (verde) aggiungendo 4 μL di un stock da 5 mM a 2 mL di LB. - Rimuovere lentamente il terreno da ciascun pozzetto e lavare 2 volte con 200 μl di LB fresco.
- Aggiungere 200 μL di LB fresco a ciascun pozzetto e procedere all'esame tramite microscopia confocale.
4. Acquisizione di immagini mediante microscopia confocale
NOTA: La procedura di elaborazione delle immagini e di analisi COMSTAT è presentata nella Figura 2. Acquisire immagini dei pozzetti lo stesso giorno della colorazione del biofilm. Se il ritardo nella visualizzazione supera 1 ora, refrigerare il vetro coprioggetto a camera al buio fino a nuova lavorazione.
- Acquisizione di immagini di pozzetti utilizzando un sistema di microscopio confocale (vedi Tabella dei materiali) con lunghezze d'onda di eccitazione laser appropriate e set di filtri per l'acquisizione.
NOTA: Qui, eccitare l'anticorpo monoclonale marcato con fluorescenza (rosso) e la colorazione di cellule vive (verde) utilizzando lunghezze d'onda di eccitazione rispettivamente di 561 e 491 nm. - Acquisisci immagini z-stack stratificate (dal substrato alla parte superiore di ciascun segmento di biofilm) con incrementi di 0,3 μm con una lente a immersione in acqua 20-25x. Prendi almeno 6 pile di immagini per pozzetto.
NOTA: Qui, visualizza le immagini utilizzando una fotocamera ad alta risoluzione con un obiettivo a immersione in acqua 25x ed elaborate utilizzando un software di analisi delle immagini (vedi Tabella dei materiali). Mantieni costanti la configurazione del software e i parametri di imaging digitale (ad esempio, luminosità e sensibilità) per tutte le acquisizioni in un singolo esperimento. - Salva le immagini come OME-TIFF per l'analisi COMSTAT.
NOTA: Assicurarsi che gli OME-TIFF siano salvati separatamente per ciascun canale (ad esempio, rosso e verde). Questo passaggio varia a seconda del software di analisi delle immagini utilizzato. - Ripetere i passaggi da 2.1 a 4.3 per acquisire immagini da un totale di 3 repliche biologiche (cioè 3 esperimenti indipendenti) per isolato batterico.
5. Analisi COMSTAT
NOTA: Analizza le immagini quantitativamente utilizzando il programma informatico disponibile gratuitamente, COMSTAT16,17, riscritto come plugin (Comstat2) per ImageJ. Leggi le istruzioni generali per l'analisi delle pile di immagini di biofilm all'interno del pacchetto scaricato. Questo contributo fornisce un protocollo riassuntivo, con fasi di elaborazione ImageJ selezionate e funzionalità COMSTAT raccomandate per quantificare gli effetti degli antimicrobici sulla formazione del biofilm.
- Scarica il pacchetto Comstat2 da http://www.comstat.dk/. All'interno della cartella installata, individuare ImageJ ed eseguirlo.
- Creare una cartella di origine sul desktop e aggiungere un singolo OME-TIFF alla cartella.
- Aprire OME-TIFF dalla cartella di origine ed eliminare tutti i livelli vuoti che non contengono biomassa. Questi livelli saranno il primo o l'ultimo strato dello z-stack.
NOTA: I limiti dello z-stack definiti dal microscopio sono talvolta sovrastimati dagli utenti. L'eliminazione di questi livelli vuoti stabilisce un limite z-stack più raffinato per l'analisi COMSTAT. - Importare OME-TIFF in ImageJ selezionando File | Importazione | Sequenza di immagini. Individua la cartella di origine, evidenziala senza aprirla e fai clic su Seleziona. Apparirà una finestra "Opzioni sequenza". Selezionare OK.
NOTA: Per importare altre immagini in ImageJ, rimuovere prima il precedente OME-TIFF dalla cartella di origine, quindi aggiungere il nuovo OME-TIFF alla cartella e ripetere i passaggi 5.3 e 5.4. - Capovolgi l'orientamento del biofilm selezionando Immagine | Trasformazione | Capovolgi Z per posizionare il substrato come prima pila (più in alto).
NOTA: Gli algoritmi COMSTAT leggono i biofilm nella direzione z dall'alto (stack 1) verso il basso. A seconda del sistema di microscopio confocale utilizzato, l'uscita OME-TIFF può essere invertita. Pertanto, è importante invertire l'ordine delle sezioni posizionando il substrato come stack di immagini 1 per evitare che i dati di output diventino imperfetti. - Definire le proprietà dell'immagine selezionando Immagine | Proprietà. Apparirà una finestra "Sorgente".
- Specificare 'Unità di lunghezza' come 'micron'.
- Determina matematicamente "Larghezza in pixel" e "Altezza in pixel" utilizzando la seguente equazione:
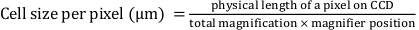 (1)
(1)
In questo caso, la "larghezza dei pixel" e l'"altezza dei pixel" sono definite come 0,427, dove la lunghezza fisica di un pixel sul dispositivo ad accoppiamento di carica (CCD) è di 16 μm; l'ingrandimento totale è 25x; e la posizione della lente d'ingrandimento è 1,5x.
NOTA: L'equazione utilizzata per calcolare la dimensione della cella per pixel può variare a seconda del produttore della fotocamera del microscopio. In alternativa, 'Larghezza pixel' e 'Altezza pixel' possono essere definite mediante calibrazione spaziale (fare riferimento a https://imagej.net/Spatial_Calibration). - Definisci "Profondità Voxel" come 0,3 (cioè spazio incrementale tra ogni livello z-stack). Seleziona OK nella finestra "Sorgente".
- Regolare la soglia dell'immagine selezionando Immagine | Regola | Soglia. Apparirà una finestra "Soglia".
NOTA: Gli oggetti nell'immagine appariranno rossi con uno sfondo in scala di grigi. In alternativa, la soglia può essere regolata in bianco e nero, utilizzando il menu a discesa nella finestra "Soglia" per cambiare "Rosso" in "B&N".- Nella finestra dell'immagine, regolare il cursore all'estrema destra (cioè il livello più in alto del biofilm). Per rimuovere il rumore di fondo, utilizzare la finestra "Soglia", che visualizza un istogramma dell'immagine, per impostare manualmente i valori di soglia massima e minima. Innanzitutto, imposta il valore di soglia massimo regolando il cursore inferiore il più a destra possibile. In secondo luogo, utilizzare il cursore superiore per regolare il valore di soglia minimo, che segmenta l'immagine in due fasi separate: biomassa rossa e sfondo in scala di grigi (Figura 3).
NOTA: La procedura di formazione del biofilm in vitro e di microscopia fluorescente qui descritta genera OME-TIFF nel caso ideale, consentendo di segmentare le immagini in due fasi distinte per mezzo di un semplice metodo di soglia dell'istogramma. Tuttavia, in alcuni casi, la distinzione dell'istogramma tra le diverse fasi non è altrettanto chiara. Ciò può essere dovuto alla presenza di un ampio rumore di fondo, a diverse intensità di fondo o a un contrasto di bassa intensità tra biomassa e fondo. In tali casi, gli utenti devono adottare una procedura di segmentazione avanzata18,19.- In alternativa, regola le soglie in modo algoritmico per le singole immagini utilizzando il menu a discesa a sinistra impostato come "Predefinito" nella finestra "Soglia". Questa funzione offre 17 diverse opzioni di soglia algoritmica tra cui scegliere (fare riferimento alla https://imagej.net/Auto_Threshold). Seleziona l'opzione più applicabile, quindi "Auto" per impostare la soglia.
- Quando i valori di soglia vengono regolati, utilizzare il cursore nella finestra dell'immagine per scorrere ogni livello in modo da assicurarsi che il rumore di fondo venga rimosso in modo sufficiente.
- Selezionare Imposta nella finestra "Soglia" per fissare prima il valore di soglia inferiore. Apparirà una finestra "Imposta livelli di soglia". Selezionare OK. Selezionare nuovamente Imposta e ripetere questo passaggio per fissare il valore di soglia massimo.
NOTA: Ogni volta che si seleziona 'Imposta', il cursore inferiore potrebbe regolarsi automaticamente. In questi casi, regolare nuovamente manualmente il cursore (o selezionare "Auto" se si utilizza una delle soglie algoritmiche) e ripetere i passaggi 5.6.2-5.6.3. L'idea principale è che ogni volta che i cursori vengono regolati automaticamente, "Imposta" dovrebbe essere selezionato altre due volte in seguito per garantire che sia la soglia superiore che quella inferiore siano fisse. - Seleziona Applica e apparirà la finestra "Converti stack in binario". Seleziona OK, quindi esci dalla finestra "Soglia".
- Salva il nuovo OME-TIFF modificato selezionando Plugin | Formati biografici | Esportatore di bioformati|. Immettere un nuovo nome per il file e salvarlo come OME-TIFF nella cartella di origine. Apparirà una finestra "Esportatore di formati biologici - File multipli". Selezionare OK. Apparirà una finestra "Opzioni di esportazione dei formati biologici". Selezionare OK.
NOTA: Assicurarsi che solo i TIFF OME-TIFF in bianco e nero appena regolati siano salvati nella cartella di origine prima di procedere con l'analisi COMSTAT. Rimuovere tutti i file OME-TIFF originali dalla cartella.
- Nella finestra dell'immagine, regolare il cursore all'estrema destra (cioè il livello più in alto del biofilm). Per rimuovere il rumore di fondo, utilizzare la finestra "Soglia", che visualizza un istogramma dell'immagine, per impostare manualmente i valori di soglia massima e minima. Innanzitutto, imposta il valore di soglia massimo regolando il cursore inferiore il più a destra possibile. In secondo luogo, utilizzare il cursore superiore per regolare il valore di soglia minimo, che segmenta l'immagine in due fasi separate: biomassa rossa e sfondo in scala di grigi (Figura 3).
- Esegui COMSTAT selezionando Plugin | Comstat2 |. Apparirà una finestra "Informazioni". Selezionare OK. Appariranno tre finestre.
- Nella finestra "Directory osservate" (in alto a destra), seleziona Aggiungi. Individua la cartella di origine, evidenziala senza aprirla e seleziona Scegli. Apparirà una finestra 'Immagini nelle directory' (in alto a sinistra) che elenca gli OME-TIFF da analizzare tramite COMSTAT.
- Nella finestra "Comstat 2.1" (in basso a destra), deselezionare "Soglia automatica (metodo di Otsu)" per assicurarsi che il software utilizzi valori di soglia precedentemente impostati per i singoli OME-TIFF. Inoltre, deselezionare il filtro a volume collegato(CVF) per garantire che le parti molto sottili del biofilm, nonché le cellule o la biomassa libere che si trovano all'interno dei vuoti della struttura del biofilm siano incluse nell'analisi.
NOTA: In questo caso, deselezionare la CVF perché l'analisi COMSTAT viene eseguita su biofilm molto precoci (24 ore di crescita iniziale) e sulle cellule/colonie planctoniche rimanenti dopo il trattamento antimicrobico. Per i biofilm maturi, selezionare il CVF per garantire che venga quantificata solo la biomassa collegata alla struttura del biofilm. - Nella finestra "Comstat 2.1" (in basso a destra), selezionare le funzioni desiderate per l'analisi quantitativa. Qui, seleziona Bio Massa, Distribuzione dello spessore e Area della superficie. Selezionare Vai per eseguire il programma. Nella finestra 'Registro' (in basso a sinistra), i dati di output vengono visualizzati in elaborazione fino a quando non viene visualizzato 'Fine con le funzioni/immagini selezionate!'. Registra le misurazioni COMSTAT. Queste misurazioni vengono inoltre salvate automaticamente come file TXT nella cartella di origine.
Risultati
Un isolato di P. aeruginosa coltivato da un paziente infetto con FC viene utilizzato per dimostrare i punti di forza di questo approccio nel quantificare accuratamente i cambiamenti indotti da antimicrobici nell'architettura del biofilm in vitro . Il flusso di lavoro complessivo di questo modello è rappresentato nella Figura 1. La procedura di elaborazione delle immagini e di analisi COMSTAT in ImageJ è illustrata nella Figura 2. Nella
Discussione
Non esiste un metodo prescritto per confrontare quantitativamente immagini tridimensionali di strutture di biofilm in vitro e le procedure descritte in questo contesto sono spesso difficili da standardizzare a causa della variabilità interoperatore20. Pertanto, questo protocollo offre un quadro semplice e riproducibile per le applicazioni COMSTAT che cercano di quantificare i cambiamenti nell'architettura del biofilm in vitro in condizioni antimicrobiche variabili. I punti di fo...
Divulgazioni
Nessuno
Riconoscimenti
Gli autori desiderano ringraziare la Cystic Fibrosis Foundation per aver fornito finanziamenti per questa ricerca.
Materiali
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
Riferimenti
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon