Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Quantifizierung der Auswirkungen von Antibiotika auf die In-vitro-Biofilmarchitektur mit der COMSTAT-Software
In diesem Artikel
Zusammenfassung
Antimikrobiell induzierte Veränderungen der Biofilmarchitektur von Pseudomonas aeruginosa unterscheiden sich zwischen klinischen Isolaten, die von Patienten mit Mukoviszidose und chronischer Lungeninfektion kultiviert wurden. Nach der konfokalen Mikroskopie kann die COMSTAT-Software verwendet werden, um Variationen in der Biofilmarchitektur (z. B. Oberfläche, Dicke, Biomasse) für einzelne Isolate zu quantifizieren und so die Wirksamkeit von Antiinfektiva zu bewerten.
Zusammenfassung
Biofilme sind Aggregate von Mikroorganismen, die auf einer selbst produzierten Matrix aus extrazellulärer Polymersubstanz zum Schutz und zur strukturellen Integrität beruhen. Es ist bekannt, dass der nosokomiale Erreger Pseudomonas aeruginosa einen Biofilm-Wachstumsmodus annimmt, der bei Patienten mit Mukoviszidose (CF) eine chronische Lungeninfektion verursacht. Das Computerprogramm COMSTAT ist ein nützliches Werkzeug zur Quantifizierung antimikrobiell induzierter Veränderungen in der Biofilmarchitektur von P. aeruginosa , indem Daten aus dreidimensionalen konfokalen Bildern extrahiert werden. Die standardisierte Bedienung der Software wird jedoch weniger häufig angesprochen, was für eine optimale Berichterstattung über das Biofilmverhalten und einen zentrumsübergreifenden Vergleich wichtig ist. Ziel dieses Protokolls ist es daher, einen einfachen und reproduzierbaren Rahmen für die Quantifizierung von In-vitro-Biofilmstrukturen unter unterschiedlichen antimikrobiellen Bedingungen über COMSTAT bereitzustellen. Die Technik wird unter Verwendung eines CF P. aeruginosa-Isolats modelliert, das in Form von Biofilmreplikaten gezüchtet und Tobramycin und dem monoklonalen Anti-Psl-Antikörper Psl0096 ausgesetzt wurde. Der Schritt-für-Schritt-Ansatz zielt darauf ab, die Mehrdeutigkeit der Benutzer zu reduzieren und die Wahrscheinlichkeit zu minimieren, wichtige Bildverarbeitungsschritte zu übersehen. Insbesondere betont das Protokoll die Eliminierung subjektiver Variationen, die mit der manuellen Bedienung von COMSTAT verbunden sind, einschließlich der Bildsegmentierung und der Auswahl geeigneter quantitativer Analysefunktionen. Obwohl diese Methode erfordert, dass Benutzer zusätzliche Zeit für die Verarbeitung konfokaler Bilder aufwenden, bevor sie COMSTAT ausführen, hilft sie, falsch dargestellte Biofilmheterogenität in automatisierten Ausgaben zu minimieren.
Einleitung
Biofilme sind Aggregate von Mikroorganismen, die in einer Matrix aus selbst produzierten extrazellulären polymeren Substanzen (EPS) orientiert sind. Die EPS-Matrix ist sehr komplex und besteht hauptsächlich aus Bakterienzellen, Wasser, Proteinen, Polysacchariden, Lipiden und Nukleinsäuren1, die sich alle deutlich von freilebenden Planktonzellen unterscheiden. Biofilm-EPS haften aneinander und an verschiedenen Oberflächen. Die EPS-Matrix hat Eigenschaften, die den Zell-zu-Zell-Austausch von Metaboliten, genetischem Material und Verbindungen, die für die interzelluläre Signalübertragung und Abwehr verwendet werden, vermitteln2. Diese Eigenschaften bieten zusammen die strukturelle Integrität der Biofilme und Schutz vor äußeren Stressoren und tragen zur Immunevasion und antimikrobiellen Resistenz bei3.
Pseudomonas aeruginosa ist ein anerkannter nosokomialer Krankheitserreger, der dafür bekannt ist, als Reaktion auf antimikrobielle Mittel eine ausweichende Biofilmwachstumsstrategie zu verfolgen. Ein Paradebeispiel dafür ist bei Patienten mit der rezessiven Erbkrankheit Mukoviszidose (CF). Biofilme spielen eine zentrale Rolle bei der Entwicklung von antimikrobiell resistenten P. aeruginosa4 und ermöglichen die Etablierung einer chronischen Lungeninfektion bei Patienten mit Mukoviszidose, was zu einer beschleunigten Verschlechterung der Lungenfunktion und vorzeitiger Mortalität führt5. Daher werden In-vitro-Biofilmstudien durchgeführt, um die Wirksamkeit von Antibiotika und neuen Antiinfektiva gegen P. aeruginosa-Isolate von Patienten mit CF 6,7 zu testen. Nach der Biofilmbildung werden antimikrobielle Mittel extern auf die Struktur aufgebracht, und die konfokale Laser-Scanning-Mikroskopie (CLSM) wird verwendet, um hochauflösende, dreidimensionale Rekonstruktionen von Biofilmsegmenten zu erzeugen. Es ist üblich, dann die Computersoftware COMSTAT als Plugin für ImageJ zu verwenden, um Veränderungen in der Biofilmarchitekturzu quantifizieren 8,9,10,11.
Obwohl COMSTAT für die Quantifizierung der Biofilmstruktur nützlich ist, wird die Reproduzierbarkeit und Standardisierung der Bildanalyse weniger häufig angesprochen. So ist z. B. die Bildverarbeitung, die vor dem Ausführen von COMSTAT durchgeführt wurde, objektiv, enthält aber ein Element der Subjektivität bei der Festlegung der Bildschwellenwerte12,13. In ähnlicher Weise ermöglicht das COMSTAT-Programm dem Bediener, zwischen grundlegenden und erweiterten Bedingungen und Parametern für die Bildsegmentierung sowie zwischen zehn quantitativen Analysefunktionen (z. B. Dickenverteilung, Oberfläche, Biomasse, dimensionsloser Rauheitskoeffizient) zu wählen. Die Vielzahl der Benutzeroptionen, kombiniert mit unterschiedlichen Fachkenntnissen der Bediener, kann zu einer fehlgeleiteten Berichterstattung über das Verhalten von Biofilmen führen.
Das Ziel dieses Protokolls ist es daher, eine relativ einfache Methode zum quantitativen Vergleich von in vitro Biofilmstrukturen mit COMSTAT vorzustellen. Hierin werden dreidimensionale Bilder von Biofilmsegmenten aus einem CF P. aeruginosa-Isolat mittels CLSM unter Verwendung des kammerförmigen Deckglasmodells14 aufgenommen – einer etablierten Technik, die zur Durchführung reproduzierbarer In-vitro-Biofilmexperimente verwendet wird. Durch die Verwendung von COMSTAT als Plugin für ImageJ ermöglicht diese Methode den Forschern, Veränderungen in der Biofilmarchitektur in Gegenwart von antimikrobiellen Mitteln unter verschiedenen Bedingungen quantitativ zu identifizieren. Insgesamt zielt diese Methode darauf ab, subjektive Variationen, die mit der manuellen Bedienung von COMSTAT verbunden sind, zu eliminieren und so die Standardisierung von Protokollen über Zentren hinweg zu erleichtern.
Protokoll
1. Entnahme von Bakterienisolat
- Beziehen Sie P. aeruginosa-Isolate aus einer Kohorte von pädiatrischen Patienten mit Mukoviszidose, die sich einer Eradikationsbehandlung mit inhalativem Tobramycin bei SickKids (Toronto) unterziehen. Frieren Sie die Isolate vor der Verwendung mindestens dreimal bei -80 °C in Glycerincitrat und Subkultur ein.
2. Bildung von In-vitro-Biofilmen
HINWEIS: Verwenden Sie eine Kammer-Deckglasmethode1 für die In-vitro-Biofilmbildung mit Modifikationen. Der gesamte Arbeitsablauf dieses Modells ist in Abbildung 1 dargestellt.
- P. aeruginosa-Isolat über Nacht bei 37 °C auf Blutagar züchten, das mit tryptischem Sojaagar und 5 % Schafsblut hergestellt wurde (siehe Materialtabelle).
- Impfen Sie 1–2 Bakterienkolonien aus dem Blutagar in 4 ml Lysogenese-Bouillon (LB). Anbau über Nacht bei 37 °C auf einem Shaker mit 225 U/min.
- Bereiten Sie eine Verdünnung des Overnight-Inokulums von 1:100 vor, indem Sie 40 μl der Kultur in 4 mL frisches LB geben. Wachsen Sie 3–4 Stunden lang bei 37 °C auf einem Shaker mit 225 U/min, um eine optische Dichte von ca. 0,1 bei 600 nm (OD600) zu erreichen (frühe logarithmische Phase).
- Übertragen Sie 220 μl des Inokulums in jede Vertiefung eines 8-Kammer-Deckglasobjektträgers. Ungestört bei 37 °C für 24 h inkubieren.
- Entfernen Sie das Medium langsam aus jeder Vertiefung, um zu verhindern, dass sich die Biofilme an der Basis ablösen.
HINWEIS: Kippen Sie den Schieber in einem Winkel von 45° nach vorne und saugen Sie das Medium aus den unteren Ecken jeder Vertiefung an, ohne die Basis mit der Pipettenspitze zu berühren. - Bereiten Sie 100 μl 56 μg/ml fluoreszenzmarkierten monoklonalen Antikörper (mAb) vor und geben Sie ihn langsam an die Seite der dafür vorgesehenen Kammer-Wells. 1 h bei Raumtemperatur (RT) inkubieren, um die Bindung von Antikörpern an das bakterielle Antigenepitop zu ermöglichen.
HINWEIS: Verdünnen Sie vor der Verwendung den fluoreszierend markierten (roten) mAb, Psl0096, in LB, um eine Endkonzentration von 56 μg/ml zu erhalten. Psl0096 ist ein Anti-Psl-mAb (optimiertes Affinitätsderivat von Cam003), das an das Klasse-I-Psl-Epitop bindet – eine wichtige EPS-Matrixkomponente von P. aeruginosa-Biofilmen, die an der anfänglichen Zelladhäsion und der strukturellen Integrität beteiligt sind15. - Bereiten Sie 100 μl einer 1000 μg/ml Antibiotikalösung vor und geben Sie sie langsam an die Seite der dafür vorgesehenen Vertiefungen. Ungestört bei 37 °C für 24 h inkubieren.
HINWEIS: Verdünnen Sie vor der Anwendung eine Brühe von 50 mg/ml Tobramycin-Antibiotikum in LB, um eine Endkonzentration von 1000 μg/ml zu erhalten.
3. Fluoreszenzfärbung von Biofilmen
- Bereiten Sie eine 0,01 mM Lösung eines lebendzellfärbenden Fluoreszenzfarbstoffs vor. Entfernen Sie das Medium langsam aus den gekammerten Vertiefungen und geben Sie 200 μl der Farbstoffmischung in jede Vertiefung. Inkubieren Sie bei RT im Dunkeln für 45 min.
HINWEIS: Bereiten Sie vor der Verwendung den Fluoreszenzfarbstoff für die Lebendzellfärbung (grün) vor, indem Sie 4 μl eines 5 mM-Stammmaterials zu 2 mL LB hinzufügen. - Entfernen Sie langsam das Medium aus jeder Vertiefung und waschen Sie es 2x mit 200 μl frischem LB.
- Geben Sie 200 μl frisches LB in jede Vertiefung und fahren Sie mit der Untersuchung durch konfokale Mikroskopie fort.
4. Bildaufnahme durch konfokale Mikroskopie
HINWEIS: Das Verfahren zur Bildverarbeitung und COMSTAT-Analyse ist in Abbildung 2 dargestellt. Erfassen Sie Bilder von Vertiefungen am selben Tag, an dem der Biofilm gefärbt wurde. Wenn die Verzögerung bei der Visualisierung 1 h überschreitet, kühlen Sie das Deckglas bis zur weiteren Verarbeitung im Dunkeln.
- Erfassen Sie Bilder von Vertiefungen mit einem konfokalen Mikroskopsystem (siehe Materialtabelle) mit geeigneten Laseranregungswellenlängen und Filtersätzen für die Erfassung.
HINWEIS: Stimulieren Sie hier die fluoreszenzmarkierte (rote) mAb-Färbung und die Lebendzellfärbung (grün) mit Anregungswellenlängen von 561 nm bzw. 491 nm. - Erfassen Sie geschichtete Z-Stapel-Bilder (vom Substrat bis zur Spitze jedes Biofilmsegments) in Schritten von 0,3 μm mit einer 20- bis 25-fachen Wasserimmersionslinse. Nehmen Sie mindestens 6 Bildstapel pro Well.
HINWEIS: Visualisieren Sie hier die Bilder mit einer hochauflösenden Kamera mit einer 25-fachen Wasserimmersionslinse und bearbeiten Sie sie mit einer Bildanalysesoftware (siehe Materialtabelle). Halten Sie die Parameter für die Softwareeinrichtung und die digitale Bildgebung (d. h. Helligkeit und Empfindlichkeit) für alle Aufnahmen in einem einzigen Experiment konstant. - Speichern Sie Bilder als OME-TIFFs für die COMSTAT-Analyse.
HINWEIS: Stellen Sie sicher, dass OME-TIFFs für jeden Kanal separat gespeichert werden (d. h. rot und grün). Dieser Schritt variiert je nach verwendeter Bildanalysesoftware. - Wiederholen Sie die Schritte 2.1 bis 4.3, um Bilder von insgesamt 3 biologischen Replikaten (d. h. 3 unabhängigen Experimenten) pro Bakterienisolat aufzunehmen.
5. COMSTAT-Analyse
HINWEIS: Analysieren Sie Bilder quantitativ mit dem frei verfügbaren Computerprogramm COMSTAT 16,17, das als Plugin (Comstat2) in ImageJ umgeschrieben wurde. Lesen Sie die allgemeinen Anweisungen zur Analyse von Bildstapeln von Biofilmen innerhalb des heruntergeladenen Pakets. Dieser Beitrag enthält ein zusammengefasstes Protokoll mit ausgewählten ImageJ-Verarbeitungsschritten und COMSTAT-Funktionen, die zur Quantifizierung der Auswirkungen von Antibiotika auf die Biofilmbildung empfohlen werden.
- Laden Sie das Comstat2-Paket von http://www.comstat.dk/ herunter. Suchen Sie im installierten Ordner ImageJ und führen Sie es aus.
- Erstellen Sie einen Quellordner auf dem Desktop, und fügen Sie dem Ordner ein einzelnes OME-TIFF hinzu.
- Öffnen Sie OME-TIFF aus dem Quellordner, und löschen Sie alle leeren Layer, die keine Biomasse enthalten. Bei diesen Layern handelt es sich entweder um die ersten oder die letzten Layer des Z-Stapels.
HINWEIS: Mikroskopdefinierte Z-Stapel-Grenzen werden von Benutzern manchmal überschätzt. Durch das Löschen dieser leeren Layer wird eine verfeinerte Z-Stapel-Grenze für die COMSTAT-Analyse erstellt. - Importieren Sie OME-TIFF in ImageJ, indem Sie Datei | Importieren | Bildsequenz. Suchen Sie den Quellordner, markieren Sie ihn, ohne ihn zu öffnen, und klicken Sie auf Auswählen. Das Fenster "Sequenzoptionen" wird angezeigt. Wählen Sie OK aus.
HINWEIS: Um weitere Bilder in ImageJ zu importieren, entfernen Sie zuerst das vorherige OME-TIFF aus dem Quellordner, fügen Sie dann das neue OME-TIFF zum Ordner hinzu und wiederholen Sie die Schritte 5.3 und 5.4. - Kehren Sie die Ausrichtung des Biofilms um, indem Sie Bild | Transformieren | Drehen Sie Z um, um das Substrat als ersten (obersten) Stapel zu positionieren.
HINWEIS: COMSTAT-Algorithmen lesen Biofilme in z-Richtung von oben (Stapel 1) nach unten. Je nach verwendetem konfokalem Mikroskopsystem kann die OME-TIFF-Ausgabe invertiert werden. Daher ist es wichtig, die Reihenfolge der Slices umzukehren, indem das Substrat als Bildstapel 1 positioniert wird, um zu verhindern, dass die Ausgabedaten fehlerhaft werden. - Definieren Sie Bildeigenschaften, indem Sie Bild | Eigenschaften. Das Fenster "Quelle" wird angezeigt.
- Geben Sie "Längeneinheit" als "Mikrometer" an.
- Bestimmen Sie mathematisch "Pixelbreite" und "Pixelhöhe" mit der folgenden Gleichung:
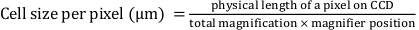 (1)
(1)
Hier sind "Pixelbreite" und "Pixelhöhe" definiert als 0,427, wobei die physikalische Länge eines Pixels auf dem ladungsgekoppelten Bauelement (CCD) 16 μm beträgt; Die Gesamtvergrößerung beträgt das 25-fache; und die Position der Lupe ist 1,5x.
HINWEIS: Die Gleichung, die zur Berechnung der Zellengröße pro Pixel verwendet wird, kann je nach Hersteller der Mikroskopkamera variieren. Alternativ können "Pixelbreite" und "Pixelhöhe" durch räumliche Kalibrierung definiert werden (siehe https://imagej.net/Spatial_Calibration). - Definieren Sie "Voxeltiefe" als 0,3 (d. h. inkrementellen Abstand zwischen den einzelnen Z-Stack-Schichten). Wählen Sie OK im Fenster "Quelle".
- Passen Sie den Bildschwellenwert an, indem Sie Bild | Anpassen | Schwelle. Das Fenster "Schwellenwert" wird angezeigt.
HINWEIS: Objekte im Bild werden rot mit einem Graustufenhintergrund angezeigt. Alternativ kann der Schwellenwert in Schwarzweiß eingestellt werden, indem Sie das Dropdown-Menü im Fenster "Schwellenwert" verwenden, um "Rot" in "S/W" zu ändern.- Stellen Sie den Schieberegler im Bildfenster ganz nach rechts ein (d. h. auf die oberste Schicht des Biofilms). Um Hintergrundgeräusche zu entfernen, verwenden Sie das Fenster "Schwellenwert", in dem ein Histogramm des Bildes angezeigt wird, um die maximalen und minimalen Schwellenwerte manuell festzulegen. Stellen Sie zunächst den maximalen Schwellenwert ein, indem Sie den unteren Schieberegler so weit wie möglich nach rechts stellen. Zweitens verwenden Sie den oberen Schieberegler, um den minimalen Schwellenwert anzupassen, der das Bild in zwei separate Phasen unterteilt: rote Biomasse und Graustufenhintergrund (Abbildung 3).
HINWEIS: Das hierin beschriebene In-vitro-Biofilmbildungs- und Fluoreszenzmikroskopieverfahren erzeugt im Idealfall OME-TIFFs, was die Segmentierung von Bildern in zwei verschiedene Phasen mittels einer einfachen Histogramm-Schwellenmethode ermöglicht. In einigen Fällen ist die Unterscheidung zwischen den verschiedenen Phasen im Histogramm jedoch nicht so klar. Dies kann auf das Vorhandensein von starkem Hintergrundrauschen, unterschiedlichen Hintergrundintensitäten oder einen geringen Intensitätskontrast zwischen Biomasse und Hintergrund zurückzuführen sein. In solchen Fällen sollten die Nutzer ein erweitertes Segmentierungsverfahren anwenden18,19.- Alternativ können Sie die Schwellenwerte für einzelne Bilder algorithmisch anpassen, indem Sie das linke Dropdown-Menü verwenden, das im Fenster "Schwellenwert" auf "Standard" eingestellt ist. Diese Funktion bietet 17 verschiedene algorithmische Schwellenwertoptionen zur Auswahl (siehe https://imagej.net/Auto_Threshold). Wählen Sie die am besten zutreffende Option und dann "Auto", um den Schwellenwert festzulegen.
- Wenn die Schwellenwerte angepasst sind, verwenden Sie den Schieberegler im Bildfenster, um durch die einzelnen Ebenen zu scrollen, um sicherzustellen, dass Hintergrundgeräusche durchgehend ausreichend entfernt werden.
- Wählen Sie im Fenster "Schwellenwert" die Option "Festlegen ", um zuerst den unteren Schwellenwert festzulegen. Das Fenster "Schwellenwerte festlegen" wird angezeigt. Wählen Sie OK aus. Wählen Sie erneut Festlegen aus, und wiederholen Sie diesen Schritt, um den maximalen Schwellenwert festzulegen.
HINWEIS: Jedes Mal, wenn "Einstellen" ausgewählt wird, kann sich der untere Schieberegler automatisch anpassen. Stellen Sie in solchen Fällen den Schieberegler manuell neu ein (oder wählen Sie "Auto", wenn Sie einen der algorithmischen Schwellenwerte verwenden) und wiederholen Sie die Schritte 5.6.2 bis 5.6.3. Die Grundidee ist, dass bei jeder automatischen Neueinstellung der Schieberegler "Setzen" danach zwei weitere Male gewählt werden sollte, um sicherzustellen, dass sowohl der obere als auch der untere Schwellenwert fest sind. - Wählen Sie Anwenden aus, und das Fenster "Stack in Binary konvertieren" wird angezeigt. Wählen Sie OK und schließen Sie dann das Fenster "Schwellenwert".
- Speichern Sie das neu angepasste OME-TIFF, indem Sie Plugins | Bio-Formate | Exporteur von Bioformaten|. Geben Sie einen neuen Dateinamen ein und speichern Sie sie als OME-TIFF im Quellordner. Das Fenster "Bio-Formats Exporter – Mehrere Dateien" wird angezeigt. Wählen Sie OK aus. Das Fenster "Optionen für den Export von Bioformaten" wird angezeigt. Wählen Sie OK aus.
HINWEIS: Stellen Sie sicher, dass nur die neu angepassten Schwarzweiß-OME-TIFFs im Quellordner gespeichert werden, bevor Sie mit der COMSTAT-Analyse fortfahren. Entfernen Sie alle ursprünglichen OME-TIFFs aus dem Ordner.
- Stellen Sie den Schieberegler im Bildfenster ganz nach rechts ein (d. h. auf die oberste Schicht des Biofilms). Um Hintergrundgeräusche zu entfernen, verwenden Sie das Fenster "Schwellenwert", in dem ein Histogramm des Bildes angezeigt wird, um die maximalen und minimalen Schwellenwerte manuell festzulegen. Stellen Sie zunächst den maximalen Schwellenwert ein, indem Sie den unteren Schieberegler so weit wie möglich nach rechts stellen. Zweitens verwenden Sie den oberen Schieberegler, um den minimalen Schwellenwert anzupassen, der das Bild in zwei separate Phasen unterteilt: rote Biomasse und Graustufenhintergrund (Abbildung 3).
- Führen Sie COMSTAT aus, indem Sie Plugins | Comstat2 |. Es erscheint ein "Info"-Fenster. Wählen Sie OK aus. Es werden drei Fenster angezeigt.
- Wählen Sie im Fenster "Beobachtete Verzeichnisse" (oben rechts) die Option "Hinzufügen" aus. Suchen Sie den Quellordner, markieren Sie ihn, ohne ihn zu öffnen, und wählen Sie Auswählen. Es erscheint ein Fenster 'Bilder in Verzeichnissen' (oben links), in dem die OME-TIFFs aufgelistet sind, die über COMSTAT analysiert werden sollen.
- Deaktivieren Sie im Fenster "Comstat 2.1" (unten rechts) die Option "Automatische Schwellenwerte (Otsu-Methode)", um sicherzustellen, dass die Software Schwellenwerte verwendet, die zuvor für einzelne OME-TIFFs eingerichtet wurden. Deaktivieren Sie außerdem den Connected Volume Filtering (CVF), um sicherzustellen, dass sehr dünne Teile des Biofilms sowie frei schwebende Zellen oder Biomasse, die sich in Hohlräumen der Biofilmstruktur befinden, in die Analyse einbezogen werden.
HINWEIS: Deaktivieren Sie hier CVF, da die COMSTAT-Analyse an sehr frühen Biofilmen (24 h anfängliches Wachstum) und verbleibenden planktonischen Zellen/Kolonien nach antimikrobieller Behandlung durchgeführt wird. Für reife Biofilme wählen Sie CVF, um sicherzustellen, dass nur Biomasse, die mit der Biofilmstruktur verbunden ist, quantifiziert wird. - Wählen Sie im Fenster "Comstat 2.1" (unten rechts) die gewünschten Merkmale für die quantitative Analyse aus. Wählen Sie hier Biomasse, Dickenverteilung und Oberfläche aus. Wählen Sie Gehe zu , um das Programm auszuführen. Im 'Log'-Fenster (unten links) werden die Ausgabedaten in Bearbeitung angezeigt, bis 'Fertig mit ausgewählten Funktionen/Bildern!' erscheint. Zeichnen Sie die COMSTAT-Messungen auf. Diese Messungen werden auch automatisch als TXT-Dateien im Quellordner gespeichert.
Ergebnisse
Ein P. aeruginosa-Isolat , das von einem infizierten Patienten mit CF kultiviert wurde, wird verwendet, um die Stärken dieses Ansatzes bei der genauen Quantifizierung von antimikrobiell induzierten Veränderungen in der In-vitro-Biofilmarchitektur zu demonstrieren. Der gesamte Arbeitsablauf dieses Modells ist in Abbildung 1 dargestellt. Das Bildverarbeitungs- und COMSTAT-Analyseverfahren in ImageJ ist in Abbildung 2 dargestellt.
Diskussion
Es gibt keine vorgeschriebene Methode zum quantitativen Vergleich von dreidimensionalen Bildern von in vitro Biofilmstrukturen, und die in diesem Zusammenhang beschriebenen Verfahren sind aufgrund der Variabilität zwischen den Betreibern oft schwer zu standardisieren20. Somit bietet dieses Protokoll einen einfachen und reproduzierbaren Rahmen für COMSTAT-Anwendungen, die darauf abzielen, Veränderungen in der In-vitro-Biofilmarchitektur unter unterschiedlichen antimikrobiellen ...
Offenlegungen
Nichts
Danksagungen
Die Autoren danken der Cystic Fibrosis Foundation für die Finanzierung dieser Forschung.
Materialien
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
Referenzen
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten