Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Quantification des effets des antimicrobiens sur l’architecture des biofilms in vitro à l’aide du logiciel COMSTAT
Dans cet article
Résumé
Les modifications induites par les antimicrobiens de l’architecture du biofilm de Pseudomonas aeruginosa diffèrent entre les isolats cliniques cultivés chez des patients atteints de mucoviscidose et d’infection pulmonaire chronique. Après la microscopie confocale, le logiciel COMSTAT peut être utilisé pour quantifier les variations de l’architecture du biofilm (p. ex., surface, épaisseur, biomasse) pour des isolats individuels afin d’évaluer l’efficacité des agents anti-infectieux.
Résumé
Les biofilms sont des agrégats de micro-organismes qui dépendent d’une matrice autoproduite de substance polymère extracellulaire pour la protection et l’intégrité structurelle. L’agent pathogène nosocomial, Pseudomonas aeruginosa, est connu pour adopter un mode de croissance de biofilm, provoquant une infection pulmonaire chronique chez les patients atteints de mucoviscidose (FK). Le programme informatique COMSTAT est un outil utile pour quantifier les changements induits par les antimicrobiens dans l’architecture du biofilm de P. aeruginosa en extrayant des données à partir d’images confocales tridimensionnelles. Cependant, le fonctionnement standardisé du logiciel est moins souvent abordé, ce qui est important pour un rapport optimal du comportement du biofilm et une comparaison intercentrique. Ainsi, l’objectif de ce protocole est de fournir un cadre simple et reproductible pour quantifier les structures de biofilms in vitro dans diverses conditions antimicrobiennes via COMSTAT. La technique est modélisée à l’aide d’un isolat de P. aeruginosa de la mucoviscidose, cultivé sous forme de réplicats de biofilm et exposé à la tobramycine et à l’anticorps monoclonal anti-Psl, Psl0096. L’approche étape par étape vise à réduire l’ambiguïté de l’utilisateur et à minimiser le risque de négliger des étapes cruciales de traitement d’image. Plus précisément, le protocole met l’accent sur l’élimination des variations subjectives associées au fonctionnement manuel de COMSTAT, y compris la segmentation d’images et la sélection de fonctions d’analyse quantitative appropriées. Bien que cette méthode oblige les utilisateurs à passer plus de temps à traiter des images confocales avant d’exécuter COMSTAT, elle permet de minimiser l’hétérogénicité erronée des biofilms dans les sorties automatisées.
Introduction
Les biofilms sont des agrégats de micro-organismes orientés dans une matrice de substances polymères extracellulaires (EPS) autoproduites. La matrice EPS est très complexe, composée principalement de cellules bactériennes, d’eau, de protéines, de polysaccharides, de lipides et d’acides nucléiques1, qui rendent tous les biofilms nettement différents des cellules planctoniques libres. Les biofilms EPS sont adhérents les uns aux autres et à diverses surfaces. La matrice EPS possède des propriétés qui interviennent dans l’échange de métabolites, de matériel génétique et de composés utilisés pour la signalisation et la défense intercellulaires2. Ces propriétés fournissent collectivement aux biofilms une intégrité structurelle et une protection contre les facteurs de stress externes, contribuant à l’évasion immunitaire et à la résistance aux antimicrobiens3.
Pseudomonas aeruginosa est un pathogène nosocomial bien connu, connu pour adopter une stratégie de croissance évasive du biofilm en réponse aux antimicrobiens. Un excellent exemple de cela se produit chez les patients atteints de la maladie génétique récessive, la mucoviscidose (FK). Les biofilms jouent un rôle central dans le développement de P. aeruginosa4 résistant aux antimicrobiens et permettent l’établissement d’une infection pulmonaire chronique chez les patients atteints de mucoviscidose, entraînant un déclin accéléré de la fonction pulmonaire et une mortalité prématurée5. Par conséquent, des études in vitro sur biofilm sont réalisées pour tester l’efficacité des antibiotiques et de nouveaux agents anti-infectieux contre les isolats de P. aeruginosa obtenus chez des patients atteints de FC 6,7. Après la formation du biofilm, les antimicrobiens sont appliqués à l’extérieur de la structure, et la microscopie confocale à balayage laser (CLSM) est utilisée pour générer des reconstructions tridimensionnelles à haute résolution des segments de biofilm. Il est courant d’utiliser ensuite le logiciel informatique, COMSTAT, en tant que plug-in à ImageJ, pour quantifier les changements dans l’architecture du biofilm 8,9,10,11.
Bien que COMSTAT soit utile pour quantifier la structure du biofilm, la reproductibilité et la standardisation de l’analyse d’images sont moins souvent abordées. Par exemple, la procédure de traitement d’image, effectuée avant l’exécution de COMSTAT, est objective, mais contient un élément de subjectivité lors de la définition des seuils d’image12,13. De la même manière, le programme COMSTAT permet à l’opérateur de choisir des conditions et des paramètres de base à avancés pour la segmentation d’images ainsi que dix fonctions d’analyse quantitative (par exemple, la distribution de l’épaisseur, la surface, la biomasse, le coefficient de rugosité sans dimension). La multitude d’options d’utilisation, combinée aux différents niveaux d’expertise de l’opérateur, peut entraîner des rapports erronés sur le comportement du biofilm.
Ainsi, l’objectif de ce protocole est de présenter une méthode relativement simple pour la comparaison quantitative des structures de biofilms in vitro à l’aide de COMSTAT. Ici, des images tridimensionnelles de segments de biofilm d’un isolat de P. aeruginosa de la FC sont capturées par CLSM à l’aide du modèle14 à chambre de protection, une technique établie utilisée pour réaliser des expériences in vitro reproductibles sur biofilm. Utilisant COMSTAT comme plug-in d’ImageJ, cette méthode permet aux chercheurs d’identifier quantitativement les changements dans l’architecture du biofilm en présence d’antimicrobiens dans diverses conditions. Dans l’ensemble, cette méthode vise à éliminer les variations subjectives associées au fonctionnement manuel de COMSTAT, facilitant ainsi la standardisation des protocoles entre les centres.
Protocole
1. Collecte d’isolats bactériens
- Obtenir des isolats de P. aeruginosa d’une cohorte de patients pédiatriques atteints de fibrose kystique subissant un traitement d’éradication par tobramycine inhalée à l’Hôpital SickKids (Toronto). Congeler les isolats à -80 °C dans du citrate de glycérol et une sous-culture au moins trois fois avant de les utiliser.
2. Formation du biofilm in vitro
REMARQUE : Utilisez une fenêtre de couverture à chambreméthode 1 pour la formation de biofilm in vitro avec modifications. Le flux de travail global de ce modèle est illustré à la figure 1.
- Cultiver l’isolat de P. aeruginosa pendant une nuit à 37 °C sur une gélose au sang préparée avec de la gélose tryptique de soja et 5 % de sang de mouton (voir le tableau des matières).
- Inoculer 1 à 2 colonies bactériennes de la gélose au sang dans 4 mL de bouillon de lysogénie (LB). Cultivez toute la nuit à 37 °C sur un agitateur réglé à 225 tr/min.
- Préparez une dilution de 1:100 de l’inoculum pendant la nuit en ajoutant 40 μL de culture dans 4 ml de LB frais. Cultiver pendant 3 à 4 h à 37 °C sur un agitateur réglé à 225 tr/min, pour obtenir une densité optique d’environ 0,1 à 600 nm (OD600) (phase logarithmique précoce).
- Transférez 220 μL de l’inoculum dans chaque puits d’une lame de recouvrement à 8 chambres. Incuber sans être dérangé à 37 °C pendant 24 h.
- Retirez lentement le milieu de chaque puits pour éviter que les biofilms ne se détachent à la base.
REMARQUE : Inclinez la glissière vers l’avant à un angle de 45° et aspirez le fluide par les coins inférieurs de chaque puits chambré sans toucher la base avec la pointe de la pipette. - Préparez et ajoutez lentement 100 μL d’anticorps monoclonaux (AcM) marqués par fluorescence à 56 μg/mL (voir le tableau des matériaux) sur le côté des puits chambrés désignés. Incuber à température ambiante (RT) pendant 1 h pour permettre la fixation de l’anticorps à l’épitope de l’antigène bactérien.
REMARQUE : Avant l’utilisation, diluer l’anticorps monoclonal marqué par fluorescence (rouge), Psl0096, dans LB pour obtenir une concentration finale de 56 μg/mL. Psl0096 est un mAb (dérivé d’affinité optimisée de Cam003), qui se lie à l’épitope Psl de classe I, un composant clé de la matrice EPS des biofilms de P. aeruginosa impliqué dans la fixation cellulaire initiale et l’intégrité structurelle15. - Préparez et ajoutez lentement 100 μL d’une solution d’antibiotique de 1000 μg/mL (voir le tableau des matériaux) sur le côté des puits chambrés désignés. Incuber sans être dérangé à 37 °C pendant 24 h.
REMARQUE : Avant l’utilisation, diluer une dose de 50 mg/mL d’antibiotique tobramycine dans LB pour obtenir une concentration finale de 1000 μg/mL.
3. Coloration fluorescente du biofilm
- Préparez une solution de 0,01 mM d’un colorant fluorescent colorant les cellules vivantes. Retirez lentement le fluide des puits chambrés et ajoutez 200 μL du mélange de colorant dans chaque puits. Incuber à RT dans l’obscurité pendant 45 min.
REMARQUE : Avant l’utilisation, préparez le colorant fluorescent de coloration des cellules vivantes (vert) en ajoutant 4 μL d’un stock de 5 mM à 2 mL de LB. - Retirez lentement le milieu de chaque puits et lavez-le 2 fois avec 200 μL de LB frais.
- Ajouter 200 μL de LB frais dans chaque puits et procéder à l’examen par microscopie confocale.
4. Acquisition d’images par microscopie confocale
REMARQUE : La procédure de traitement d’image et d’analyse COMSTAT est présentée à la figure 2. Acquérir des images des puits le jour même de la coloration du biofilm. Si le retard de visualisation dépasse 1 h, réfrigérer le verre de protection à chambre dans l’obscurité jusqu’à ce qu’il soit traité ultérieurement.
- Acquérir des images de puits à l’aide d’un système de microscope confocal (voir le tableau des matériaux) avec des longueurs d’onde d’excitation laser appropriées et des ensembles de filtres pour l’acquisition.
REMARQUE : Ici, excitez l’Acm marqué fluorescent (rouge) et la coloration de la cellule vivante (verte) en utilisant les longueurs d’onde d’excitation de 561 et 491 nm, respectivement. - Capturez des images z-stack superposées (du substrat au sommet de chaque segment de biofilm) par incréments de 0,3 μm avec un objectif d’immersion dans l’eau 20 à 25x. Prenez au moins 6 piles d’images par puits.
REMARQUE : Ici, visualisez les images à l’aide d’une caméra haute résolution avec un objectif d’immersion dans l’eau 25x et traitées à l’aide d’un logiciel d’analyse d’images (voir Tableau des matériaux). Maintenez la configuration du logiciel et les paramètres d’imagerie numérique (c’est-à-dire la luminosité et la sensibilité) constants pour toutes les acquisitions dans une seule expérience. - Enregistrez des images au format OME-TIFF pour l’analyse COMSTAT.
REMARQUE : Assurez-vous que les OME-TIFF sont enregistrés séparément pour chaque canal (c’est-à-dire rouge et vert). Cette étape varie en fonction du logiciel d’analyse d’images utilisé. - Répétez les étapes 2.1 à 4.3 pour capturer des images d’un total de 3 répétitions biologiques (c.-à-d. 3 expériences indépendantes) par isolat bactérien.
5. Analyse COMSTAT
REMARQUE : Analysez les images quantitativement à l’aide du programme informatique disponible gratuitement, COMSTAT16,17, réécrit en tant que plugin (Comstat2) pour ImageJ. Lisez les instructions générales pour l’analyse des piles d’images de biofilms dans le package téléchargé. Cette contribution fournit un protocole résumé, avec des étapes de traitement ImageJ sélectionnées et des fonctionnalités COMSTAT recommandées pour quantifier les effets des antimicrobiens sur la formation du biofilm.
- Téléchargez le package Comstat2 à partir de http://www.comstat.dk/. Dans le dossier installé, localisez ImageJ et exécutez-le.
- Créez un dossier source sur le bureau et ajoutez un seul OME-TIFF au dossier.
- Ouvrez OME-TIFF à partir du dossier source et supprimez toutes les couches vides ne contenant pas de biomasse. Ces couches seront soit la première soit la dernière couche de la pile z.
REMARQUE : Les limites de la pile z définies par le microscope sont parfois surestimées par les utilisateurs. La suppression de ces couches vides établit une limite de pile z plus précise pour l’analyse COMSTAT. - Importez OME-TIFF dans ImageJ en sélectionnant Fichier | Importation | Séquence d’images. Localisez le dossier source, mettez-le en surbrillance sans l’ouvrir, puis cliquez sur Sélectionner. Une fenêtre 'Options de séquence' apparaîtra. Sélectionnez OK.
REMARQUE : Pour importer des images supplémentaires dans ImageJ, supprimez d’abord l’ancien OME-TIFF du dossier source, puis ajoutez le nouvel OME-TIFF au dossier et répétez les étapes 5.3 et 5.4. - Inversez l’orientation du biofilm en sélectionnant Image | Transformer | Retournez la touche Z pour positionner le substrat en tant que première pile (la plus élevée).
REMARQUE : Les algorithmes COMSTAT lisent les biofilms dans la direction z du haut (pile 1) vers le bas. Selon le système de microscope confocal utilisé, la sortie OME-TIFF peut être inversée. Il est donc important d’inverser l’ordre des tranches en positionnant le substrat comme pile d’images 1 pour éviter que les données de sortie ne deviennent erronées. - Définissez les propriétés de l’image en sélectionnant Image | Propriétés. Une fenêtre « Source » apparaîtra.
- Spécifiez 'Unité de longueur' comme 'micron'.
- Déterminez mathématiquement la « largeur des pixels » et la « hauteur des pixels » à l’aide de l’équation suivante :
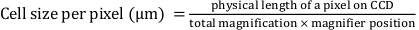 (1)
(1)
Ici, la « largeur de pixel » et la « hauteur de pixel » sont définies comme 0,427, où la longueur physique d’un pixel sur le dispositif à couplage de charge (CCD) est de 16 μm ; le grossissement total est de 25x ; et la position de la loupe est de 1,5x.
REMARQUE : L’équation utilisée pour calculer la taille de cellule par pixel peut varier en fonction du fabricant de la caméra du microscope. Alternativement, la 'Largeur des pixels' et la 'Hauteur des pixels' peuvent être définies par étalonnage spatial (voir https://imagej.net/Spatial_Calibration). - Définissez la « profondeur du voxel » comme 0,3 (c’est-à-dire l’espace incrémentiel entre chaque couche z-stack). Sélectionnez OK dans la fenêtre 'Source'.
- Ajustez le seuil d’image en sélectionnant Image | Ajuster | Seuil. Une fenêtre « Seuil » apparaîtra.
REMARQUE : Les objets de l’image apparaîtront en rouge avec un fond en niveaux de gris. Alternativement, le seuil peut être ajusté en noir et blanc, en utilisant le menu déroulant de la fenêtre « Seuil » pour changer « Rouge » en « N&B ».- Dans la fenêtre de l’image, ajustez le curseur à l’extrême droite (c’est-à-dire la couche supérieure du biofilm). Pour supprimer le bruit de fond, utilisez la fenêtre « Seuil », qui affiche un histogramme de l’image, pour définir manuellement les valeurs de seuil maximale et minimale. Tout d’abord, définissez la valeur de seuil maximale en ajustant le curseur inférieur aussi à droite que possible. Ensuite, utilisez le curseur supérieur pour ajuster la valeur de seuil minimale, qui segmente l’image en deux phases distinctes : biomasse rouge et arrière-plan en niveaux de gris (Figure 3).
REMARQUE : La procédure de formation de biofilm in vitro et de microscopie fluorescente décrite dans le présent document génère des OME-TIFF dans le cas idéal, ce qui permet de segmenter les images en deux phases distinctes au moyen d’une simple méthode de seuil d’histogramme. Cependant, dans certains cas, la distinction de l’histogramme entre les différentes phases n’est pas aussi claire. Cela peut être dû à la présence d’un bruit de fond important, à des intensités de fond variables ou à un contraste de faible intensité entre la biomasse et le bruit de fond. Dans de tels cas, les utilisateurs doivent adopter une procédure de segmentation renforcée18,19.- Vous pouvez également ajuster les seuils de manière algorithmique pour des images individuelles à l’aide du menu déroulant de gauche défini comme « Par défaut » dans la fenêtre « Seuil ». Cette fonctionnalité offre 17 options de seuil algorithmique différentes parmi lesquelles choisir (voir https://imagej.net/Auto_Threshold). Sélectionnez l’option la plus applicable, puis « Auto » pour définir le seuil.
- Lorsque les valeurs de seuil sont ajustées, utilisez le curseur dans la fenêtre de l’image pour faire défiler chaque calque afin de vous assurer que le bruit de fond est suffisamment supprimé.
- Sélectionnez Définir dans la fenêtre 'Seuil' pour corriger d’abord la valeur de seuil inférieure. Une fenêtre « Définir les niveaux de seuil » apparaîtra. Sélectionnez OK. Sélectionnez à nouveau Définir et répétez cette étape pour fixer la valeur de seuil maximale.
REMARQUE : Chaque fois que 'Set' est sélectionné, le curseur inférieur peut s’ajuster automatiquement. Dans ce cas, réajustez manuellement le curseur (ou sélectionnez « Auto » si vous utilisez l’un des seuils algorithmiques) et répétez les étapes 5.6.2 et 5.6.3. L’idée principale est que chaque fois que les curseurs sont réajustés automatiquement, « Set » doit être sélectionné deux fois de plus par la suite pour s’assurer que les seuils supérieur et inférieur sont fixes. - Sélectionnez Appliquer et une fenêtre « Convertir la pile en binaire » apparaîtra. Sélectionnez OK, puis quittez la fenêtre « Seuil ».
- Enregistrez l’OME-TIFF nouvellement ajusté en sélectionnant Plugins | Bio-formats | Exportateur de bio-formats|. Entrez un nouveau nom de fichier et enregistrez-le sous le nom OME-TIFF dans le dossier source. Une fenêtre « Exportateur de bio-formats – Plusieurs fichiers » apparaîtra. Sélectionnez OK. Une fenêtre 'Options d’exportation de bio-formats' apparaîtra. Sélectionnez OK.
REMARQUE : Assurez-vous que seuls les OME-TIFF en noir et blanc nouvellement ajustés sont enregistrés dans le dossier source avant de passer à l’analyse COMSTAT. Retirez tous les OME-TIFF originaux du dossier.
- Dans la fenêtre de l’image, ajustez le curseur à l’extrême droite (c’est-à-dire la couche supérieure du biofilm). Pour supprimer le bruit de fond, utilisez la fenêtre « Seuil », qui affiche un histogramme de l’image, pour définir manuellement les valeurs de seuil maximale et minimale. Tout d’abord, définissez la valeur de seuil maximale en ajustant le curseur inférieur aussi à droite que possible. Ensuite, utilisez le curseur supérieur pour ajuster la valeur de seuil minimale, qui segmente l’image en deux phases distinctes : biomasse rouge et arrière-plan en niveaux de gris (Figure 3).
- Exécutez COMSTAT en sélectionnant Plugins | Comstat2 |. Une fenêtre « À propos » apparaîtra. Sélectionnez OK. Trois fenêtres apparaîtront.
- Dans la fenêtre « Répertoires observés » (en haut à droite), sélectionnez Ajouter. Localisez le dossier source, mettez-le en surbrillance sans l’ouvrir, puis sélectionnez Choisir. Une fenêtre 'Images dans les répertoires' apparaîtra (en haut à gauche) qui répertorie les OME-TIFF à analyser via COMSTAT.
- Dans la fenêtre « Comstat 2.1 » (en bas à droite), désélectionnez « Seuillage automatique (méthode d’Otsu) » pour vous assurer que le logiciel utilise les valeurs de seuil précédemment configurées pour chaque OME-TIFF. De plus, désélectionnez l’option Connected Volume Filtering (CVF) pour vous assurer que les parties très minces du biofilm ainsi que les cellules flottantes ou la biomasse trouvées dans les vides de la structure du biofilm sont incluses dans l’analyse.
REMARQUE : Ici, désélectionnez le CVF car l’analyse COMSTAT est effectuée sur des biofilms très précoces (croissance initiale de 24 heures) et des cellules/colonies planctoniques restantes après le traitement antimicrobien. Pour les biofilms matures, sélectionner CVF pour s’assurer que seule la biomasse liée à la structure du biofilm est quantifiée. - Dans la fenêtre « Comstat 2.1 » (en bas à droite), sélectionnez les caractéristiques souhaitées pour l’analyse quantitative. Ici, sélectionnez Biomasse, Distribution de l’épaisseur et Surface. Sélectionnez Aller pour exécuter le programme. Dans la fenêtre 'Log' (en bas à gauche), les données de sortie sont affichées en traitement jusqu’à ce que 'Terminé avec les fonctions/images sélectionnées !' apparaisse. Enregistrez les mesures COMSTAT. Ces mesures sont également automatiquement enregistrées sous forme de fichiers TXT dans le dossier source.
Résultats
Un isolat de P. aeruginosa cultivé chez un patient infecté par la mucoviscidose est utilisé pour démontrer les forces de cette approche dans la quantification précise des changements induits par les antimicrobiens dans l’architecture du biofilm in vitro . Le flux de travail global de ce modèle est représenté à la figure 1. La procédure de traitement d’image et d’analyse COMSTAT dans ImageJ est illustrée à la figure 2. La
Discussion
Il n’existe pas de méthode prescrite pour comparer quantitativement les images tridimensionnelles des structures de biofilms in vitro , et les procédures décrites dans ce contexte sont souvent difficiles à normaliser en raison de la variabilité inter-opérateurs20. Ainsi, ce protocole offre un cadre simple et reproductible pour les applications COMSTAT cherchant à quantifier les changements dans l’architecture du biofilm in vitro dans diverses conditions antimicrobienne...
Déclarations de divulgation
Aucun
Remerciements
Les auteurs tiennent à remercier la Fondation de la fibrose kystique d’avoir financé cette recherche.
matériels
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
Références
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon