JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
COMSTAT 소프트웨어를 사용하여 체외 생물막 아키텍처에 대한 항균제의 효과 정량화
요약
녹농균 생물막 구조에 대한 항균 유도 변화는 낭포성 섬유증 및 만성 폐 감염 환자로부터 배양한 임상 분리물 간에 다릅니다. 컨포칼 현미경 검사 후 COMSTAT 소프트웨어를 사용하여 항감염제의 효능을 평가하기 위해 개별 분리체에 대한 생물막 구조(예: 표면적, 두께, 바이오매스)의 변화를 정량화할 수 있습니다.
초록
생물막은 보호 및 구조적 무결성을 위해 자체 생산된 세포외 고분자 물질 매트릭스에 의존하는 미생물의 집합체입니다. 병원내 병원체인 녹농균( Pseudomonas aeruginosa )은 낭포성 섬유증(CF) 환자에게 만성 폐 감염을 일으키는 생물막 성장 방식을 채택하는 것으로 알려져 있습니다. 컴퓨터 프로그램인 COMSTAT는 3차원 공초점 이미지에서 데이터를 추출하여 녹농균 생물막 구조에서 항균 유도 변화를 정량화하는 데 유용한 도구입니다. 그러나 소프트웨어의 표준화된 작동은 일반적으로 다루어지지 않으며, 이는 생물막 거동에 대한 최적의 보고 및 센터 간 비교에 중요합니다. 따라서 이 프로토콜의 목표는 COMSTAT를 통해 다양한 항균 조건에서 체외 생물막 구조를 정량화하기 위한 간단하고 재현 가능한 프레임워크를 제공하는 것입니다. 이 기술은 생물막 복제 형태로 성장한 CF P. aeruginosa 분리물을 사용하여 모델링하고 tobramycin 및 항-Psl 단클론 항체인 Psl0096에 노출시킵니다. 단계별 접근 방식은 사용자의 모호성을 줄이고 중요한 이미지 처리 단계를 간과할 가능성을 최소화하는 것을 목표로 합니다. 특히, 이 프로토콜은 이미지 분할 및 적절한 정량 분석 기능 선택을 포함하여 COMSTAT의 수동 작동과 관련된 주관적 변동의 제거를 강조합니다. 이 방법을 사용하려면 사용자가 COMSTAT를 실행하기 전에 컨포칼 이미지를 처리하는 데 추가 시간을 소비해야 하지만, 자동화된 출력에서 잘못 표현된 생물막 이질성을 최소화하는 데 도움이 됩니다.
서문
생물막은 자체 생산된 세포외 고분자 물질(EPS)의 매트릭스를 배향하는 미생물의 집합체입니다. EPS 매트릭스는 주로 박테리아 세포, 물, 단백질, 다당류, 지질 및 핵산1으로 구성된 매우 복잡하며, 이 모든 것은 생물막을 자유 생활 플랑크톤 세포와 뚜렷하게 다르게 만듭니다. 생물막 EPS는 서로 접착되고 다양한 표면에 부착됩니다. EPS 매트릭스는 세포 간 신호 전달 및 방어에 사용되는 대사 산물, 유전 물질 및 화합물의 세포 간 교환을 매개하는 특성을 가지고 있습니다2. 이러한 특성은 집합적으로 생물막의 구조적 무결성과 외부 스트레스 요인에 대한 보호를 제공하여 면역 회피 및 항생제 내성에 기여합니다3.
녹농균(Pseudomonas aeruginosa)은 잘 알려진 병원내 병원체로, 항균제에 대한 반응으로 회피성 생물막 성장 전략을 채택하는 것으로 알려져 있습니다. 이에 대한 대표적인 예가 열성 유전 질환인 낭포성 섬유증(CF)이 있는 환자에서 발생합니다. 생물막은 항생제 내성 녹농균(P. aeruginosa)4의 발병에 중추적인 역할을 하며, CF 환자에서 만성 폐 감염을 일으켜 폐 기능 저하를 가속화하고 조기 사망을 초래한다5. 따라서 CF 6,7 환자로부터 얻은 녹농균 분리제에 대한 항생제 및 새로운 항감염제의 효능을 테스트하기 위해 시험관 내 생물막 연구를 수행합니다. 생물막 형성 후 항균제를 구조물 외부에 적용하고 공초점 레이저 스캐닝 현미경(CLSM)을 사용하여 생물막 세그먼트의 고해상도 3차원 재구성을 생성합니다. 그런 다음 컴퓨터 소프트웨어인 COMSTAT를 ImageJ의 플러그인으로 사용하여 생물막 아키텍처 8,9,10,11의 변화를 정량화하는 것이 일반적입니다.
COMSTAT는 생물막 구조를 정량화하는 데 유용하지만 이미지 분석의 재현성 및 표준화는 일반적으로 다루어지지 않습니다. 예를 들어, COMSTAT를 실행하기 전에 수행된 이미지 처리 절차는 객관적이지만 이미지 임계값(12,13)을 설정할 때 주관성 요소를 포함합니다. 유사한 방식으로 COMSTAT 프로그램을 사용하면 작업자가 이미지 분할을 위한 기본 및 고급 조건 및 매개변수를 선택할 수 있을 뿐만 아니라 10가지 정량 분석 기능(예: 두께 분포, 표면적, 바이오매스, 무차원 거칠기 계수)을 선택할 수 있습니다. 다양한 사용자 옵션과 다양한 작업자 전문 지식 수준이 결합되어 생물막 거동에 대한 잘못된 보고가 발생할 수 있습니다.
따라서 이 프로토콜의 목표는 COMSTAT를 사용하여 체외 생물막 구조의 정량적 비교를 위한 비교적 간단한 방법을 제시하는 것입니다. 여기에서, CF P. aeruginosa 분리체의 생물막 세그먼트의 3차원 이미지는 재현 가능한 시험관 내 생물막 실험을 수행하는 데 사용되는 확립된 기술인 챔버형 커버글라스 모델14를 사용하여 CLSM을 통해 캡처됩니다. COMSTAT를 ImageJ의 플러그인으로 활용하는 이 방법을 통해 연구원은 다양한 조건에서 항균제가 있는 경우 생물막 구조의 변화를 정량적으로 식별할 수 있습니다. 전반적으로 이 방법은 COMSTAT의 수동 작동과 관련된 주관적인 변동을 제거하여 센터 간 프로토콜의 표준화를 용이하게 하는 것을 목표로 합니다.
프로토콜
1. 세균 분리 수집
- SickKids(토론토)에서 흡입 토브라마이신으로 박멸 치료를 받고 있는 CF가 있는 소아 환자 코호트에서 분리한 P. 녹농균을 얻습니다. -80 °C에서 글리세롤 시트레이트 및 하위 배양물에서 사용 전에 최소 3회 동결합니다.
2. 생체외 생물막 형성
참고: 변형을 가한 체외 생물막 형성을 위해 챔버형 커버글라스 방법1을 사용하십시오. 이 모델의 전체 워크플로는 그림 1에 나와 있습니다.
- 트립틱 대두 한천과 5% 양의 혈액으로 제조된 혈액 한천에서 37°C에서 밤새 분리한 P. aeruginosa 를 성장시킵니다( 재료 표 참조).
- 혈액 한천에서 1-2개의 박테리아 콜로니를 4mL의 용모성 육수(LB)에 접종합니다. 225 rpm으로 설정된 쉐이커에서 37 °C에서 밤새 성장시킵니다.
- 4mL의 신선한 LB에 40μL의 배양액을 추가하여 하룻밤 접종물의 1:100 희석액을 준비합니다. 225rpm으로 설정된 shaker에서 37°C에서 3-4시간 동안 성장시켜 600nm(OD600)에서 약 0.1(초기 로그 단계)의 광학 밀도를 달성합니다.
- 접종물 220 μL를 8 챔버 커버 글라스 슬라이드의 각 웰로 옮깁니다. 37 °C에서 24 시간 동안 방해받지 않고 배양합니다.
- 생물막이 바닥에서 분리되는 것을 방지하기 위해 각 웰에서 매체를 천천히 제거합니다.
알림: 슬라이드를 45° 각도로 앞으로 기울이고 피펫 팁이 있는 베이스를 건드리지 않고 각 챔버 웰의 하단 모서리에서 매체를 흡입합니다. - 100μL의 56μg/mL 형광 라벨링 단클론 항체(mAb)( 재료 표 참조)를 준비하고 지정된 챔버 웰 측면에 천천히 추가합니다. 박테리아 항원 에피토프에 항체가 부착될 수 있도록 실온(RT)에서 1시간 동안 배양합니다.
참고: 사용하기 전에 형광 라벨링된(적색) mAb, Psl0096을 LB로 희석하여 최종 농도 56μg/mL를 얻으십시오. Psl0096은 항-Psl mAb(Cam003의 최적화된 친화성 유도체)로, 초기 세포 부착 및 구조적 무결성에 관여하는 녹농균 생물막의 핵심 EPS 매트릭스 구성 요소인 클래스 I Psl 에피토프에 결합합니다15. - 100μg/mL 항생제 용액 1000μL( 재료 표 참조)를 준비하고 지정된 챔버 웰 측면에 천천히 추가합니다. 37 °C에서 24 시간 동안 방해받지 않고 배양합니다.
참고: 사용하기 전에 LB에 토브라마이신 항생제 50mg/mL 스톡을 희석하여 최종 농도 1000μg/mL를 얻습니다.
3. 생물막 형광 염색
- 0.01mM 용액의 살아있는 세포 염색 형광 염료를 준비합니다. 챔버가 있는 웰에서 매체를 천천히 제거하고 각 웰에 200μL의 염료 혼합물을 추가합니다. 어두운 곳에서 45분 동안 RT에서 배양합니다.
참고: 사용하기 전에 5mM 스톡 4μL를 2mL의 LB에 추가하여 생세포 염색(녹색) 형광 염료를 준비합니다. - 각 웰에서 배지를 천천히 제거하고 200μL의 새 LB로 2회 세척합니다.
- 각 웰에 200μL의 새 LB를 추가하고 컨포칼 현미경을 통해 검사를 진행합니다.
4. 컨포칼 현미경을 통한 이미지 획득
참고: 이미지 처리 및 COMSTAT 분석 절차는 그림 2에 나와 있습니다. 생물막 염색 당일에 웰의 이미지를 획득합니다. 시각화 지연이 1시간을 초과하는 경우 추가 처리할 때까지 챔버 커버글라스를 어두운 곳에서 냉장 보관하십시오.
- 컨포칼 현미경 시스템( 재료 표 참조)을 사용하여 적절한 레이저 여기 파장과 획득을 위한 필터 세트를 사용하여 웰의 이미지를 획득합니다.
참고: 여기에서는 각각 561 및 491 nm excitation 파장을 사용하여 형광 표지(빨간색) mAb 및 live cell(녹색) 염색을 여기시킵니다. - 20–25x Water Immersion Lens를 사용하여 0.3 μm 단위로 층이 쌓인 z-stack 이미지(하층에서 각 생물막 세그먼트의 상단까지)를 캡처합니다. 웰당 최소 6개의 이미지 스택을 가져옵니다.
참고: 여기서는 25x 수침 렌즈가 장착된 고해상도 카메라를 사용하여 이미지를 시각화하고 이미지 분석 소프트웨어를 사용하여 처리합니다( 재료 표 참조). 소프트웨어 설정 및 디지털 이미징 파라미터(즉, 밝기 및 감도)를 단일 실험의 모든 획득에 대해 일정하게 유지합니다. - COMSTAT 분석을 위해 이미지를 OME-TIFF로 저장합니다.
참고: OME-TIFF가 각 채널(예: 빨간색 및 녹색)에 대해 별도로 저장되어야 합니다. 이 단계는 사용하는 이미지 분석 소프트웨어에 따라 다릅니다. - 2.1–4.3단계를 반복하여 분리 박테리아당 총 3개의 생물학적 복제(즉, 3개의 독립적인 실험)에서 이미지를 캡처합니다.
5. COMSTAT 분석
참고: ImageJ에 플러그인(Comstat2)으로 다시 작성된 무료 컴퓨터 프로그램인 COMSTAT 16,17을 사용하여 이미지를 정량적으로 분석합니다. 다운로드한 패키지 내에 있는 생물막의 이미지 스택을 분석하기 위한 일반 지침을 읽어보십시오. 이 기여는 생물막 형성에 대한 항균제의 효과를 정량화하기 위해 권장되는 선택된 ImageJ 처리 단계 및 COMSTAT 기능과 함께 요약된 프로토콜을 제공합니다.
- http://www.comstat.dk/ 에서 Comstat2 패키지를 다운로드합니다. 설치된 폴더 내에서 ImageJ를 찾아 실행합니다.
- 바탕 화면에 원본 폴더를 만들고 폴더에 단일 OME-TIFF를 추가합니다.
- 소스 폴더에서 OME-TIFF를 열고 바이오매스가 없는 빈 레이어를 삭제합니다. 이러한 계층은 z 스택의 처음 또는 마지막 몇 개 계층이 됩니다.
참고: 현미경으로 정의한 z-stack 경계는 때때로 사용자에 의해 과대평가됩니다. 이러한 빈 도면층을 삭제하면 COMSTAT 해석을 위한 보다 정교한 z 스택 경계가 설정됩니다. - File(파일) | 가져오기 | 이미지 시퀀스. 원본 폴더를 찾아 열지 않고 강조 표시한 다음 Select(선택)를 클릭합니다. '시퀀스 옵션' 창이 나타납니다. 확인을 선택합니다.
참고: ImageJ에서 추가 이미지를 가져오려면 먼저 소스 폴더에서 이전 OME-TIFF를 제거한 다음 새 OME-TIFF를 폴더에 추가하고 5.3단계와 5.4단계를 반복합니다. - 이미지를 선택하여 생물막의 방향을 뒤집습니다 . 변환 | Z를 대칭 이동하여 지층을 첫 번째(맨 위) 스택으로 배치합니다.
참고: COMSTAT 알고리즘은 위(스택 1)에서 맨 아래로 z 방향으로 생물막을 읽습니다. 사용하는 컨포칼 현미경 시스템에 따라 OME-TIFF 출력이 반전될 수 있습니다. 따라서 출력 데이터에 결함이 생기는 것을 방지하기 위해 기판을 이미지 스택 1로 배치하여 슬라이스의 순서를 반대로 하는 것이 중요합니다. - 이미지 속성을 정의하여 Image | 속성. '소스' 창이 나타납니다.
- 'Unit of length'를 'micron'으로 지정합니다.
- 다음 방정식을 사용하여 'Pixel width'와 'Pixel height'를 수학적으로 결정합니다.
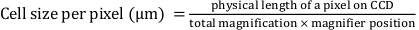 (1)
(1)
여기서 '픽셀 너비'와 '픽셀 높이'는 0.427로 정의되며, 여기서 CCD(charge-coupled device)에서 픽셀의 물리적 길이는 16μm입니다. 총 배율은 25x입니다. 돋보기 위치는 1.5x입니다.
참고: 픽셀당 셀 크기를 계산하는 데 사용되는 방정식은 현미경 카메라 제조업체에 따라 다를 수 있습니다. 또는 'Pixel width'와 'Pixel height'는 공간 보정으로 정의할 수 있습니다(https://imagej.net/Spatial_Calibration 참조). - 'Voxel depth'를 0.3(즉, 각 z-stack 레이어 사이의 증분 공간)으로 정의합니다. '소스' 창에서 확인을 선택합니다.
- 이미지 | 조정 | 임계값. '임계값' 창이 나타납니다.
참고: 이미지의 개체는 회색조 배경에 빨간색으로 표시됩니다. 또는 '임계값' 창의 드롭다운 메뉴를 사용하여 'Red'를 'B&W'로 변경하여 임계값을 흑백으로 조정할 수 있습니다.- 이미지 창에서 슬라이더를 맨 오른쪽(즉, 생물막의 맨 위 층)으로 조정합니다. 배경 잡음을 제거하려면 이미지의 히스토그램을 표시하는 'Threshold' 창을 사용하여 최대 및 최소 임계값을 수동으로 설정합니다. 먼저 하단 슬라이더를 가능한 한 오른쪽으로 조정하여 최대 임계값을 설정합니다. 둘째, 상단 슬라이더를 사용하여 최소 임계값을 조정하면 이미지가 적색 바이오매스와 회색조 배경의 두 단계로 분할됩니다(그림 3).
참고: 본 문서에 설명된 체외 생물막 형성 및 형광 현미경 검사 절차는 이상적인 경우에 OME-TIFF를 생성하여 간단한 히스토그램 임계값 방법을 통해 이미지를 두 개의 별개 단계로 분할할 수 있습니다. 그러나 경우에 따라 서로 다른 위상 간의 히스토그램 구분이 명확하지 않습니다. 이는 광범위한 배경 소음, 다양한 배경 강도 또는 바이오매스와 배경 사이의 낮은 강도 대비의 존재 때문일 수 있습니다. 이러한 경우에, 사용자는 향상된 세분화 절차(18,19)를 채택해야 한다.- 또는 '임계값' 창에서 '기본값'으로 설정된 왼쪽 드롭다운 메뉴를 사용하여 개별 이미지에 대한 임계값을 알고리즘적으로 조정합니다. 이 기능은 선택할 수 있는 17가지 알고리즘 임계값 옵션을 제공합니다(https://imagej.net/Auto_Threshold 참조). 가장 적용 가능한 옵션을 선택한 다음 '자동'을 선택하여 임계값을 설정합니다.
- 임계값을 조정하면 이미지 창의 슬라이더를 사용하여 각 레이어를 스크롤하여 배경 노이즈가 전체적으로 충분히 제거되었는지 확인합니다.
- '임계값' 창에서 설정을 선택하여 먼저 더 낮은 임계값을 수정합니다. '임계값 수준 설정' 창이 나타납니다. 확인을 선택합니다. 설정을 다시 선택하고 이 단계를 반복하여 최대 임계값을 수정합니다.
알림: '설정'을 선택할 때마다 하단 슬라이더가 자동으로 조정될 수 있습니다. 이러한 경우 슬라이더를 수동으로 다시 조정하고(또는 알고리즘 임계값 중 하나를 사용하는 경우 '자동' 선택) 5.6.2–5.6.3단계를 반복합니다. 주요 아이디어는 슬라이더가 자동으로 다시 조정될 때마다 '설정'을 나중에 두 번 더 선택하여 상한 임계값과 하한 임계값이 모두 고정되도록 해야 한다는 것입니다. - 적용을 선택하면 '스택을 바이너리로 변환' 창이 나타납니다. 확인을 선택한 다음 '임계값' 창을 종료합니다.
- Plugins | 바이오 형식 | 바이오 형식 내보내기|. 새 파일 이름을 입력하고 원본 폴더에 OME-TIFF로 저장합니다. 'Bio-Formats Exporter – Multiple Files' 창이 나타납니다. 확인을 선택합니다. 'Bio-Formats Exporter Options' 창이 나타납니다. 확인을 선택합니다.
참고: COMSTAT 분석을 진행하기 전에 새로 조정된 흑백 OME-TIFF만 소스 폴더에 저장되어 있는지 확인하십시오. 폴더에서 원본 OME-TIFF를 모두 제거합니다.
- 이미지 창에서 슬라이더를 맨 오른쪽(즉, 생물막의 맨 위 층)으로 조정합니다. 배경 잡음을 제거하려면 이미지의 히스토그램을 표시하는 'Threshold' 창을 사용하여 최대 및 최소 임계값을 수동으로 설정합니다. 먼저 하단 슬라이더를 가능한 한 오른쪽으로 조정하여 최대 임계값을 설정합니다. 둘째, 상단 슬라이더를 사용하여 최소 임계값을 조정하면 이미지가 적색 바이오매스와 회색조 배경의 두 단계로 분할됩니다(그림 3).
- 플러그인(Plugins) | 컴스타트2 |. '정보' 창이 나타납니다. 확인을 선택합니다. 세 개의 창이 나타납니다.
- 'Observed Directories' 창(오른쪽 상단)에서 Add(추가)를 선택합니다. 원본 폴더를 찾아 열지 않고 강조 표시한 다음 선택을 선택합니다. COMSTAT를 통해 분석할 OME-TIFF를 나열하는 'Images in Directories' 창(왼쪽 상단)이 나타납니다.
- 'Comstat 2.1' 창(오른쪽 하단)에서 '자동 임계값 설정(Otsu's method)'을 선택 취소하여 소프트웨어가 개별 OME-TIFF에 대해 이전에 설정한 임계값을 사용하도록 합니다. 또한 CVF(Connected Volume Filtering)를 선택 취소하여 생물막의 매우 얇은 부분과 생물막 구조의 공극 내에서 발견되는 자유 부유 세포 또는 바이오매스가 분석에 포함되도록 합니다.
참고: COMSTAT 분석은 매우 초기 생물막(24시간 초기 성장)과 항균 처리 후 남은 플랑크톤 세포/콜로니에 대해 수행되므로 여기에서 CVF를 선택 취소하십시오. 성숙한 생물막의 경우 CVF를 선택하여 생물막 구조에 연결된 생물량만 정량화되도록 합니다. - 'Comstat 2.1' 창(오른쪽 하단)에서 정량 분석을 위해 원하는 기능을 선택합니다. 여기에서 Bio Mass, Thickness Distribution 및 Surface Area를 선택합니다. 이동 을 선택하여 프로그램을 실행합니다. '로그' 창(왼쪽 하단)에서 '선택한 기능/이미지 사용 완료!'가 나타날 때까지 출력 데이터가 처리 중으로 표시됩니다. COMSTAT 측정값을 기록합니다. 또한 이러한 측정값은 소스 폴더에 TXT 파일로 자동 저장됩니다.
결과
CF에 감염된 환자로부터 배양한 녹농균 분리물은 체외 생물막 구조에서 항균제에 의한 변화를 정확하게 정량화하는 데 있어 이 접근법의 강점을 입증하는 데 사용됩니다. 이 모델의 전체 워크플로는 그림 1에 나와 있습니다. ImageJ의 이미지 처리 및 COMSTAT 분석 절차는 그림 2에 나와 있습니다. CLSM z-stack 이미지(OME-TIFF로 저장됨)에 적용된 Imag...
토론
in vitro biofilm 구조의 3차원 이미지를 정량적으로 비교하기 위한 규정된 방법은 없으며, 이러한 맥락에서 설명된 절차는 작업자 간 변동성(inter-operator variability)으로 인해 표준화하기 어려운 경우가 많다(20). 따라서 이 프로토콜은 다양한 항균 조건에서 체외 생물막 구조의 변화를 정량화하려는 COMSTAT 응용 분야에 간단하고 재현 가능한 프레임워크를 제공합니다. 이 ?...
공개
없음
감사의 말
저자들은 이 연구에 자금을 제공한 낭포성 섬유증 재단(Cystic Fibrosis Foundation)에 감사를 표하고자 합니다.
자료
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
참고문헌
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유