Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Количественная оценка влияния противомикробных препаратов на архитектуру биопленки in vitro с помощью программного обеспечения COMSTAT
В этой статье
Резюме
Антимикробные изменения архитектуры биопленки Pseudomonas aeruginosa различаются среди клинических изолятов, культивируемых у пациентов с муковисцидозом и хронической легочной инфекцией. После конфокальной микроскопии программное обеспечение COMSTAT может быть использовано для количественной оценки вариаций в архитектуре биопленки (например, площади поверхности, толщины, биомассы) для отдельных изолятов с целью оценки эффективности противоинфекционных агентов.
Аннотация
Биопленки представляют собой агрегаты микроорганизмов, которые полагаются на самостоятельно производимую матрицу внеклеточного полимерного вещества для защиты и структурной целостности. Известно, что внутрибольничный патоген, Pseudomonas aeruginosa, принимает биопленочный режим роста, вызывая хроническую легочную инфекцию у пациентов с муковисцидозом (МВ). Компьютерная программа COMSTAT является полезным инструментом для количественной оценки антимикробных изменений в архитектуре биопленки P. aeruginosa путем извлечения данных из трехмерных конфокальных изображений. Тем не менее, стандартизированная работа программного обеспечения рассматривается реже, что важно для оптимальной отчетности о поведении биопленки и кросс-центрового сравнения. Таким образом, целью данного протокола является обеспечение простой и воспроизводимой основы для количественного определения биопленочных структур in vitro в различных антимикробных условиях с помощью COMSTAT. Метод смоделирован с использованием изолята CF P. aeruginosa , выращенного в виде репликатов биопленки и подвергнутого воздействию тобрамицина и моноклонального антитела против Psl Psl0096. Пошаговый подход направлен на уменьшение неопределенности для пользователя и минимизацию вероятности пропуска важных этапов обработки изображений. В частности, в протоколе подчеркивается устранение субъективных вариаций, связанных с ручной работой COMSTAT, включая сегментацию изображений и выбор соответствующих функций количественного анализа. Несмотря на то, что этот метод требует от пользователей дополнительных затрат времени на обработку конфокальных изображений перед запуском COMSTAT, он помогает свести к минимуму искаженную гетерогенность биопленки при автоматизированных выводах.
Введение
Биопленки представляют собой агрегаты микроорганизмов, ориентированные в матрице самостоятельно производимых внеклеточных полимерных веществ (ЭПС). Матрица ЭПС очень сложна и состоит в основном из бактериальных клеток, воды, белков, полисахаридов, липидов и нуклеиновых кислот1, которые делают биопленки заметно отличающимися от свободно живущих планктонных клеток. Биопленочные ЭПС прилипают друг к другу и к различным поверхностям. Матрица EPS обладает свойствами, которые опосредуют межклеточный обмен метаболитов, генетического материала и соединений, используемых для межклеточной передачи сигналов и защиты2. Эти свойства в совокупности обеспечивают структурную целостность биопленки и защиту от внешних стрессоров, способствуя уклонению от иммунитета и устойчивости к противомикробным препаратам3.
Pseudomonas aeruginosa является хорошо известным внутрибольничным патогеном, который, как известно, использует стратегию уклончивого роста биопленки в ответ на противомикробные препараты. Ярким примером этого являются пациенты с рецессивным генетическим заболеванием, муковисцидозом (МВ). Биопленки играют ключевую роль в развитии устойчивой к противомикробным препаратам P. aeruginosa4 и позволяют установить хроническую легочную инфекцию у пациентов с муковисцидозом, вызывающую ускоренное снижение функции легких и преждевременную смертность5. В связи с этим проводятся исследования биопленки in vitro для проверки эффективности антибиотиков и новых противоинфекционных средств против изолятов P. aeruginosa, полученных от пациентов с CF 6,7. После формирования биопленки противомикробные препараты наносятся наружно на структуру, а конфокальная лазерная сканирующая микроскопия (CLSM) используется для создания трехмерных реконструкций сегментов биопленки с высоким разрешением. Обычной практикой является использование компьютерного программного обеспечения COMSTAT в качестве плагина к ImageJ для количественной оценки изменений в архитектуре биопленки 8,9,10,11.
Несмотря на то, что COMSTAT полезен для количественной оценки структуры биопленки, воспроизводимость и стандартизация анализа изображений рассматриваются реже. Например, процедура обработки изображений, выполняемая до запуска COMSTAT, является объективной, но содержит элемент субъективности при установке пороговых значений изображения12,13. Аналогичным образом, программа COMSTAT позволяет оператору выбирать от базовых до расширенных условий и параметров для сегментации изображения, а также десять функций количественного анализа (например, распределение толщины, площадь поверхности, биомасса, коэффициент безразмерной шероховатости). Множество пользовательских опций в сочетании с различным уровнем квалификации оператора может привести к ошибочному представлению информации о поведении биопленки.
Таким образом, целью данного протокола является представление относительно простого метода количественного сравнения биопленочных структур in vitro с использованием COMSTAT. В данной работе трехмерные изображения сегментов биопленки изолята CF P. aeruginosa захватываются с помощью CLSM с использованием модели покровного стекла в камере14 — признанной методики, используемой для проведения воспроизводимых экспериментов с биопленкой in vitro . Используя COMSTAT в качестве плагина к ImageJ, этот метод позволяет исследователям количественно идентифицировать изменения в архитектуре биопленки в присутствии противомикробных препаратов в различных условиях. В целом, этот метод направлен на устранение субъективных вариаций, связанных с ручным управлением COMSTAT, тем самым способствуя стандартизации протоколов в разных центрах.
протокол
1. Сбор бактериальных изолятов
- Получите изоляты P. aeruginosa из когорты педиатрических пациентов с муковисцидозом, проходящих эрадикационное лечение ингаляционным тобрамицином в клинике SickKids (Торонто). Замораживайте изоляты при температуре -80 °C в цитрате глицерина и субкультуре не менее трех раз перед использованием.
2. Образование биопленки in vitro
ПРИМЕЧАНИЕ: Используйте камерный методпокровного стекла 1 для формирования биопленки in vitro с модификациями. Общий рабочий процесс этой модели показан на рисунке 1.
- Изолят P. aeruginosa выращивают в течение ночи при 37 °C на кровяном агаре, приготовленном из триптического соевого агара и 5% овечьей крови (см. Таблицу материалов).
- Инокулируйте 1–2 колонии бактерий из кровяного агара в 4 мл лизогенного бульона (ЛБ). Выращивайте в течение ночи при температуре 37 °C на шейкере, настроенном на 225 об/мин.
- Приготовьте ночное разведение инокулюма в соотношении 1:100, добавив 40 мкл культуры в 4 мл свежего LB. Выращивайте в течение 3–4 ч при 37 °C в шейкере, настроенном на 225 об/мин, чтобы достичь оптической плотности примерно 0,1 при 600 нм (наружный диаметр600) (ранняя фаза логарифма).
- Перекачайте по 220 мкл инокулюма в каждую лунку 8-камерного покровного стекла. Спокойно инкубировать при температуре 37 °C в течение 24 часов.
- Медленно удаляйте среду из каждой лунки, чтобы предотвратить отслоение биопленок у основания.
ПРИМЕЧАНИЕ: Наклоните предметное стекло вперед под углом 45° и отсасывайте среду из нижних углов каждой лунки с камерой, не касаясь основания наконечником пипетки. - Приготовьте и медленно добавьте 100 мкл флуоресцентного меченого моноклонального антитела (mAb) с концентрацией 56 мкг/мл (см. Таблицу материалов) на боковую сторону обозначенных камерных лунок. Инкубировать при комнатной температуре (ОТ) в течение 1 ч, чтобы обеспечить присоединение антител к эпитопу бактериального антигена.
ПРИМЕЧАНИЕ: Перед применением разбавьте флуоресцентный меченый (красный) mAb, Psl0096, в LB до получения конечной концентрации 56 мкг/мл. Psl0096 представляет собой анти-Psl mAb (оптимизированное аффинное производное Cam003), которое связывается с эпитопом Psl класса I — ключевым компонентом матрицы EPS биопленок P. aeruginosa, участвующим в первоначальном прикреплении клеток и структурной целостности15. - Приготовьте и медленно добавьте 100 мкл раствора антибиотика в концентрации 1000 мкг/мл (см. Таблицу материалов) в боковые части предназначенных для этого камерных лунок. Спокойно инкубировать при температуре 37 °C в течение 24 часов.
ПРИМЕЧАНИЕ: Перед применением разведите запас антибиотика тобрамицина в дозе 50 мг/мл в LB до получения конечной концентрации 1000 мкг/мл.
3. Биопленочное флуоресцентное окрашивание
- Приготовьте 0,01 мМ раствор флуоресцентного красителя, окрашивающего живые клетки. Медленно удалите среду из камерных лунок и добавьте по 200 мкл смеси красителей в каждую лунку. Инкубировать в RT в темноте в течение 45 минут.
ПРИМЕЧАНИЕ: Перед использованием приготовьте флуоресцентный краситель для окрашивания живых клеток (зеленый), добавив 4 мкл 5 мМ материала к 2 мл LB. - Медленно удалите среду из каждой лунки и промойте 2 раза 200 мкл свежего LB.
- Добавьте по 200 мкл свежего LB в каждую лунку и приступайте к исследованию с помощью конфокальной микроскопии.
4. Получение изображений с помощью конфокальной микроскопии
ПРИМЕЧАНИЕ: Процедура обработки изображений и анализа COMSTAT представлена на рисунке 2. Получите снимки лунок в тот же день окрашивания биопленкой. Если задержка в визуализации превышает 1 ч, камерное покровное стекло поставьте в холодильник в темное время суток до дальнейшей обработки.
- Получение изображений лунок с помощью системы конфокального микроскопа (см. Таблицу материалов) с соответствующими длинами волн лазерного возбуждения и наборами фильтров для получения изображений.
ПРИМЕЧАНИЕ: Здесь возбуждайте флуоресцентную метку (красный) mAb и окрашивание живых клеток (зеленый), используя длины волн возбуждения 561 и 491 нм соответственно. - Захватывайте многослойные изображения z-стека (от субстрата до вершины каждого сегмента биопленки) с шагом 0,3 мкм с помощью 20–25-кратной линзы для погружения в воду. Возьмите не менее 6 стеков изображений на лунку.
ПРИМЕЧАНИЕ: Здесь визуализируйте изображения с помощью камеры высокого разрешения с 25-кратным водяным иммерсионным объективом и обработанные с помощью программного обеспечения для анализа изображений (см. Таблицу материалов). Поддерживайте постоянную настройку программного обеспечения и параметры цифровой визуализации (т. е. яркость и чувствительность) для всех полученных данных в одном эксперименте. - Сохраняйте изображения в формате OME-TIFF для анализа COMSTAT.
ПРИМЕЧАНИЕ: Убедитесь, что OME-TIFF сохраняются отдельно для каждого канала (т. е. красного и зеленого). Этот шаг зависит от используемого программного обеспечения для анализа изображений. - Повторите шаги 2.1–4.3 для получения изображений в общей сложности 3 биологических репликатов (т. е. 3 независимых экспериментов) для каждого бактериального изолята.
5. Анализ COMSTAT
ПРИМЕЧАНИЕ: Количественно анализируйте изображения с помощью свободно доступной компьютерной программы COMSTAT16,17, переписанной в виде плагина (Comstat2) для ImageJ. Ознакомьтесь с общими инструкциями по анализу стеков изображений биопленок в загруженном пакете. В этом материале представлен обобщенный протокол с выбранными этапами обработки ImageJ и функциями COMSTAT, рекомендованными для количественной оценки влияния противомикробных препаратов на образование биопленки.
- Загрузите пакет Comstat2 с http://www.comstat.dk/. В установленной папке найдите ImageJ и запустите его.
- Создайте исходную папку на рабочем столе и добавьте в нее один OME-TIFF.
- Откройте OME-TIFF из исходной папки и удалите все пустые слои, не содержащие биомассы. Эти слои будут либо первыми, либо последними несколькими слоями z-стека.
ПРИМЕЧАНИЕ: Определяемые микроскопом границы z-стека иногда переоцениваются пользователями. Удаление этих пустых слоев создает более точную границу z-стека для анализа COMSTAT. - Импортируйте OME-TIFF в ImageJ, выбрав Файл | Импорт | Последовательность изображений. Найдите исходную папку, выделите ее, не открывая ее, и нажмите кнопку Выбрать. Появится окно «Параметры последовательности». Нажмите OK.
ПРИМЕЧАНИЕ: Чтобы импортировать дополнительные изображения в ImageJ, сначала удалите предыдущий OME-TIFF из исходной папки, затем добавьте новый OME-TIFF в папку и повторите шаги 5.3 и 5.4. - Измените ориентацию биопленки, выбрав Изображение | Трансформация | Отразите Z, чтобы расположить субстрат как первый (самый верхний) стек.
ПРИМЕЧАНИЕ: Алгоритмы COMSTAT считывают биопленки по оси z сверху (стек 1) вниз. В зависимости от используемой системы конфокального микроскопа выход OME-TIFF может быть инвертирован. Таким образом, важно изменить порядок срезов, расположив субстрат как стек изображений 1, чтобы предотвратить искажение выходных данных. - Определите свойства изображения, выбрав Изображение | Свойства. Появится окно «Источник».
- Укажите «Единицу длины» как «микрон».
- Математически определите «Ширину в пикселях» и «Высоту в пикселях» с помощью следующего уравнения:
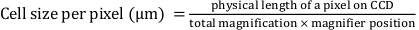 (1)
(1)
Здесь «Ширина в пикселях» и «Высота в пикселях» определяются как 0,427, где физическая длина пикселя на устройстве с зарядовой связью (ПЗС) составляет 16 мкм; общее увеличение составляет 25x; и положение лупы составляет 1,5x.
ПРИМЕЧАНИЕ: Уравнение, используемое для расчета размера ячейки на пиксель, может отличаться в зависимости от производителя камеры микроскопа. Кроме того, «Ширина в пикселях» и «Высота в пикселях» могут быть определены с помощью пространственной калибровки (см. https://imagej.net/Spatial_Calibration). - Определите «Глубину воксела» как 0,3 (т. е. инкрементальное пространство между каждым слоем z-стека). Выберите OK в окне «Источник».
- Отрегулируйте пороговое значение изображения, выбрав Изображение | Настройка | Порог. Появится окно «Порог».
ПРИМЕЧАНИЕ: Объекты на изображении будут отображаться красным цветом на фоне в оттенках серого. Кроме того, порог можно настроить в черно-белом варианте, используя выпадающее меню в окне «Порог», чтобы изменить «Красный» на «Черно-белый».- В окне изображения переместите ползунок вправо (т.е. в самый верхний слой биопленки). Чтобы убрать фоновый шум, используйте окно «Порог», в котором отображается гистограмма изображения, чтобы вручную установить максимальное и минимальное пороговые значения. Сначала установите максимальное пороговое значение, отрегулировав нижний ползунок как можно дальше вправо. Во-вторых, используйте верхний ползунок для настройки минимального порогового значения, которое разделяет изображение на две отдельные фазы: красная биомасса и серый фон (Рисунок 3).
ПРИМЕЧАНИЕ: Описанная здесь процедура формирования биопленки in vitro и флуоресцентной микроскопии генерирует OME-TIFF в идеальном случае, позволяя сегментировать изображения на две различные фазы с помощью простого метода пороговых значений гистограммы. Однако в некоторых случаях различие в гистограмме между различными фазами не так очевидно. Это может быть связано с наличием обширного фонового шума, различной интенсивностью фона или низким контрастом между биомассой и фоном. В таких случаях пользователи должны принять усовершенствованную процедуру сегментации18,19.- В качестве альтернативы можно настроить пороговые значения алгоритмически для отдельных изображений с помощью левого выпадающего меню, установленного как «По умолчанию» в окне «Порог». Эта функция предоставляет на выбор 17 различных вариантов алгоритмических пороговых значений (см. https://imagej.net/Auto_Threshold). Выберите наиболее подходящий вариант, затем нажмите «Авто», чтобы установить порог.
- Когда пороговые значения скорректированы, используйте ползунок в окне изображения для прокрутки каждого слоя, чтобы убедиться, что фоновый шум достаточно удален по всему слою.
- Выберите «Установить » в окне «Порог», чтобы сначала зафиксировать нижнее пороговое значение. Появится окно «Установить пороговые уровни». Нажмите OK. Нажмите кнопку Задать еще раз и повторите этот шаг, чтобы зафиксировать максимальное пороговое значение.
ПРИМЕЧАНИЕ: Каждый раз, когда выбрано «Установить», нижний ползунок может регулироваться автоматически. В таких случаях вручную перенастройте ползунок (или выберите «Авто», если используется один из алгоритмических порогов) и повторите шаги 5.6.2–5.6.3. Основная идея заключается в том, что всякий раз, когда ползунки автоматически перенастраиваются, «Установить» следует выбрать еще два раза, чтобы убедиться, что и верхний, и нижний пороги фиксированы. - Нажмите «Применить », и появится окно «Конвертировать стек в двоичный». Нажмите OK, а затем выйдите из окна «Порог».
- Сохраните только что настроенный OME-TIFF, выбрав «Плагины» | Биоформаты | Экспортер биоформатов|. Введите новое имя файла и сохраните его как OME-TIFF в исходной папке. Появится окно «Экспортер биоформатов – несколько файлов». Нажмите OK. Появится окно «Параметры экспортера биоформатов». Нажмите OK.
ПРИМЕЧАНИЕ: Перед переходом к анализу COMSTAT убедитесь, что в исходной папке сохранены только только новые черно-белые OME-TIFF. Извлеките из папки все оригинальные OME-TIFF.
- В окне изображения переместите ползунок вправо (т.е. в самый верхний слой биопленки). Чтобы убрать фоновый шум, используйте окно «Порог», в котором отображается гистограмма изображения, чтобы вручную установить максимальное и минимальное пороговые значения. Сначала установите максимальное пороговое значение, отрегулировав нижний ползунок как можно дальше вправо. Во-вторых, используйте верхний ползунок для настройки минимального порогового значения, которое разделяет изображение на две отдельные фазы: красная биомасса и серый фон (Рисунок 3).
- Запустите COMSTAT, выбрав Плагины | Comstat2 |. Появится окно «О нас». Нажмите OK. Появятся три окна.
- В окне "Наблюдаемые каталоги" (вверху справа) выберите Добавить. Найдите исходную папку, выделите ее, не открывая ее, и нажмите кнопку Выбрать. Появится окно «Изображения в каталогах» (вверху слева), в котором перечислены OME-TIFF, которые будут проанализированы с помощью COMSTAT.
- В окне «Comstat 2.1» (внизу справа) снимите флажок «Автоматическое пороговое значение (метод Отсу)», чтобы убедиться, что программное обеспечение использует пороговые значения, ранее установленные для отдельных OME-TIFF. Кроме того, снимите флажок Connected Volume Filtering (CVF), чтобы убедиться, что в анализ включены очень тонкие части биопленки, а также свободно плавающие клетки или биомасса, обнаруженные в пустотах структуры биопленки.
ПРИМЕЧАНИЕ: Здесь следует отменить выбор CVF, поскольку анализ COMSTAT проводится на очень ранних биопленках (начальный рост через 24 часа) и оставшихся планктонных клетках/колониях после антимикробной обработки. Для зрелых биопленок выберите CVF, чтобы обеспечить количественное определение только биомассы, связанной со структурой биопленки. - В окне «Comstat 2.1» (справа внизу) выберите нужные признаки для количественного анализа. Здесь выберите «Биомасса», «Распределение толщины» и «Площадь поверхности». Нажмите кнопку Перейти , чтобы запустить программу. В окне «Журнал» (слева внизу) отображается обработка выходных данных до тех пор, пока не появится сообщение «Завершено с выбранными функциями/изображениями!». Запишите измерения COMSTAT. Эти измерения также автоматически сохраняются в виде файлов TXT в исходной папке.
Результаты
Изолят P. aeruginosa, культивируемый у инфицированного пациента с муковисцидозом, используется для демонстрации преимуществ этого подхода в точном количественном определении антимикробных индуцированных изменений в архитектуре биопленки in vitro . Общий рабочий процесс этой модели...
Обсуждение
Не существует предписанного метода количественного сравнения трехмерных изображений биопленочных структур in vitro , и процедуры, описанные в этом контексте, часто трудно стандартизировать из-за межоператорной вариабельности. Таким образом, этот протокол предлагает пр...
Раскрытие информации
Никакой
Благодарности
Авторы выражают признательность Фонду муковисцидоза за предоставление финансирования для этого исследования.
Материалы
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
Ссылки
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены