需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
使用 COMSTAT 软件量化抗菌剂对 体外 生物膜结构的影响
摘要
抗菌诱导的 铜绿假单胞 菌生物膜结构变化在从囊性纤维化和慢性肺部感染患者培养的临床分离株中有所不同。在共聚焦显微镜之后,COMSTAT软件可用于量化单个分离株的生物膜结构(例如,表面积、厚度、生物量)的变化,以评估抗感染剂的功效。
摘要
生物膜是微生物的聚集体,它们依赖于自产生的细胞外聚合物物质基质来保护和结构完整性。已知医院病原体 铜绿假单胞 菌采用生物膜生长模式,导致囊性纤维化 (CF) 患者发生慢性肺部感染。计算机程序COMSTAT是一种有用的工具,用于通过从三维共聚焦图像中提取数据来量化 铜绿假单胞菌 生物膜结构中抗菌诱导的变化。然而,软件的标准化操作较少得到解决,这对于生物膜行为的最佳报告和跨中心比较非常重要。因此,该协议的目的是提供一个简单且可重复的框架,用于通过COMSTAT量化不同抗菌条件下的 体外 生物膜结构。该技术使用铜 绿假单胞菌 分离物建模,以生物膜重复的形式生长,并暴露于妥布霉素和抗 Psl 单克隆抗体 Psl0096。循序渐进的方法旨在减少用户的歧义,并最大限度地减少忽视关键图像处理步骤的机会。具体而言,该协议强调消除与COMSTAT手动操作相关的主观变化,包括图像分割和选择适当的定量分析功能。尽管这种方法要求用户在运行COMSTAT之前花费额外的时间处理共聚焦图像,但它有助于最大限度地减少自动输出中错位的生物膜异质性。
引言
生物膜是微生物的聚集体,定向在自产生的细胞外聚合物物质 (EPS) 基质中。EPS 基质非常复杂,主要由细菌细胞、水、蛋白质、多糖、脂质和核酸1 组成,所有这些都使生物膜与自由生活的浮游细胞截然不同。生物膜EPS相互粘附在各种表面上。EPS 基质具有介导代谢物、遗传物质和用于细胞间信号转导和防御的化合物的细胞间交换的特性2。这些特性共同提供了生物膜结构完整性和抵御外部压力源的保护,有助于免疫逃避和抗菌素耐药性3.
铜绿假单胞菌是一种公认的医院病原体,已知它对抗菌药物采取逃避生物膜生长策略。一个典型的例子发生在患有隐性遗传疾病囊性纤维化 (CF) 的患者身上。生物膜在抗菌耐药性铜绿假单胞菌4 的发展中起着关键作用,并允许 CF 患者建立慢性肺部感染,导致肺功能加速下降和过早死亡5。因此,进行体外生物膜研究以测试抗生素和新型抗感染剂对从 CF 6,7 患者获得的铜绿假单胞菌分离株的疗效。生物膜形成后,将抗菌剂应用于结构外部,并使用共聚焦激光扫描显微镜 (CLSM) 生成生物膜段的高分辨率三维重建。通常的做法是使用计算机软件COMSTAT作为ImageJ的插件,以量化生物膜结构的变化8,9,10,11。
尽管COMSTAT可用于量化生物膜结构,但图像分析的可重复性和标准化问题较少得到解决。例如,在运行COMSTAT之前执行的图像处理程序是客观的,但在设置图像阈值12,13时包含主观性因素。以类似的方式,COMSTAT程序允许操作员选择从基本到高级的图像分割条件和参数,以及十种定量分析功能(例如,厚度分布、表面积、生物量、无量纲粗糙度系数)。众多的用户选项,再加上不同的操作人员专业知识水平,可能会导致对生物膜行为的误导性报告。
因此,该协议的目标是提出一种相对简单的方法,用于使用COMSTAT对体外生物膜结构进行定量比较。本文中,使用带腔盖玻片模型14 通过 CLSM 捕获来自铜绿假单胞菌分离物的生物膜片段的三维图像——这是一种用于执行可重复体外生物膜实验的成熟技术。利用 COMSTAT 作为 ImageJ 的插件,该方法使研究人员能够在不同条件下定量识别存在抗菌剂的情况下生物膜结构的变化。总体而言,该方法旨在消除与COMSTAT手动操作相关的主观差异,从而促进各中心协议的标准化。
研究方案
1. 细菌分离物收集
- 从 SickKids(多伦多)接受吸入妥布霉素根除治疗的 CF 儿科患者队列中获取 铜绿假单胞菌 分离株。在-80°C的柠檬酸甘油中冷冻分离物,并在使用前进行至少三次传代培养。
2. 体外 生物膜的形成
注:使用腔室盖玻片方法1 进行体外生物膜形成并进行修饰。该模型的整体工作流程如图 1 所示。
- 在用胰蛋白酶大豆琼脂和5%绵羊血制备的血琼脂上培养 铜绿假单胞菌 分离物过夜(参见 材料表)。
- 将血琼脂中的 1-2 个细菌菌落接种到 4 mL 溶原肉汤 (LB) 中。在设置为225rpm的振荡器上在37°C下生长过夜。
- 通过在 4 mL 新鲜 LB 中加入 40 μL 培养物来制备 1:100 稀释的过夜接种物。 在 37 °C 下在设置为 225 rpm 的振荡器上生长 3-4 小时,以在 600 nm (OD600) 处达到约 0.1 的光密度(早期对数期)。
- 将 220 μL 接种物转移到 8 室盖玻片的每个孔中。在37°C下不受干扰地孵育24小时。
- 从每个孔中缓慢取出培养基,以防止生物膜在基底脱落。
注意: 将载玻片向前倾斜 45° 角,并从每个腔室孔的底角吸出培养基,而不用移液器吸头接触底座。 - 制备 100 μL 56 μg/mL 荧光标记单克隆抗体 (mAb)(参见 材料表)并缓慢加入到指定腔室孔的一侧。在室温 (RT) 下孵育 1 小时,以使抗体附着在细菌抗原表位上。
注:使用前,将荧光标记的(红色)mAb Psl0096 稀释在 LB 中,以获得 56 μg/mL 的终浓度。Psl0096 是一种抗 Psl mAb(Cam003 的优化亲和衍生物),它与 I 类 Psl 表位结合,Psl 表位是 铜绿假单胞菌 生物膜的关键 EPS 基质成分,参与初始细胞附着和结构完整性15。 - 准备并缓慢加入 100 μL 的 1000 μg/mL 抗生素溶液(参见 材料表)到指定腔室孔的一侧。在37°C下不受干扰地孵育24小时。
注:使用前,在 LB 中稀释 50 mg/mL 妥布霉素抗生素储备液,以获得 1000 μg/mL 的终浓度。
3. 生物膜荧光染色
- 制备 0.01 mM 活细胞染色荧光染料溶液。慢慢地从腔室孔中取出培养基,并向每个孔中加入 200 μL 染料混合物。在室温下在黑暗中孵育 45 分钟。
注:使用前,通过将 4 μL 的 5 mM 原液加入 2 mL LB 中来制备活细胞染色(绿色)荧光染料。 - 从每个孔中缓慢取出培养基,并用 200 μL 新鲜 LB 洗涤 2 次。
- 向每个孔中加入 200 μL 新鲜 LB,并通过共聚焦显微镜进行检查。
4. 通过共聚焦显微镜进行图像采集
注:图像处理和 COMSTAT 分析程序如图 2 所示。在生物膜染色的同一天采集孔的图像。如果可视化延迟超过 1 小时,则将带腔室的盖玻片在黑暗中冷藏,直到进一步处理。
- 使用具有适当激光激发波长和滤光片组的共聚焦显微镜系统(参见 材料表)获取孔的图像。
注:此处,分别使用 561 nm 和 491 nm 激发波长激发荧光标记(红色)mAb 和活细胞(绿色)染料。 - 使用 20-25 倍水浸镜头以 0.3 μm 的增量捕获分层 z 堆栈图像(从基质到每个生物膜段的顶部)。每孔至少采集 6 个图像堆栈。
注意:在这里,使用带有 25 倍水浸镜头的高分辨率相机可视化图像,并使用图像分析软件进行处理(参见 材料表)。在单次实验中保持所有采集的软件设置和数字成像参数(即亮度和灵敏度)不变。 - 将图像另存为 OME-TIFF 以进行 COMSTAT 分析。
注意: 确保为每个通道(即红色和绿色)单独保存 OME-TIFF。此步骤因所使用的图像分析软件而异。 - 重复步骤 2.1-4.3,从每个细菌分离株总共 3 个生物学重复(即 3 个独立实验)中捕获图像。
5. COMSTAT分析
注意:使用免费提供的计算机程序 COMSTAT16,17 定量分析图像,该程序被重写为插件 (Comstat2) 到 ImageJ。阅读下载的软件包中分析生物膜图像堆栈的一般说明。本文提供了一个总结的方案,其中推荐了选定的 ImageJ 处理步骤和 COMSTAT 特征,用于量化抗菌剂对生物膜形成的影响。
- 从 http://www.comstat.dk/ 下载 Comstat2 包。在已安装的文件夹中,找到 ImageJ 并运行它。
- 在桌面上创建一个源文件夹,并向该文件夹添加一个 OME-TIFF。
- 从源文件夹中打开 OME-TIFF,并删除任何不包含生物质的空图层。这些层将是 z 堆栈的第一层或最后几层。
注意:显微镜定义的 z 堆栈边界有时会被用户高估。删除这些空层可为 COMSTAT 分析建立更精细的 z 堆栈边界。 - 通过选择 "文件"|" 在 ImageJ 中导入 OME-TIFF进口 |图像序列。找到源文件夹,高亮显示而不打开它,然后单击" 选择"。将出现一个"序列选项"窗口。选择" 确定"。
注意:要在 ImageJ 中导入其他图像,请先从源文件夹中删除之前的 OME-TIFF,然后将新的 OME-TIFF 添加到文件夹中,并重复步骤 5.3 和 5.4。 - 通过选择 "图像"|"来翻转生物膜的方向 |转型 |翻转 Z 形,将基质定位为第一个(最顶部)堆栈。
注意:COMSTAT 算法从顶部(堆栈 1)到底部在 z 方向上读取生物膜。根据所使用的共聚焦显微镜系统,OME-TIFF输出可以反转。因此,通过将基质定位为图像堆栈 1 来颠倒切片的顺序非常重要,以防止输出数据产生缺陷。 - 通过选择" 图像"|"属性。将出现一个"源"窗口。
- 将"长度单位"指定为"微米"。
- 使用以下公式以数学方式确定"像素宽度"和"像素高度":
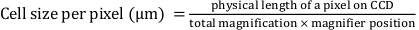 (1)
(1)
这里,"像素宽度"和"像素高度"定义为0.427,其中电荷耦合器件(CCD)上像素的物理长度为16 μm;总放大倍率为25倍;放大镜位置为 1.5 倍。
注意:用于计算每个像素的单元大小的公式可能因显微镜相机制造商而异。或者,"像素宽度"和"像素高度"可以通过空间校准来定义(参见 https://imagej.net/Spatial_Calibration)。 - 将"体素深度"定义为 0.3(即每个 z 堆栈层之间的增量空间)。在"源"窗口中选择 "确定 "。
- 通过选择" 图像"|"调整 |阈值。将出现一个"阈值"窗口。
注意:图像中的对象将显示为红色,背景为灰度。或者,可以使用"阈值"窗口中的下拉菜单将"红色"更改为"黑白",从而以黑白方式调整阈值。- 在图像窗口中,将滑块调整到最右侧(即生物膜的最顶层)。要消除背景噪声,请使用显示图像直方图的"阈值"窗口手动设置最大和最小阈值。首先,通过尽可能向右调整下滑块来设置最大阈值。其次,使用上部滑块调整最小阈值,该阈值将图像分割为两个独立的阶段:红色生物量和灰度背景(图 3)。
注:本文所述的体外生物膜形成和荧光显微镜程序在理想情况下生成 OME-TIFF,允许通过简单的直方图阈值方法将图像分割成两个不同的阶段。然而,在某些情况下,不同阶段之间的直方图区别并不那么清楚。这可能是由于存在广泛的背景噪声、不同的背景强度或生物量和背景之间的低强度对比。在这种情况下,用户应采用增强的分割程序18,19。- 或者,使用"阈值"窗口中设置为"默认"的左侧下拉菜单通过算法调整单个图像的阈值。此功能提供了 17 种不同的算法阈值选项供您选择(请参阅 https://imagej.net/Auto_Threshold)。选择最适用的选项,然后选择"自动"以设置阈值。
- 调整阈值后,使用图像窗口中的滑块滚动浏览每一层,以确保在整个过程中充分消除背景噪声。
- 在"阈值"窗口中选择" 设置 "以首先修复较低的阈值。将出现"设置阈值水平"窗口。选择" 确定"。再次选择" 设置 ",然后重复此步骤以修复最大阈值。
注意: 每次选择"设置"时,下方滑块可能会自动调整。在这种情况下,请手动重新调整滑块(如果使用其中一个算法阈值,请选择"自动")并重复步骤 5.6.2–5.6.3。主要思想是,每当滑块自动重新调整时,应在之后再选择两次"设置",以确保上限和下限阈值都固定。 - 选择" 应用 ",将出现"将堆栈转换为二进制"窗口。选择" 确定",然后退出"阈值"窗口。
- 通过选择 Plugins |生物格式 |生物格式导出器|.输入新文件名并在源文件夹中另存为 OME-TIFF。将出现"生物格式导出器 - 多个文件"窗口。选择" 确定"。将出现一个"生物格式导出器选项"窗口。选择" 确定"。
注意:在继续进行 COMSTAT 分析之前,请确保仅将新调整的黑白 OME-TIFF 保存在源文件夹中。从文件夹中删除所有原始 OME-TIFF。
- 在图像窗口中,将滑块调整到最右侧(即生物膜的最顶层)。要消除背景噪声,请使用显示图像直方图的"阈值"窗口手动设置最大和最小阈值。首先,通过尽可能向右调整下滑块来设置最大阈值。其次,使用上部滑块调整最小阈值,该阈值将图像分割为两个独立的阶段:红色生物量和灰度背景(图 3)。
- 通过选择 Plugins |Comstat2 |.将出现一个"关于"窗口。选择" 确定"。将出现三个窗口。
- 在"观察到的目录"窗口(右上角)中,选择 "添加"。找到源文件夹,高亮显示而不打开它,然后选择"选择"。将出现一个"目录中的图像"窗口(左上角),其中列出了要通过 COMSTAT 分析的 OME-TIFF。
- 在"Comstat 2.1"窗口(右下角)上,取消选择"自动阈值(Otsu 方法)",以确保软件使用之前为单个 OME-TIFF 设置的阈值。此外,取消选择 连接体积过滤器g (CVF),以确保分析中包括生物膜的非常薄的部分以及在生物膜结构空隙中发现的自由漂浮细胞或生物质。
注意:在这里,取消选择CVF,因为COMSTAT分析是在非常早期的生物膜(24小时初始生长)和抗菌处理后剩余的浮游细胞/菌落上进行的。对于成熟的生物膜,选择CVF以确保仅对连接到生物膜结构的生物量进行量化。 - 在"Comstat 2.1"窗口(右下角)上,选择所需的定量分析特征。在这里,选择 生物质量、厚度分布 和 表面积。选择 "转到 "以运行程序。在"日志"窗口(左下角)中,输出数据显示正在处理中,直到出现"完成选定的功能/图像!记录 COMSTAT 测量值。这些测量值也会自动保存为源文件夹中的 TXT 文件。
结果
从感染的CF患者中培养的 铜绿假单胞菌 分离物用于证明该方法在准确量化抗菌诱导 的体外 生物膜结构变化方面的优势。该模型的整体工作流程如图 1 所示。ImageJ 中的图像处理和 COMSTAT 分析过程如图 2 所示。 图 3 显示了 ImageJ 中用于图像分割的简单直方图阈值方法,该方法应用于 CLSM z 堆栈图像(另存为 OME-TIFF)。?...
讨论
没有规定的方法来定量比较 体外 生物膜结构的三维图像,并且由于操作者之间的可变性,在这种情况下描述的程序通常难以标准化20。因此,该协议为寻求量化不同抗菌条件下 体外 生物膜结构变化的COMSTAT应用提供了一个简单且可重复的框架。该技术的优势是使用 CF 铜绿假单胞菌 分离物建模的,该分离物以生物膜重复的形式生长,并暴露于妥布霉素和抗 Psl...
披露声明
没有
致谢
作者感谢囊性纤维化基金会为这项研究提供资金。
材料
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
参考文献
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。