Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Cuantificación de los efectos de los antimicrobianos en la arquitectura de biopelículas in vitro mediante el software COMSTAT
En este artículo
Resumen
Los cambios inducidos por antimicrobianos en la arquitectura de la biopelícula de Pseudomonas aeruginosa difieren entre los aislados clínicos cultivados de pacientes con fibrosis quística e infección pulmonar crónica. Después de la microscopía confocal, el software COMSTAT se puede utilizar para cuantificar las variaciones en la arquitectura de la biopelícula (por ejemplo, área de superficie, espesor, biomasa) para aislados individuales para evaluar la eficacia de los agentes antiinfecciosos.
Resumen
Las biopelículas son agregados de microorganismos que dependen de una matriz autoproducida de sustancia polimérica extracelular para su protección e integridad estructural. Se sabe que el patógeno nosocomial, Pseudomonas aeruginosa, adopta un modo de crecimiento de biopelícula, causando infección pulmonar crónica en pacientes con fibrosis quística (FQ). El programa informático, COMSTAT, es una herramienta útil para cuantificar los cambios inducidos por antimicrobianos en la arquitectura de la biopelícula de P. aeruginosa mediante la extracción de datos de imágenes confocales tridimensionales. Sin embargo, el funcionamiento estandarizado del software se aborda con menos frecuencia, lo cual es importante para la generación óptima de informes sobre el comportamiento de la biopelícula y la comparación entre centros. Por lo tanto, el objetivo de este protocolo es proporcionar un marco simple y reproducible para cuantificar estructuras de biopelículas in vitro en diferentes condiciones antimicrobianas a través de COMSTAT. La técnica se modela utilizando un aislado de CF P. aeruginosa , cultivado en forma de réplicas de biopelícula y expuesto a tobramicina y al anticuerpo monoclonal anti-Psl, Psl0096. El enfoque paso a paso tiene como objetivo reducir la ambigüedad del usuario y minimizar la posibilidad de pasar por alto pasos cruciales del procesamiento de imágenes. Específicamente, el protocolo enfatiza la eliminación de las variaciones subjetivas asociadas con la operación manual de COMSTAT, incluida la segmentación de imágenes y la selección de funciones de análisis cuantitativo apropiadas. Aunque este método requiere que los usuarios dediquen tiempo adicional a procesar imágenes confocales antes de ejecutar COMSTAT, ayuda a minimizar la heterogenicidad de la biopelícula tergiversada en las salidas automatizadas.
Introducción
Los biofilms son agregados de microorganismos orientados en una matriz de sustancias poliméricas extracelulares (EPS) autoproducidas. La matriz de EPS es muy compleja y consta principalmente de células bacterianas, agua, proteínas, polisacáridos, lípidos y ácidos nucleicos1, todos los cuales producen biopelículas claramente diferentes de las células planctónicas de vida libre. Los EP de biofilm se adhieren entre sí y a varias superficies. La matriz de EPS tiene propiedades que median el intercambio de metabolitos de célula a célula, material genético y compuestos utilizados para la señalización y defensa intercelular2. Estas propiedades proporcionan colectivamente a las biopelículas integridad estructural y protección contra los factores estresantes externos, lo que contribuye a la evasión inmunitaria y la resistencia a los antimicrobianos3.
Pseudomonas aeruginosa es un patógeno nosocomial bien reconocido, conocido por adoptar una estrategia evasiva de crecimiento de biopelículas en respuesta a los antimicrobianos. Un buen ejemplo de esto ocurre en pacientes con el trastorno genético recesivo, la fibrosis quística (FQ). Las biopelículas desempeñan un papel fundamental en el desarrollo de P. aeruginosa resistente a los antimicrobianos 4 y permiten el establecimiento de infección pulmonar crónica en pacientes con FQ, lo que provoca un deterioro acelerado de la función pulmonar y una mortalidad prematura5. Por lo tanto, se realizan estudios de biofilm in vitro para probar la eficacia de antibióticos y nuevos agentes antiinfecciosos contra aislados de P. aeruginosa obtenidos de pacientes con FQ 6,7. Después de la formación de la biopelícula, los antimicrobianos se aplican externamente a la estructura y se utiliza la microscopía de escaneo láser confocal (CLSM) para generar reconstrucciones tridimensionales de alta resolución de los segmentos de la biopelícula. Es una práctica común utilizar el software informático, COMSTAT, como complemento de ImageJ, para cuantificar los cambios en la arquitectura de la biopelícula 8,9,10,11.
Aunque COMSTAT es útil para cuantificar la estructura de la biopelícula, la reproducibilidad y la estandarización del análisis de imágenes se abordan con menos frecuencia. Por ejemplo, el procedimiento de procesamiento de imágenes, realizado antes de ejecutar COMSTAT, es objetivo, pero contiene un elemento de subjetividad al establecer umbrales de imagen12,13. De manera similar, el programa COMSTAT permite al operador elegir entre condiciones y parámetros básicos y avanzados para la segmentación de imágenes, así como diez funciones de análisis cuantitativas (por ejemplo, distribución de espesor, área de superficie, biomasa, coeficiente de rugosidad adimensional). La multitud de opciones de usuario, junto con los diferentes niveles de experiencia del operador, puede dar lugar a informes erróneos sobre el comportamiento de la biopelícula.
Por lo tanto, el objetivo de este protocolo es presentar un método relativamente sencillo para la comparación cuantitativa de estructuras de biopelículas in vitro utilizando COMSTAT. En este trabajo, se capturan imágenes tridimensionales de segmentos de biopelícula de un aislado de CF P. aeruginosa a través de CLSM utilizando el modelo14 de cubreobjetos con cámara, una técnica establecida que se utiliza para realizar experimentos de biopelícula in vitro reproducibles. Utilizando COMSTAT como un complemento de ImageJ, este método permite a los investigadores identificar cuantitativamente los cambios en la arquitectura de la biopelícula en presencia de antimicrobianos en condiciones variables. En general, este método tiene como objetivo eliminar las variaciones subjetivas asociadas con la operación manual de COMSTAT, facilitando así la estandarización de los protocolos en todos los centros.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Recolección de aislados bacterianos
- Obtención de aislados de P. aeruginosa de una cohorte de pacientes pediátricos con FQ en tratamiento de erradicación con tobramicina inhalada en SickKids (Toronto). Congele los aislados a -80 °C en citrato de glicerol y subcultivo al menos tres veces antes de su uso.
2. Formación de biofilm in vitro
NOTA: Utilice un método de cubreobjetos con cámara1 para la formación de biopelículas in vitro con modificaciones. El flujo de trabajo general de este modelo se muestra en la figura 1.
- Cultivar P. aeruginosa aislada durante la noche a 37 °C en agar sangre preparado con agar tríptico de soja y 5% de sangre de oveja (ver Tabla de Materiales).
- Inocular 1-2 colonias bacterianas del agar sangre en 4 mL de caldo de lisogenia (LB). Cultive durante la noche a 37 °C en un agitador ajustado a 225 rpm.
- Prepare una dilución 1:100 de inóculo durante la noche añadiendo 40 μL del cultivo en 4 mL de LB fresco. Cultive durante 3-4 h a 37 °C en un agitador ajustado a 225 rpm, para lograr una densidad óptica de aproximadamente 0,1 a 600 nm (OD600) (fase logarítmica temprana).
- Transfiera 220 μL del inóculo a cada pocillo de un portaobjetos de cubreobjetos de 8 cámaras. Incubar sin perturbaciones a 37 °C durante 24 h.
- Retire lentamente el medio de cada pocillo para evitar que las biopelículas se desprendan en la base.
NOTA: Incline la corredera hacia adelante en un ángulo de 45° y aspire el medio desde las esquinas inferiores de cada pocillo con cámara sin tocar la base con la punta de la pipeta. - Prepare y agregue lentamente 100 μL de 56 μg/mL de anticuerpo monoclonal (mAb) marcado con fluorescencia (ver Tabla de Materiales) al lado de los pocillos con cámara designados. Incubar a temperatura ambiente (RT) durante 1 h para permitir la unión del anticuerpo al epítopo del antígeno bacteriano.
NOTA: Antes de usar, diluya el mAb marcado con fluorescente (rojo), Psl0096, en LB para obtener una concentración final de 56 μg/mL. Psl0096 es un mAb anti-Psl (derivado de afinidad optimizada de Cam003), que se une al epítopo Psl de clase I, un componente clave de la matriz de EPS de las biopelículas de P. aeruginosa involucrado en la unión celular inicial y la integridad estructural15. - Prepare y agregue lentamente 100 μL de una solución antibiótica de 1000 μg/mL (consulte la Tabla de Materiales) al costado de los pocillos con cámaras designados. Incubar sin perturbaciones a 37 °C durante 24 h.
NOTA: Antes de usar, diluya un stock de 50 mg/mL de antibiótico tobramicina en LB para obtener una concentración final de 1000 μg/mL.
3. Tinción fluorescente de biopelícula
- Prepare una solución de 0,01 mM de un tinte fluorescente de tinción de células vivas. Retire lentamente el medio de los pocillos con cámara y agregue 200 μL de la mezcla de colorante a cada pocillo. Incubar en RT en la oscuridad durante 45 min.
NOTA: Antes de usar, prepare el tinte fluorescente de tinción de células vivas (verde) agregando 4 μL de un caldo de 5 mM a 2 mL de LB. - Retire lentamente el medio de cada pocillo y lave 2 veces con 200 μL de LB fresco.
- Añadir 200 μL de LB fresco a cada pocillo y proceder al examen mediante microscopía confocal.
4. Adquisición de imágenes por microscopía confocal
NOTA: El procedimiento de procesamiento de imágenes y análisis COMSTAT se presenta en la Figura 2. Adquiera imágenes de los pozos el mismo día de la tinción de la biopelícula. Si el retraso en la visualización supera 1 hora, refrigere el cubreobjetos con cámara en la oscuridad hasta que se procese más.
- Adquiera imágenes de pozos utilizando un sistema de microscopio confocal (consulte la tabla de materiales) con longitudes de onda de excitación láser adecuadas y conjuntos de filtros para la adquisición.
NOTA: Aquí, excite el mAb marcado con fluorescente (rojo) y la tinción de células vivas (verde) utilizando longitudes de onda de excitación de 561 y 491 nm, respectivamente. - Capture imágenes de pila z en capas (desde el sustrato hasta la parte superior de cada segmento de biopelícula) en incrementos de 0,3 μm con una lente de inmersión en agua de 20 a 25x. Tome al menos 6 pilas de imágenes por pozo.
NOTA: Aquí, visualice las imágenes utilizando una cámara de alta resolución con una lente de inmersión en agua de 25x y procesadas con software de análisis de imágenes (consulte la Tabla de materiales). Mantenga constante la configuración del software y los parámetros de imagen digital (es decir, brillo y sensibilidad) para todas las adquisiciones en un solo experimento. - Guarde las imágenes como OME-TIFF para el análisis COMSTAT.
NOTA: Asegúrese de que los OME-TIFF se guarden por separado para cada canal (es decir, rojo y verde). Este paso varía en función del software de análisis de imágenes utilizado. - Repita los pasos 2.1 a 4.3 para capturar imágenes de un total de 3 réplicas biológicas (es decir, 3 experimentos independientes) por aislado bacteriano.
5. Análisis COMSTAT
NOTA: Analice las imágenes cuantitativamente utilizando el programa informático de libre acceso, COMSTAT16,17, reescrito como un complemento (Comstat2) en ImageJ. Lea las instrucciones generales para analizar pilas de imágenes de biopelículas dentro del paquete descargado. Esta contribución proporciona un protocolo resumido, con pasos de procesamiento de ImageJ seleccionados y características de COMSTAT recomendadas para cuantificar los efectos de los antimicrobianos en la formación de biopelículas.
- Descargue el paquete Comstat2 de http://www.comstat.dk/. Dentro de la carpeta instalada, localice ImageJ y ejecútelo.
- Cree una carpeta de origen en el escritorio y agregue un solo OME-TIFF a la carpeta.
- Abra OME-TIFF desde la carpeta de origen y elimine las capas vacías que no contengan biomasa. Estas capas serán la primera o la última capa de la pila z.
NOTA: Los límites de la pila z definidos por el microscopio a veces son sobreestimados por los usuarios. La eliminación de estas capas vacías establece un límite de pila z más refinado para el análisis COMSTAT. - Importar OME-TIFF en ImageJ seleccionando Archivo | Importación | Secuencia de imágenes. Localice la carpeta de origen, resáltela sin abrirla y haga clic en Seleccionar. Aparecerá una ventana de 'Opciones de secuencia'. Seleccione Aceptar.
NOTA: Para importar imágenes adicionales en ImageJ, primero elimine el OME-TIFF anterior de la carpeta de origen, luego agregue el nuevo OME-TIFF a la carpeta y repita los pasos 5.3 y 5.4. - Invierta la orientación de la biopelícula seleccionando Imagen | Transformar | Gire Z para colocar el sustrato como la primera pila (superior).
NOTA: Los algoritmos COMSTAT leen las biopelículas en la dirección z desde la parte superior (pila 1) hasta la parte inferior. Dependiendo del sistema de microscopio confocal utilizado, la salida OME-TIFF se puede invertir. Por lo tanto, es importante invertir el orden de los segmentos colocando el sustrato como pila de imágenes 1 para evitar que los datos de salida se vuelvan defectuosos. - Defina las propiedades de la imagen seleccionando Imagen | Propiedades. Aparecerá una ventana de 'Fuente'.
- Especifique 'Unidad de longitud' como 'micras'.
- Determine matemáticamente 'Ancho de píxel' y 'Altura de píxel' usando la siguiente ecuación:
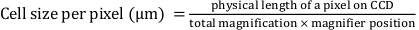 (1)
(1)
En este caso, la «anchura de píxel» y la «altura de píxel» se definen como 0,427, donde la longitud física de un píxel en el dispositivo de carga acoplada (CCD) es de 16 μm; El aumento total es de 25x; y la posición de la lupa es de 1,5x.
NOTA: La ecuación utilizada para calcular el tamaño de celda por píxel puede variar según el fabricante de la cámara microscópica. Alternativamente, 'Ancho de píxel' y 'Altura de píxel' se pueden definir mediante calibración espacial (consulte https://imagej.net/Spatial_Calibration). - Defina la "profundidad de vóxel" como 0,3 (es decir, el espacio incremental entre cada capa de pila z). Seleccione Aceptar en la ventana 'Fuente'.
- Ajuste el umbral de la imagen seleccionando Imagen | Ajustar | Umbral. Aparecerá una ventana de 'Umbral'.
NOTA: Los objetos de la imagen aparecerán en rojo con un fondo en escala de grises. Alternativamente, el umbral se puede ajustar en blanco y negro, utilizando el menú desplegable en la ventana 'Umbral' para cambiar 'Rojo' a 'Blanco y negro'.- En la ventana de la imagen, ajuste el control deslizante hacia el extremo derecho (es decir, la capa superior de la biopelícula). Para eliminar el ruido de fondo, utilice la ventana 'Umbral', que muestra un histograma de la imagen, para establecer manualmente los valores de umbral máximo y mínimo. Primero, establezca el valor máximo del umbral ajustando el control deslizante inferior lo más a la derecha posible. En segundo lugar, utilice el control deslizante superior para ajustar el valor umbral mínimo, que segmenta la imagen en dos fases separadas: biomasa roja y fondo en escala de grises (Figura 3).
NOTA: El procedimiento de formación de biofilm in vitro y microscopía fluorescente descrito en este documento genera OME-TIFF en el caso ideal, lo que permite segmentar las imágenes en dos fases distintas mediante un método de umbral de histograma simple. Sin embargo, en algunos casos, la distinción del histograma entre las diferentes fases no es tan clara. Esto puede deberse a la presencia de un ruido de fondo extenso, intensidades de fondo variables o un contraste de baja intensidad entre la biomasa y el fondo. En tales casos, los usuarios deben adoptar un procedimiento de segmentación mejorado18,19.- Alternativamente, ajuste los umbrales algorítmicamente para imágenes individuales usando el menú desplegable de la izquierda establecido como 'Predeterminado' en la ventana 'Umbral'. Esta función proporciona 17 opciones de umbral algorítmico diferentes para elegir (consulte https://imagej.net/Auto_Threshold). Seleccione la opción más aplicable, luego 'Auto' para establecer el umbral.
- Cuando se ajustan los valores de umbral, utilice el control deslizante de la ventana de la imagen para desplazarse por cada capa y asegurarse de que el ruido de fondo se elimine lo suficiente.
- Seleccione Establecer en la ventana "Umbral" para corregir primero el valor del umbral inferior. Aparecerá la ventana 'Establecer niveles de umbral'. Seleccione Aceptar. Seleccione Establecer de nuevo y repita este paso para fijar el valor de umbral máximo.
NOTA: Cada vez que se selecciona 'Establecer', el control deslizante inferior puede ajustarse automáticamente. En tales casos, vuelva a ajustar manualmente el control deslizante (o seleccione "Automático" si usa uno de los umbrales algorítmicos) y repita los pasos 5.6.2 a 5.6.3. La idea principal es que cada vez que los controles deslizantes se reajusten automáticamente, 'Establecer' debe seleccionarse dos veces más después para garantizar que tanto el umbral superior como el inferior sean fijos. - Seleccione Aplicar y aparecerá la ventana 'Convertir pila a binario'. Seleccione Aceptar y, a continuación, salga de la ventana "Umbral".
- Guarde el OME-TIFF recién ajustado seleccionando Plugins | Bio-Formatos | Exportador de Bio-Formatos|. Introduzca un nuevo nombre de archivo y guárdelo como OME-TIFF en la carpeta de origen. Aparecerá una ventana 'Exportador de bioformatos - Múltiples archivos'. Seleccione Aceptar. Aparecerá una ventana de 'Opciones de exportador de bioformatos'. Seleccione Aceptar.
NOTA: Asegúrese de que solo los OME-TIFF en blanco y negro recién ajustados se guarden en la carpeta de origen antes de continuar con el análisis COMSTAT. Elimine todos los OME-TIFF originales de la carpeta.
- En la ventana de la imagen, ajuste el control deslizante hacia el extremo derecho (es decir, la capa superior de la biopelícula). Para eliminar el ruido de fondo, utilice la ventana 'Umbral', que muestra un histograma de la imagen, para establecer manualmente los valores de umbral máximo y mínimo. Primero, establezca el valor máximo del umbral ajustando el control deslizante inferior lo más a la derecha posible. En segundo lugar, utilice el control deslizante superior para ajustar el valor umbral mínimo, que segmenta la imagen en dos fases separadas: biomasa roja y fondo en escala de grises (Figura 3).
- Ejecute COMSTAT seleccionando Plugins | Comstat2 |. Aparecerá una ventana 'Acerca de'. Seleccione Aceptar. Aparecerán tres ventanas.
- En la ventana "Directorios observados" (arriba a la derecha), seleccione Agregar. Localice la carpeta de origen, resalte sin abrirla y seleccione Elegir. Aparecerá una ventana de 'Imágenes en directorios' (arriba a la izquierda) que enumera los OME-TIFF que se analizarán a través de COMSTAT.
- En la ventana 'Comstat 2.1' (abajo a la derecha), anule la selección de 'Umbral automático (método de Otsu)' para asegurarse de que el software utiliza los valores de umbral previamente configurados para OME-TIFF individuales. Además, anule la selección del filtro de volumen conectadog (CVF) para asegurarse de que se incluyan en el análisis partes muy delgadas de la biopelícula, así como las células flotantes o la biomasa que se encuentran dentro de los vacíos de la estructura de la biopelícula.
NOTA: En este caso, anule la selección de CVF porque el análisis COMSTAT se realiza en biopelículas muy tempranas (crecimiento inicial de 24 horas) y en células/colonias planctónicas restantes después del tratamiento antimicrobiano. En el caso de las biopelículas maduras, seleccione CVF para asegurarse de que solo se cuantifique la biomasa conectada a la estructura de la biopelícula. - En la ventana 'Comstat 2.1' (abajo a la derecha), seleccione las características deseadas para el análisis cuantitativo. Aquí, seleccione Biomasa, Distribución de espesor y Área de superficie. Seleccione Ir para ejecutar el programa. En la ventana 'Registro' (abajo a la izquierda), se muestran los datos de salida procesando hasta que aparece '¡Terminado con las funciones/imágenes seleccionadas!'. Registre las mediciones de COMSTAT. Estas mediciones también se guardan automáticamente como archivos TXT en la carpeta de origen.
Access restricted. Please log in or start a trial to view this content.
Resultados
Se utiliza un aislado de P. aeruginosa cultivado a partir de un paciente infectado con FQ para demostrar las fortalezas de este enfoque en la cuantificación precisa de los cambios inducidos por los antimicrobianos en la arquitectura de la biopelícula in vitro . El flujo de trabajo general de este modelo se representa en la Figura 1. En la Figura 2 se muestra el procedimiento de procesamiento de imágenes y análisis COMSTAT en ImageJ. En la <...
Access restricted. Please log in or start a trial to view this content.
Discusión
No existe un método prescrito para comparar cuantitativamente imágenes tridimensionales de estructuras de biopelículas in vitro , y los procedimientos descritos en este contexto son a menudo difíciles de estandarizar debido a la variabilidad entre operadores20. Por lo tanto, este protocolo ofrece un marco simple y reproducible para las aplicaciones de COMSTAT que buscan cuantificar los cambios en la arquitectura de la biopelícula in vitro bajo diversas condiciones antimicrob...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Ninguno
Agradecimientos
Los autores desean agradecer a la Fundación de Fibrosis Quística por proporcionar fondos para esta investigación.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
Referencias
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. Ø, Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819(2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , Kongens Lyngby. Denmark. (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260(2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677(2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados