A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
כימות ההשפעות של אנטי-מיקרוביאלים על ארכיטקטורת ביופילם במבחנה באמצעות תוכנת COMSTAT
In This Article
Summary
שינויים אנטי-מיקרוביאליים בארכיטקטורת הביופילם Pseudomonas aeruginosa שונים בין מבודדים קליניים שגודלו בתרבית מחולים עם סיסטיק פיברוזיס וזיהום ריאתי כרוני. לאחר מיקרוסקופיה קונפוקלית, ניתן להשתמש בתוכנת COMSTAT כדי לכמת וריאציות בארכיטקטורת ביופילם (למשל, שטח פנים, עובי, ביומסה) עבור מבודדים בודדים כדי להעריך את היעילות של חומרים אנטי זיהומיים.
Abstract
ביופילמים הם אגרגטים של מיקרואורגניזמים המסתמכים על מטריצה בייצור עצמי של חומר פולימרי חוץ-תאי להגנה ולשלמות מבנית. הפתוגן הנוזוקומיאלי, Pseudomonas aeruginosa, ידוע כמאמץ מצב גידול ביופילם, הגורם לזיהום ריאתי כרוני בחולים עם סיסטיק פיברוזיס (CF). תוכנת המחשב, COMSTAT, היא כלי שימושי לכימות שינויים אנטי-מיקרוביאליים בארכיטקטורת הביופילם P . aeruginosa על ידי חילוץ נתונים מתמונות קונפוקליות תלת-ממדיות. עם זאת, פעולה סטנדרטית של התוכנה פחות נפוצה, וזה חשוב לדיווח אופטימלי של התנהגות ביופילם והשוואה בין מרכזים. לפיכך, מטרת פרוטוקול זה היא לספק מסגרת פשוטה וניתנת לשחזור לכימות מבני ביופילם במבחנה בתנאים אנטי-מיקרוביאליים משתנים באמצעות COMSTAT. הטכניקה מעוצבת באמצעות CF P. aeruginosa מבודד, גדל בצורה של שכפול ביופילם, ונחשף לטוברמיצין ולנוגדן החד-שבטי נגד Psl, Psl0096. הגישה שלב אחר שלב שואפת להפחית את העמימות של המשתמשים ולמזער את הסיכוי להתעלם משלבים קריטיים של עיבוד תמונה. באופן ספציפי, הפרוטוקול מדגיש את חיסול וריאציות סובייקטיביות הקשורות לפעולה הידנית של COMSTAT, כולל פילוח תמונה ובחירת פונקציות ניתוח כמותיות מתאימות. למרות ששיטה זו דורשת מהמשתמשים להשקיע זמן נוסף בעיבוד תמונות קונפוקליות לפני הפעלת COMSTAT, היא מסייעת למזער הטרוגניות של ביופילם במצג שווא בפלטים אוטומטיים.
Introduction
ביופילמים הם צברים של מיקרואורגניזמים המכוונים במטריצה של חומרים פולימריים חוץ-תאיים המיוצרים בייצור עצמי (EPS). מטריצת EPS מורכבת מאוד, ומורכבת בעיקר מתאי חיידקים, מים, חלבונים, רב-סוכרים, ליפידים וחומצות גרעין1, שכולם יוצרים ביופילמים שונים באופן מובהק מתאים פלנקטוניים חופשיים. ביופילם EPS דבקים זה בזה ומשטחים שונים. למטריצת EPS תכונות המתווכות חילופי מטבוליטים, חומרים גנטיים ותרכובות המשמשות לאיתות והגנה בין-תאיים2. תכונות אלה יחד מספקות לביופילמים שלמות מבנית והגנה מפני גורמי עקה חיצוניים, ותורמות להתחמקות חיסונית ולעמידות מיקרוביאלית3.
Pseudomonas aeruginosa הוא פתוגן נוסוקומיאלי מוכר היטב, הידוע כמאמץ אסטרטגיית גידול ביופילם מתחמקת בתגובה לאנטי-מיקרוביאליים. דוגמה מצוינת לכך מתרחשת בחולים עם הפרעה גנטית רצסיבית, סיסטיק פיברוזיס (CF). ביופילמים ממלאים תפקיד מרכזי בהתפתחות P. aeruginosa4 עמיד לחיידקים ומאפשרים התבססות של זיהום ריאתי כרוני בחולים עם CF, הגורם לירידה מואצת בתפקוד הריאות ולתמותה מוקדמת5. לפיכך, מחקרי ביופילם במבחנה מבוצעים כדי לבחון את היעילות של אנטיביוטיקה וחומרים אנטי זיהומיים חדשים נגד מבודדי P. aeruginosa המתקבלים מחולים עם CF 6,7. לאחר היווצרות ביופילם, אנטי-מיקרוביאלים מיושמים חיצונית על המבנה, ומיקרוסקופ סריקת לייזר קונפוקלי (CLSM) משמש ליצירת שחזורים תלת-ממדיים ברזולוציה גבוהה של מקטעי ביופילם. לאחר מכן מקובל להשתמש בתוכנת המחשב, COMSTAT, כתוסף ל-ImageJ, כדי לכמת שינויים בארכיטקטורת ביופילם 8,9,10,11.
למרות ש- COMSTAT שימושי לכימות מבנה הביופילם, השכפול והסטנדרטיזציה של ניתוח תמונות פחות נפוצים. לדוגמה, הליך עיבוד התמונה, המבוצע לפני הפעלת COMSTAT, הוא אובייקטיבי, אך מכיל אלמנט של סובייקטיביות בעת הגדרת ספי תמונה12,13. באופן דומה, תוכנית COMSTAT מאפשרת למפעיל לבחור בין תנאים ופרמטרים בסיסיים למתקדמים עבור פילוח תמונה וכן עשר פונקציות ניתוח כמותיות (למשל, התפלגות עובי, שטח פנים, ביומסה, מקדם חספוס חסר ממד). ריבוי אפשרויות המשתמש, בשילוב עם רמות מומחיות שונות של המפעילים, עלול לגרום לדיווח מוטעה על התנהגות ביופילם.
לפיכך, מטרת פרוטוקול זה היא להציג שיטה פשוטה יחסית להשוואה כמותית של מבני ביופילם במבחנה באמצעות COMSTAT. כאן, תמונות תלת-ממדיות של מקטעי ביופילם מבודד CF P. aeruginosa מצולמות באמצעות CLSM באמצעות דגם זכוכית כיסוי תא14 – טכניקה מבוססת המשמשת לביצוע ניסויי ביופילם הניתנים לשחזור במבחנה . באמצעות COMSTAT כתוסף ל-ImageJ, שיטה זו מאפשרת לחוקרים לזהות באופן כמותי שינויים בארכיטקטורת הביופילם בנוכחות אנטי-מיקרוביאלים בתנאים משתנים. בסך הכל, שיטה זו שואפת לבטל וריאציות סובייקטיביות הקשורות לפעולה הידנית של COMSTAT, ובכך להקל על סטנדרטיזציה של פרוטוקולים בין מרכזים.
Protocol
1. איסוף חיידקים מבודדים
- קבל P. aeruginosa מבודד מקבוצה של חולים ילדים עם CF שעברו טיפול מיגור עם tobramycin בשאיפה ב SickKids (טורונטו). יש להקפיא מבודד בטמפרטורה של -80°C בגליצרול ציטראט ובתת-תרבית לפחות שלוש פעמים לפני השימוש.
2. היווצרות ביופילם במבחנה
הערה: השתמש בשיטת זכוכית כיסוי תא1 ליצירת ביופילם במבחנה עם שינויים. זרימת העבודה הכוללת של מודל זה מוצגת באיור 1.
- לגדל P. aeruginosa לבודד לילה ב 37 ° C על אגר דם מוכן עם אגר סויה טריפטי 5% דם כבשים (ראה טבלה של חומרים).
- חסן 1-2 מושבות חיידקים מאגר הדם לתוך 4 מ"ל של מרק ליזוגני (LB). יש לגדול לילה ב-37°C בשייקר המכוון ל-225 סל"ד.
- הכינו דילול של 1:100 של חיסון לילה על ידי הוספת 40 מיקרוליטר של התרבית ב-4 מ"ל של ליברות טריות. גדלו במשך 3-4 שעות ב-37°C על שייקר המכוון ל-225 סל"ד, כדי להשיג צפיפות אופטית של כ-0.1 ב-600 ננומטר (OD600) (שלב הרישום המוקדם).
- מעבירים 220 μL של החיסון לכל באר של מגלשת זכוכית כיסוי בעלת 8 תאים. לדגור ללא הפרעה ב 37 ° C במשך 24 שעות.
- הסר באיטיות את המדיום מכל באר כדי למנוע מהביופילמים להתנתק בבסיס.
הערה: הטה את ההחלקה קדימה בזווית של 45° ושאפו את התווך מהפינות התחתונות של כל תא היטב מבלי לגעת בבסיס עם קצה פיפטה. - הכינו והוסיפו באיטיות 100 μL של נוגדן חד-שבטי (mAb) פלואורסצנטי (ראו טבלת חומרים) לצד הבארות המיועדות לתאים. יש לדגור בטמפרטורת החדר (RT) למשך שעה אחת כדי לאפשר הצמדת נוגדנים לאפיטופ האנטיגן החיידקי.
הערה: לפני השימוש, יש לדלל את ה-mAb הפלואורסצנטי (אדום), Psl0096, ב-LB כדי לקבל ריכוז סופי של 56 מיקרוגרם/מ"ל. Psl0096 הוא anti-Psl mAb (נגזרת זיקה ממוטבת של Cam003), אשר נקשר לאפיטופ Psl Class I - רכיב מטריצת EPS מרכזי של ביופילמים P . aeruginosa המעורבים בהתקשרות ראשונית של תאים ושלמות מבנית15. - הכינו והוסיפו באיטיות 100 מיקרוליטר של תמיסה אנטיביוטית של 1000 מק"ג/מ"ל (ראו טבלת חומרים) לצד הבארות הייעודיות. לדגור ללא הפרעה ב 37 ° C במשך 24 שעות.
הערה: לפני השימוש, יש לדלל מלאי של 50 מ"ג/מ"ל של אנטיביוטיקה מסוג טוברמיצין ב-LB כדי להגיע לריכוז סופי של 1000 מיקרוגרם/מ"ל.
3. צביעה פלואורסצנטית ביופילם
- הכינו תמיסה של 0.01 מילימטר של צבע פלואורסצנטי מכתים תאים חיים. מוציאים באיטיות את המדיום מהבארות הקאמריות ומוסיפים 200 מיקרוליטר של תערובת הצבע לכל באר. דגרו ב-RT בחושך במשך 45 דקות.
הערה: לפני השימוש, הכינו את הצבע הפלואורסצנטי החי לצביעת תאים (ירוק) על ידי הוספת 4 μL של ציר 5 mM ל-2 מ"ל של LB. - הסר באיטיות את המדיום מכל באר, ושטוף 2x עם 200 μL של LB טרי.
- הוסף 200 μL של LB טרי לכל באר ולהמשיך לבדיקה באמצעות מיקרוסקופ קונפוקלי.
4. רכישת תמונה במיקרוסקופ קונפוקלי
הערה: הליך עיבוד התמונה וניתוח COMSTAT מוצג באיור 2. לרכוש תמונות של בארות באותו יום של צביעת ביופילם. אם העיכוב בהדמיה עולה על שעה אחת, יש לקרר את הזכוכית בחושך עד לעיבוד נוסף.
- קבל תמונות של בארות באמצעות מערכת מיקרוסקופ קונפוקלי (ראה טבלת חומרים) עם אורכי גל עירור לייזר מתאימים וערכות סינון לרכישה.
הערה: כאן, עורר את הכתם הפלואורסצנטי המסומן (אדום) mAb ואת כתם התא החי (ירוק) באמצעות אורכי גל עירור של 561 ו- 491 ננומטר, בהתאמה. - צלם תמונות בשכבות מסוג z stack (מתת-השכבה ועד לחלק העליון של כל מקטע ביופילם) בדרגות של 0.3 מיקרומטר באמצעות עדשת טבילה במים של 20-25x. צלם לפחות 6 ערימות תמונות לכל באר.
הערה: כאן, הצג את התמונות באופן חזותי באמצעות מצלמה ברזולוציה גבוהה עם עדשת טבילה במים של 25x ועובד באמצעות תוכנה לניתוח תמונות (ראה טבלת חומרים). שמור על הגדרות תוכנה ופרמטרי הדמיה דיגיטלית (כלומר, בהירות ורגישות) קבועים עבור כל הרכישות בניסוי יחיד. - שמרו תמונות כקובצי OME-TIFF לצורך ניתוח COMSTAT.
הערה: ודא שקובצי OME-TIFF נשמרים בנפרד עבור כל ערוץ (כלומר, אדום וירוק). שלב זה משתנה בהתאם לתוכנת ניתוח התמונה שבה נעשה שימוש. - חזור על שלבים 2.1-4.3 כדי לצלם תמונות מ-3 עותקים ביולוגיים משוכפלים (כלומר, 3 ניסויים בלתי תלויים) לכל מבודד חיידקי.
5. ניתוח COMSTAT
הערה: נתח תמונות באופן כמותי באמצעות תוכנת המחשב הזמינה באופן חופשי, COMSTAT16,17, ששוכתבה כתוסף (Comstat2) ל- ImageJ. קרא את ההוראות הכלליות לניתוח ערימות תמונות של ביופילמים בתוך החבילה שהורדת. תרומה זו מספקת פרוטוקול מסוכם, עם שלבי עיבוד נבחרים של ImageJ ותכונות COMSTAT מומלצות לכימות ההשפעות של אנטי-מיקרוביאלים על היווצרות ביופילם.
- הורד את חבילת Comstat2 מ- http://www.comstat.dk/. בתוך התיקיה המותקנת, אתר את ImageJ והפעל אותו.
- צור תיקיית מקור בשולחן העבודה והוסף OME-TIFF יחיד לתיקיה.
- פתח את OME-TIFF מתיקיית המקור ומחק שכבות ריקות שאינן מכילות ביומסה. שכבות אלה יהיו השכבות הראשונות או האחרונות של ערימת z.
הערה: גבולות z-stack המוגדרים על-ידי מיקרוסקופ מוערכים לעתים יתר על המידה על-ידי משתמשים. מחיקת שכבות ריקות אלה קובעת גבול z מחסנית מעודן יותר לניתוח COMSTAT. - ייבא OME-TIFF ב- ImageJ על-ידי בחירה באפשרות קובץ | ייבוא | רצף תמונות. אתר את תיקיית המקור, סמן מבלי לפתוח אותה ולחץ על בחר. יופיע חלון 'אפשרויות רצף'. בחר אישור.
הערה: כדי לייבא תמונות נוספות ב-ImageJ, הסר תחילה את ה-OME-TIFF הקודם מתיקיית המקור, לאחר מכן הוסף את ה-OME-TIFF החדש לתיקייה וחזור על שלבים 5.3 ו-5.4. - הפוך את כיוון הביופילם על-ידי בחירה באפשרות תמונה | טרנספורמציה | הפוך Z כדי למקם את תת-השכבה כערימה הראשונה (העליונה).
הערה: אלגוריתמים של COMSTAT קוראים ביופילמים בכיוון z מלמעלה (מחסנית 1) למטה. בהתאם למערכת המיקרוסקופ הקונפוקלי בה נעשה שימוש, פלט OME-TIFF יכול להיות הפוך. לכן, חשוב להפוך את סדר הפרוסות על ידי מיקום תת-השכבה כאוסף תמונות 1 כדי למנוע פגם בנתוני הפלט. - הגדר מאפייני תמונה על-ידי בחירה באפשרות תמונה | מאפיינים. יופיע חלון 'מקור'.
- ציין 'יחידת אורך' כ'מיקרון'.
- קבע מתמטית 'רוחב פיקסל' ו'גובה פיקסל' באמצעות המשוואה הבאה:
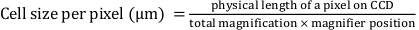 (1)
(1)
כאן, 'רוחב פיקסל' ו'גובה פיקסל' מוגדרים כ-0.427, כאשר האורך הפיזי של פיקסל במכשיר מצמיד הטעינה (CCD) הוא 16 מיקרומטר; ההגדלה הכוללת היא פי 25; ומיקום 'זכוכית מגדלת' הוא 1.5x.
הערה: המשוואה המשמשת לחישוב גודל תא לפיקסל עשויה להשתנות בהתאם ליצרן מצלמת המיקרוסקופ. לחלופין, ניתן להגדיר 'רוחב פיקסל' ו'גובה פיקסל' באמצעות כיול מרחבי (עיין https://imagej.net/Spatial_Calibration). - הגדירו 'עומק ווקסל' כ-0.3 (כלומר, רווח מצטבר בין כל שכבת ערימת z). בחר אישור בחלון 'מקור'.
- התאם את סף התמונה על-ידי בחירה באפשרות תמונה | התאמה | סף. יופיע חלון 'סף'.
הערה: אובייקטים בתמונה יופיעו באדום עם רקע בגווני אפור. לחלופין, ניתן לכוונן את הסף בשחור-לבן, על ידי שימוש בתפריט הנפתח בחלון 'סף' כדי לשנות את 'אדום' ל- 'B&W'.- בחלון התמונה, התאימו את המחוון לקצה הימני (כלומר, השכבה העליונה ביותר של הביופילם). כדי להסיר רעשי רקע, השתמש בחלון 'סף', המציג היסטוגרמה של התמונה, כדי להגדיר ידנית את ערכי הסף המרביים והמינימליים. תחילה, הגדר את ערך הסף המרבי על-ידי התאמת המחוון התחתון ימינה ככל האפשר. שנית, השתמשו במחוון העליון כדי להתאים את ערך הסף המינימלי, אשר מחלק את התמונה לשני שלבים נפרדים: ביומסה אדומה ורקע בגווני אפור (איור 3).
הערה: הליך היווצרות ביופילם במבחנה ומיקרוסקופ פלואורסצנטי המתואר כאן מייצר OME-TIFFs במקרה האידיאלי, ומאפשר לפצל תמונות לשני שלבים נפרדים באמצעות שיטת סף היסטוגרמה פשוטה. עם זאת, במקרים מסוימים, ההבחנה בהיסטוגרמה בין השלבים השונים אינה כה ברורה. זה יכול להיות בגלל נוכחות של רעשי רקע נרחבים, עוצמות רקע משתנות, או ניגודיות בעוצמה נמוכה בין ביומסה לרקע. במקרים כאלה, על המשתמשים לאמץ הליך פילוח משופר18,19.- לחלופין, התאימו את ערכי הסף באופן אלגוריתמי לתמונות בודדות באמצעות התפריט הנפתח השמאלי שהוגדר כ'ברירת מחדל' בחלון 'סף'. תכונה זו מספקת 17 אפשרויות סף אלגוריתמיות שונות לבחירה (עיין בסעיף https://imagej.net/Auto_Threshold). בחר באפשרות המתאימה ביותר, ולאחר מכן 'אוטומטי' כדי להגדיר סף.
- בשעת התאמת ערכי הסף, השתמשו במחוון בחלון התמונה כדי לגלול בכל אחת מהשכבות כדי לוודא שרעשי הרקע יוסרו במידה מספקת לכל אורכה.
- בחר הגדר בחלון 'סף' כדי לתקן תחילה את ערך הסף התחתון. יופיע חלון 'הגדר רמות סף'. בחר אישור. בחר הגדר שוב וחזור על שלב זה כדי לתקן את ערך הסף המרבי.
הערה: בכל פעם שנבחר 'הגדר', המחוון התחתון עשוי להשתנות באופן אוטומטי. במקרים כאלה, כוונן מחדש את המחוון באופן ידני (או בחר 'אוטומטי' אם אתה משתמש באחד מערכי הסף האלגוריתמיים) וחזור על שלבים 5.6.2-5.6.3. הרעיון המרכזי הוא שבכל פעם שהמחוונים מותאמים מחדש באופן אוטומטי, יש לבחור 'סט' פעמיים נוספות לאחר מכן כדי להבטיח שגם הסף העליון וגם התחתון קבועים. - בחר החל ויופיע חלון 'המר ערימה לבינארי'. בחר אישור ולאחר מכן צא מהחלון 'סף'.
- שמור את ה- OME-TIFF החדש שהותאם על-ידי בחירת תוספים | פורמטים ביולוגיים | ביו-פורמטים יצואן|. הזן שם קובץ חדש ושמור כ- OME-TIFF בתיקיית המקור. יופיע חלון 'Bio-Formats Exporter – Multiple Files'. בחר אישור. יופיע חלון 'אפשרויות יצואן של פורמטים ביולוגיים'. בחר אישור.
הערה: ודא שרק רכיבי OME-TIFF בשחור-לבן שהותאמו לאחרונה נשמרים בתיקיית המקור לפני שתמשיך לניתוח COMSTAT. הסר את כל רכיבי ה- OME-TIFF המקוריים מהתיקיה.
- בחלון התמונה, התאימו את המחוון לקצה הימני (כלומר, השכבה העליונה ביותר של הביופילם). כדי להסיר רעשי רקע, השתמש בחלון 'סף', המציג היסטוגרמה של התמונה, כדי להגדיר ידנית את ערכי הסף המרביים והמינימליים. תחילה, הגדר את ערך הסף המרבי על-ידי התאמת המחוון התחתון ימינה ככל האפשר. שנית, השתמשו במחוון העליון כדי להתאים את ערך הסף המינימלי, אשר מחלק את התמונה לשני שלבים נפרדים: ביומסה אדומה ורקע בגווני אפור (איור 3).
- הפעל את COMSTAT על-ידי בחירה באפשרות תוספים | קומסטט2 |. יופיע חלון 'אודות'. בחר אישור. יופיעו שלושה חלונות.
- בחלון 'ספריות נצפות' (בפינה השמאלית העליונה), בחר הוסף. אתר את תיקיית המקור, סמן מבלי לפתוח אותה ובחר בחר. יופיע חלון 'תמונות בספריות' (למעלה משמאל) המפרט את ה- OME-TIFFs שיש לנתח באמצעות COMSTAT.
- בחלון 'Comstat 2.1' (בפינה השמאלית התחתונה), בטל את הבחירה באפשרות 'סף אוטומטי (השיטה של Otsu)' כדי לוודא שהתוכנה משתמשת בערכי סף שהוגדרו בעבר עבור רכיבי OME-TIFF בודדים. כמו כן, בטל את הבחירה במסנן נפח מחוברg (CVF) כדי להבטיח שחלקים דקים מאוד של הביופילם, כמו גם תאים צפים חופשיים או ביומסה הנמצאים בתוך חללים של מבנה הביופילם נכללים בניתוח.
הערה: כאן, בטל את הבחירה ב- CVF מכיוון שניתוח COMSTAT מבוצע על ביופילמים מוקדמים מאוד (צמיחה ראשונית של 24 שעות) ותאים פלנקטוניים / מושבות שנותרו לאחר טיפול מיקרוביאלי. עבור ביופילמים בוגרים, בחר CVF כדי להבטיח שרק ביומסה המחוברת למבנה הביופילם מכומתת. - בחלון 'Comstat 2.1' (בפינה השמאלית התחתונה), בחר תכונות רצויות לניתוח כמותי. כאן, בחר Bio Mass, Thickness Distribution ו - Surface Area. בחר עבור כדי להפעיל את התוכנית. בחלון 'יומן' (למטה משמאל), נתוני הפלט מוצגים כמעבדים עד להופעת 'בוצע עם פונקציות/תמונות נבחרות!'. הקלט את מדידות COMSTAT. מדידות אלה נשמרות אוטומטית גם כקובצי TXT בתיקיית המקור.
תוצאות
מבודד P. aeruginosa בתרבית מחולה נגוע ב- CF משמש כדי להדגים את נקודות החוזק של גישה זו בכימות מדויק של שינויים הנגרמים על ידי מיקרוביאלית בארכיטקטורה של ביופילם במבחנה . זרימת העבודה הכוללת של מודל זה מיוצגת באיור 1. תהליך עיבוד התמונה וניתוח COMSTAT ב-ImageJ מוצג בא?...
Discussion
אין שיטה קבועה להשוואה כמותית של תמונות תלת-ממדיות של מבני ביופילם במבחנה , ולעתים קרובות קשה לתקנן הליכים המתוארים בהקשר זה בשל שונות בין-אופרטורית20. לפיכך, פרוטוקול זה מציע מסגרת פשוטה וניתנת לשחזור עבור יישומי COMSTAT המבקשים לכמת שינויים בארכיטקטורת ביופילם במבחנה...
Disclosures
ללא
Acknowledgements
המחברים מבקשים להודות לקרן סיסטיק פיברוזיס על מתן המימון למחקר זה.
Materials
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
References
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved