このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
COMSTATソフトウェアを使用した In vitro バイオフィルムアーキテクチャに対する抗菌剤の影響の定量化
要約
緑膿菌のバイオフィルム構造に対する抗菌薬による変化は、嚢胞性線維症および慢性肺感染症の患者から培養された臨床分離株によって異なります。共焦点顕微鏡法に続いて、COMSTATソフトウェアを使用して、個々の分離株のバイオフィルム構造(表面積、厚さ、バイオマスなど)の変動を定量化し、抗感染剤の有効性を評価できます。
要約
バイオフィルムは、細胞外高分子物質の自己生成マトリックスに依存して保護と構造的完全性を確保する微生物の集合体です。院内病原体である 緑膿菌は、 バイオフィルムの成長様式を採用し、嚢胞性線維症(CF)患者に慢性肺感染症を引き起こすことが知られています。コンピュータプログラムであるCOMSTATは、3次元共焦点画像からデータを抽出することにより、 緑膿菌の バイオフィルム構造における抗菌薬による変化を定量化するための有用なツールです。しかし、ソフトウェアの標準化された操作はあまり一般的ではなく、バイオフィルムの挙動を最適に報告し、中心を越えた比較を行うためには重要です。したがって、このプロトコルの目的は、COMSTATを介してさまざまな抗菌条件下で in vitro バイオフィルム構造を定量化するためのシンプルで再現性のあるフレームワークを提供することです。この手法は、CF P. aeruginosa 分離株を使用してモデル化され、バイオフィルム複製の形で成長させ、トブラマイシンと抗 Psl モノクローナル抗体 Psl0096 に曝露されます。この段階的なアプローチは、ユーザーの曖昧さを減らし、重要な画像処理ステップを見落とす可能性を最小限に抑えることを目的としています。具体的には、このプロトコルは、画像のセグメンテーションや適切な定量分析機能の選択など、COMSTATの手動操作に関連する主観的な変動の排除を強調しています。この方法では、COMSTATを実行する前に共焦点画像の処理に余分な時間を費やす必要がありますが、自動出力で誤って表現されたバイオフィルムの不均一性を最小限に抑えるのに役立ちます。
概要
バイオフィルムは、自己生成した細胞外高分子物質(EPS)のマトリックスに配向した微生物の凝集体です。EPSマトリックスは非常に複雑で、主に細菌細胞、水、タンパク質、多糖類、脂質、核酸1で構成されており、これらすべてがバイオフィルムを自由生活のプランクトン細胞とは明らかに異なるものにしています。バイオフィルムEPSは、互いに接着し、さまざまな表面に付着します。EPSマトリックスは、代謝物、遺伝物質、および細胞間シグナル伝達と防御に使用される化合物の細胞間交換を媒介する特性を持っています2。これらの特性は、バイオフィルムの構造的完全性と外部ストレス要因からの保護を総合的に提供し、免疫回避と抗菌耐性に貢献します3。
緑膿菌は、よく知られている院内病原体であり、抗菌薬に応答して回避的なバイオフィルム成長戦略を採用することが知られています。この代表的な例は、劣性遺伝性疾患である嚢胞性線維症(CF)の患者に発生します。バイオフィルムは、抗菌薬耐性緑膿菌4の発症に極めて重要な役割を果たし、CF患者の慢性肺感染症の確立を可能にし、肺機能の低下と早期死亡の加速を引き起こします5。したがって、in vitro バイオフィルム研究は、CF 6,7 患者から得られた緑膿菌分離株に対する抗生物質および新しい抗感染剤の有効性をテストするために実施されます。バイオフィルム形成後、抗菌剤を構造の外部に塗布し、共焦点レーザー走査型顕微鏡(CLSM)を使用して、バイオフィルムセグメントの高解像度の3次元再構成を生成します。その後、コンピュータソフトウェアであるCOMSTATをImageJのプラグインとして使用して、バイオフィルムアーキテクチャ8,9,10,11の変化を定量化するのが一般的な方法です。
COMSTATはバイオフィルム構造の定量化には有用ですが、画像解析の再現性と標準化についてはあまり注目されていません。例えば、COMSTAT を実行する前に実行されるイメージ処理プロシージャーは客観的ですが、イメージしきい値12,13 を設定する際には主観的な要素を含んでいます。同様に、COMSTATプログラムでは、オペレーターは画像セグメンテーションの基本条件から高度な条件、パラメータ、および10の定量分析機能(厚さ分布、表面積、バイオマス、無次元粗さ係数など)を選択できます。多数のユーザーオプションとさまざまなオペレーターの専門知識レベルが組み合わさると、バイオフィルムの動作に関する誤った報告につながる可能性があります。
したがって、このプロトコルの目標は、COMSTATを使用してin vitroバイオフィルム構造を定量的に比較するための比較的簡単な方法を提示することです。ここでは、CF P. aeruginosa分離株からのバイオフィルムセグメントの3次元画像を、再現性のあるin vitroバイオフィルム実験を行うために使用される確立された技術であるチャンバーカバーガラスモデル14を使用してCLSMを介してキャプチャします。この手法は、COMSTATをImageJのプラグインとして利用することで、研究者はさまざまな条件下で抗菌剤の存在下でのバイオフィルム構造の変化を定量的に特定することができます。全体として、この方法は、COMSTATの手動操作に関連する主観的なばらつきを排除することを目的としており、それによってセンター間でのプロトコルの標準化を促進します。
プロトコル
1.細菌分離物の収集
- SickKids(トロント)で吸入トブラマイシンによる根絶治療を受けているCFの小児患者のコホートから 緑膿菌 分離株を入手します。分離株をクエン酸グリセロールで-80°Cで凍結し、使用前に少なくとも3回サブカルチャーします。
2. in vitro バイオフィルム形成
注:改変を加えたin vitroバイオフィルム形成には、チャンバーカバーガラス法1を使用してください。このモデルの全体的なワークフローを図 1 に示します。
- 緑膿菌を分離した 緑膿菌 を、トリプシン性大豆寒天培地と5%羊の血液で調製した血液寒天培地で37°Cで一晩増殖させます( 材料の表を参照)。
- 血液寒天培地から1〜2個の細菌コロニーを4mLの溶解ブロス(LB)に接種します。225rpmに設定したシェーカーで37°Cで一晩成長させます。
- 4 mLの新鮮なLBに40 μLの培養物を加えて、一晩接種物の1:100希釈液を調製し、225 rpmに設定されたシェーカーで37°Cで3〜4時間増殖させ、600 nm(OD600)(初期対数期)で約0.1の光学密度を達成します。
- 220μLの接種材料を8チャンバーカバーガラススライドの各ウェルに移します。邪魔されずに37°Cで24時間インキュベートします。
- バイオフィルムがベースで剥がれるのを防ぐために、各ウェルから培地をゆっくりと取り出します。
注意: スライドを45°の角度で前方に傾け、ピペットチップでベースに触れずに、各チャンバーウェルの下部の角から培地を吸引します。 - 56 μg/mLの蛍光標識モノクローナル抗体(mAb)100 μLを調製し、指定のチャンバーウェルの側面にゆっくりと加えます。室温(RT)で1時間インキュベートし、細菌抗原エピトープに抗体が付着できるようにします。
注:使用前に、蛍光標識(赤)mAb Psl0096をLBで希釈して、最終濃度56 μg/mLを得てください。Psl0096は、抗Psl mAb(Cam003の最適化された親和性誘導体)であり、最初の細胞接着および構造的完全性に関与する 緑膿菌バイオ フィルムの主要なEPSマトリックス成分であるクラスI Pslエピトープに結合します15。 - 1000 μg/mLの抗生物質溶液100 μL( 材料表を参照)を調製し、指定されたチャンバーウェルの側面にゆっくりと加えます。邪魔されずに37°Cで24時間インキュベートします。
注:使用前に、LBで50 mg / mLのトブラマイシン抗生物質を希釈して、最終濃度1000 μg / mLを得ます。.
3. バイオフィルム蛍光染色
- 生細胞染色蛍光色素の0.01 mM溶液を調製します。チャンバー入りウェルから培地をゆっくりと取り出し、各ウェルに200 μLの染料混合物を加えます。RTで暗所で45分間インキュベートします。
注:使用前に、5 mMストック4 μLを2 mLのLBに加えて、生細胞染色(緑色)蛍光色素を調製します。 - 各ウェルから培地をゆっくりと取り出し、200μLの新鮮なLBで2回洗浄します。
- 各ウェルに200 μLの新鮮なLBを加え、共焦点顕微鏡による検査に進みます。
4. 共焦点顕微鏡による画像取得
注: 画像処理と COMSTAT 分析の手順を 図 2 に示します。バイオフィルム染色の同日にウェルの画像を取得します。視覚化の遅延が1時間を超える場合は、チャンバー付きカバーガラスを暗闇で冷蔵し、さらに処理します。
- 共焦点顕微鏡システム( 材料の表を参照)を使用して、適切なレーザー励起波長と取得用のフィルターセットを使用してウェルの画像を取得します。
注:ここでは、蛍光標識(赤)mAbおよび生細胞(緑)染色剤を、それぞれ561 nmおよび491 nmの励起波長を使用して励起します。 - 20〜25倍の水浸レンズを使用して、層状のzスタック画像(基層から各バイオフィルムセグメントの上部まで)を0.3μm刻みでキャプチャします。ウェルごとに少なくとも6つの画像スタックを取ります。
注:ここでは、25倍水浸レンズを備えた高解像度カメラを使用して画像を視覚化し、画像分析ソフトウェアを使用して処理します( 材料の表を参照)。ソフトウェアのセットアップとデジタルイメージングパラメータ(輝度と感度)は、1回の実験ですべての取得で一定に保ちます。 - COMSTAT解析のために画像をOME-TIFFとして保存します。
注意: OME-TIFFがチャネルごとに別々に保存されていることを確認してください(つまり、赤と緑)。この手順は、使用する画像解析ソフトウェアによって異なります。 - ステップ 2.1 から 4.3 を繰り返して、細菌分離株ごとに合計 3 つの生物学的複製 (つまり、3 つの独立した実験) から画像をキャプチャします。
5. COMSTAT分析
注:ImageJのプラグイン(Comstat2)として書き換えられた、無料で入手可能なコンピュータプログラムCOMSTAT16,17を使用して、画像を定量的に分析します。ダウンロードしたパッケージ内のバイオフィルムの画像スタックを分析するための一般的な手順をお読みください。この寄稿は、バイオフィルム形成に対する抗菌剤の影響を定量化するために推奨される、選択されたImageJ処理ステップとCOMSTAT機能を含む、要約されたプロトコルを提供します。
- http://www.comstat.dk/ から Comstat2 パッケージをダウンロードします。インストールされたフォルダ内で、ImageJを見つけて実行します。
- デスクトップ上にソースフォルダを作成し、そのフォルダに OME-TIFF を 1 つ追加します。
- ソースフォルダからOME-TIFFを開き、バイオマスを含まない空のレイヤーをすべて削除します。これらのレイヤーは、zスタックの最初または最後の数レイヤーになります。
注意: 顕微鏡で定義されたzスタック境界は、ユーザーによって過大評価されることがあります。これらの空のレイヤーを削除すると、COMSTAT 解析の Z スタック境界がより詳細に確立されます。 - ImageJ に OME-TIFF をインポートするには、[ ファイル] |インポート |画像シーケンス。ソースフォルダを見つけ、開かずに強調表示して、[ 選択]をクリックします。「シーケンスオプション」ウィンドウが表示されます。 [OK] を選択します。
注:ImageJに追加の画像をインポートするには、まずソースフォルダから以前のOME-TIFFを削除し、次に新しいOME-TIFFをフォルダに追加して、手順5.3と5.4を繰り返します。 - バイオフィルムの向きを反転するには、[ 画像] |トランスフォーム |[Z]を反転して、サブストラタムを最初(最上位)のスタックとして配置します。
注:COMSTATアルゴリズムは、上部(スタック1)から下部へのz方向のバイオフィルムを読み取ります。使用する共焦点顕微鏡システムによっては、OME-TIFF出力を反転させることができます。したがって、サブストラタムをイメージスタック1として配置することにより、スライスの順序を逆にして、出力データに欠陥が生じないようにすることが重要です。 - 画像のプロパティを定義するには、[ 画像] |プロパティ。「ソース」ウィンドウが表示されます。
- 「長さの単位」を「ミクロン」と指定します。
- 次の式を使用して、「ピクセル幅」と「ピクセル高さ」を数学的に決定します。
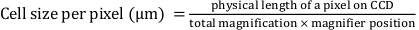 (1)
(1)
ここで、「ピクセル幅」と「ピクセル高さ」は 0.427 と定義され、電荷結合デバイス (CCD) 上のピクセルの物理的な長さは 16 μm です。総倍率は25倍です。拡大鏡の位置は1.5倍です。
注:ピクセルあたりのセルサイズの計算に使用される式は、顕微鏡カメラの製造元によって異なる場合があります。または、「ピクセル幅」と「ピクセル高さ」は、空間キャリブレーションによって定義できます(https://imagej.net/Spatial_Calibration を参照)。 - 「ボクセル深度」を 0.3 (つまり、各 Z スタック レイヤー間の増分スペース) として定義します。「ソース」ウィンドウで 「OK 」を選択します。
- 画像のしきい値を調整するには、[ Image |調整 |しきい値。「しきい値」ウィンドウが表示されます。
注:画像内のオブジェクトは、グレースケールの背景で赤く表示されます。あるいは、「Threshold」ウィンドウのドロップダウンメニューを使用して「Red」を「B&W」に変更することで、閾値を白黒で調整することもできます。- 画像ウィンドウで、スライダーを右端(つまり、バイオフィルムの最上層)に調整します。バックグラウンドノイズを除去するには、画像のヒストグラムを表示する「しきい値」ウィンドウを使用して、最大しきい値と最小しきい値を手動で設定します。まず、下のスライダーをできるだけ右に調整して、最大しきい値を設定します。次に、上のスライダーを使用して最小しきい値を調整し、画像を赤いバイオマスとグレースケールの背景の2つのフェーズに分割します(図3)。
注:本明細書に記載されている in vitro バイオフィルム形成および蛍光顕微鏡法は、理想的な場合にOME-TIFFを生成し、単純なヒストグラム閾値法によって画像を2つの異なるフェーズにセグメント化することを可能にする。ただし、場合によっては、異なるフェーズ間のヒストグラムの区別が明確でない場合があります。これは、広範なバックグラウンドノイズの存在、さまざまなバックグラウンド強度、またはバイオマスとバックグラウンド間の低強度のコントラストが原因である可能性があります。このような場合、ユーザは拡張セグメンテーション手順18、19を採用すべきである。- または、「しきい値」ウィンドウで「デフォルト」として設定された左側のドロップダウンメニューを使用して、個々の画像のしきい値をアルゴリズムで調整します。この機能では、17 種類のアルゴリズムしきい値オプションから選択できます (https://imagej.net/Auto_Threshold を参照)。最も適切なオプションを選択し、[自動]を選択してしきい値を設定します。
- しきい値を調整したら、画像ウィンドウのスライダーを使用して各レイヤーをスクロールし、バックグラウンドノイズが全体的に十分に除去されていることを確認します。
- 「しきい値」ウィンドウで 「設定 」を選択して、最初に下限しきい値を修正します。「Set Threshold Levels」ウィンドウが表示されます。 [OK] を選択します。もう一度 [設定 ] を選択し、この手順を繰り返して最大しきい値を修正します。
注意: 「設定」を選択するたびに、下のスライダーが自動的に調整される場合があります。このような場合は、スライダーを手動で再調整し(または、アルゴリズムのしきい値のいずれかを使用している場合は「自動」を選択)、手順5.6.2〜5.6.3を繰り返します。主な考え方は、スライダーが自動的に再調整されるたびに、上限と下限の両方のしきい値が固定されるように、後で「設定」を追加で2回選択する必要があるということです。 - [適用]を選択すると、[スタックをバイナリに変換]ウィンドウが表示されます。[OK] を選択し、[しきい値] ウィンドウを終了します。
- 新しく調整したOME-TIFFを保存するには、[ プラグイン]|バイオフォーマット |バイオフォーマットエクスポーター|.新しいファイル名を入力し、ソースフォルダにOME-TIFFとして保存します。「Bio-Formats Exporter – Multiple Files」ウィンドウが表示されます。 [OK] を選択します。「Bio-Formats Exporter Options」ウィンドウが表示されます。 [OK] を選択します。
メモ: COMSTAT 解析に進む前に、新しく調整された白黒の OME-TIFF のみがソース フォルダに保存されていることを確認してください。フォルダから元のOME-TIFFをすべて削除します。
- 画像ウィンドウで、スライダーを右端(つまり、バイオフィルムの最上層)に調整します。バックグラウンドノイズを除去するには、画像のヒストグラムを表示する「しきい値」ウィンドウを使用して、最大しきい値と最小しきい値を手動で設定します。まず、下のスライダーをできるだけ右に調整して、最大しきい値を設定します。次に、上のスライダーを使用して最小しきい値を調整し、画像を赤いバイオマスとグレースケールの背景の2つのフェーズに分割します(図3)。
- COMSTATを実行するには、[ Plugins |comstat2 |.「バージョン情報」ウィンドウが表示されます。 [OK] を選択します。3つのウィンドウが表示されます。
- 「監視ディレクトリ」ウィンドウ(右上)で、「 追加」を選択します。ソースフォルダを見つけ、開かずに強調表示して、[選択]を選択します。「Images in Directories」ウィンドウが表示されます(左上)COMSTATを介して分析するOME-TIFFが一覧表示されます。
- 「Comstat 2.1」ウィンドウ(右下)で、「自動しきい値設定(Otsuの方法)」の選択を解除して、ソフトウェアが個々のOME-TIFFに対して以前に設定されたしきい値を使用するようにします。また、 Connected Volume Filtering(CVF)の選択を解除して、バイオフィルムの非常に薄い部分、およびバイオフィルム構造のボイド内に見られる自由浮遊細胞またはバイオマスが分析に含まれるようにします。
注:COMSTAT分析は、抗菌治療後の非常に初期のバイオフィルム(24時間の初期増殖)および残りのプランクトン細胞/コロニーに対して行われるため、ここではCVFの選択を解除します。成熟バイオフィルムの場合は、CVFを選択して、バイオフィルム構造に接続されたバイオマスのみが定量化されるようにします。 - 「Comstat 2.1」ウィンドウ(右下)で、定量分析に必要な特徴を選択します。ここでは、[Bio Mass]、[Thickness Distribution]、[Surface Area] を選択します。[実行] を選択してプログラムを実行します。「ログ」ウィンドウ(左下)では、「選択した機能/画像で完了!」と表示されるまで、出力データが処理されている様子が表示されます。COMSTAT の測定値を記録します。これらの測定値は、ソースフォルダにTXTファイルとしても自動的に保存されます。
結果
CFに感染した患者から培養した 緑膿菌分離 株を用いて、 in vitro バイオフィルム構造における抗菌薬による変化を正確に定量化するこのアプローチの強みを実証します。このモデルの全体的なワークフローを 図 1 に示します。ImageJ での画像処理と COMSTAT 分析の手順を図 2 に示します。図 3 に、CLSM z スタック画?...
ディスカッション
in vitroバイオフィルム構造の3次元画像を定量的に比較するための規定された方法はなく、この文脈で説明される手順は、オペレーター間のばらつきのために標準化するのが難しいことがよくあります20。したがって、このプロトコルは、さまざまな抗菌条件下でのin vitroバイオフィルム構造の変化を定量化しようとするCOMSTATアプリケーションのためのシンプル?...
開示事項
何一つ
謝辞
著者らは、この研究に資金を提供してくれた嚢胞性線維症財団に感謝したいと思います。
資料
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
参考文献
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved