É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Quantificando os efeitos dos antimicrobianos na arquitetura de biofilme in vitro usando o software COMSTAT
Neste Artigo
Resumo
As alterações induzidas por antimicrobianos na arquitetura do biofilme de Pseudomonas aeruginosa diferem entre os isolados clínicos cultivados de pacientes com fibrose cística e infecção pulmonar crônica. Após a microscopia confocal, o software COMSTAT pode ser utilizado para quantificar variações na arquitetura do biofilme (por exemplo, área de superfície, espessura, biomassa) para isolados individuais para avaliar a eficácia de agentes anti-infecciosos.
Resumo
Os biofilmes são agregados de microrganismos que dependem de uma matriz autoproduzida de substância polimérica extracelular para proteção e integridade estrutural. O patógeno nosocomial, Pseudomonas aeruginosa, é conhecido por adotar um modo de crescimento de biofilme, causando infecção pulmonar crônica em pacientes com fibrose cística (FC). O programa de computador, COMSTAT, é uma ferramenta útil para quantificar as mudanças induzidas por antimicrobianos na arquitetura do biofilme de P. aeruginosa , extraindo dados de imagens confocais tridimensionais. No entanto, a operação padronizada do software é menos comumente abordada, o que é importante para o relatório ideal do comportamento do biofilme e comparação entre centros. Assim, o objetivo deste protocolo é fornecer uma estrutura simples e reprodutível para quantificar estruturas de biofilme in vitro sob diferentes condições antimicrobianas via COMSTAT. A técnica é modelada usando um isolado de CF P. aeruginosa , cultivado na forma de réplicas de biofilme e exposto à tobramicina e ao anticorpo monoclonal anti-Psl, Psl0096. A abordagem passo a passo visa reduzir a ambiguidade do usuário e minimizar a chance de ignorar etapas cruciais de processamento de imagem. Especificamente, o protocolo enfatiza a eliminação de variações subjetivas associadas à operação manual do COMSTAT, incluindo segmentação de imagem e a seleção de funções de análise quantitativa apropriadas. Embora esse método exija que os usuários gastem mais tempo processando imagens confocais antes de executar o COMSTAT, ele ajuda a minimizar a heterogeneidade de biofilme deturpada em saídas automatizadas.
Introdução
Os biofilmes são agregados de microrganismos orientados em uma matriz de substâncias poliméricas extracelulares (EPS) autoproduzidas. A matriz EPS é muito complexa, consistindo principalmente de células bacterianas, água, proteínas, polissacarídeos, lipídios e ácidos nucléicos1, todos os quais tornam os biofilmes distintamente diferentes das células planctônicas de vida livre. Os EPS de biofilme são aderentes uns aos outros e a várias superfícies. A matriz EPS possui propriedades que medeiam a troca célula a célula de metabólitos, material genético e compostos usados para sinalização e defesa intercelular2. Essas propriedades coletivamente fornecem integridade estrutural e proteção contra estressores externos, contribuindo para a evasão imunológica e resistência antimicrobiana3.
Pseudomonas aeruginosa é um patógeno nosocomial bem reconhecido, conhecido por adotar uma estratégia evasiva de crescimento de biofilme em resposta a antimicrobianos. Um excelente exemplo disso ocorre em pacientes com o distúrbio genético recessivo, fibrose cística (FC). Os biofilmes desempenham um papel fundamental no desenvolvimento de P. aeruginosa resistente a antimicrobianos 4 e permitem o estabelecimento de infecção pulmonar crônica em pacientes com FC, causando declínio acelerado da função pulmonar e mortalidade prematura5. Assim, estudos in vitro de biofilme são realizados para testar a eficácia de antibióticos e novos agentes anti-infecciosos contra isolados de P. aeruginosa obtidos de pacientes com FC 6,7. Após a formação do biofilme, os antimicrobianos são aplicados externamente à estrutura, e a microscopia confocal de varredura a laser (CLSM) é usada para gerar reconstruções tridimensionais de alta resolução de segmentos de biofilme. É prática comum usar o software de computador, COMSTAT, como um plugin para o ImageJ, para quantificar mudanças na arquitetura do biofilme 8,9,10,11.
Embora o COMSTAT seja útil para quantificar a estrutura do biofilme, a reprodutibilidade e a padronização da análise de imagens são menos comumente abordadas. Por exemplo, o procedimento de processamento de imagem, realizado antes da execução do COMSTAT, é objetivo, mas contém um elemento de subjetividade ao definir limites de imagem12,13. De maneira semelhante, o programa COMSTAT permite que o operador escolha entre condições e parâmetros básicos a avançados para segmentação de imagens, bem como dez funções de análise quantitativa (por exemplo, distribuição de espessura, área de superfície, biomassa, coeficiente de rugosidade adimensional). A multiplicidade de opções do usuário, combinada com os diferentes níveis de experiência do operador, pode resultar em relatórios equivocados do comportamento do biofilme.
Assim, o objetivo deste protocolo é apresentar um método relativamente simples para a comparação quantitativa de estruturas de biofilme in vitro usando COMSTAT. Aqui, imagens tridimensionais de segmentos de biofilme de um isolado de CF P. aeruginosa são capturadas via CLSM usando o modelo de lamínula com câmara14 - uma técnica estabelecida usada para realizar experimentos de biofilme in vitro reprodutíveis. Utilizando o COMSTAT como um plug-in para o ImageJ, esse método permite que os pesquisadores identifiquem quantitativamente as mudanças na arquitetura do biofilme na presença de antimicrobianos sob condições variadas. No geral, esse método visa eliminar variações subjetivas associadas à operação manual do COMSTAT, facilitando assim a padronização de protocolos entre os centros.
Protocolo
1. Coleta de isolados bacterianos
- Obtenha isolados de P. aeruginosa de uma coorte de pacientes pediátricos com FC submetidos a tratamento de erradicação com tobramicina inalada no SickKids (Toronto). Congelar isolados a -80 °C em citrato de glicerol e subcultura pelo menos três vezes antes da utilização.
2. Formação de biofilme in vitro
NOTA: Use um método de lamínula com câmara1 para formação de biofilme in vitro com modificações. O fluxo de trabalho geral desse modelo é mostrado na Figura 1.
- Cultive isolado de P. aeruginosa durante a noite a 37 ° C em ágar sangue preparado com ágar tríptico de soja e 5% de sangue de ovelha (consulte a Tabela de Materiais).
- Inocule 1–2 colônias bacterianas do ágar sangue em 4 mL de caldo de lisogenia (LB). Crescer durante a noite a 37 °C num shaker regulado para 225 rpm.
- Preparar uma diluição de 1:100 do inóculo durante a noite, adicionando 40 μL da cultura em 4 ml de LB fresco. Crescer durante 3-4 h a 37 °C num agitador regulado para 225 rpm, para atingir uma densidade óptica de aproximadamente 0,1 a 600 nm (OD600) (fase logarítmica inicial).
- Transferir 220 μL do inóculo para cada alvéolo de uma lâmina de lamínula de 8 câmaras. Incubar sem perturbações a 37 °C durante 24 h.
- Remova lentamente o meio de cada poço para evitar que os biofilmes se desprendam na base.
NOTA: Incline a corrediça para frente em um ângulo de 45° e aspire o meio dos cantos inferiores de cada poço com câmara sem tocar na base com a ponta da pipeta. - Prepare e adicione lentamente 100 μL de 56 μg/mL de anticorpo monoclonal (mAb) marcado com fluorescência (ver Tabela de Materiais) ao lado dos poços com câmaras designados. Incubar à temperatura ambiente (RT) durante 1 h para permitir a ligação do anticorpo ao epítopo do antigénio bacteriano.
NOTA: Antes de usar, dilua o mAb marcado com fluorescência (vermelho), Psl0096, em LB para obter uma concentração final de 56 μg / mL. Psl0096 é um mAb anti-Psl (derivado de afinidade otimizado de Cam003), que se liga ao epítopo Psl de classe I - um componente-chave da matriz EPS dos biofilmes de P. aeruginosa envolvidos na fixação celular inicial e integridade estrutural15. - Prepare e adicione lentamente 100 μL de uma solução antibiótica de 1000 μg / mL (consulte a Tabela de Materiais) ao lado dos poços com câmaras designados. Incubar sem perturbações a 37 °C durante 24 h.
NOTA: Antes de usar, dilua um estoque de 50 mg / mL de antibiótico tobramicina em LB para obter uma concentração final de 1000 μg / mL.
3. Coloração fluorescente de biofilme
- Prepare uma solução de 0,01 mM de um corante fluorescente de coloração de células vivas. Remova lentamente o meio dos poços com câmaras e adicione 200 μL da mistura de corante a cada poço. Incubar em RT no escuro por 45 min.
NOTA: Antes de usar, prepare o corante fluorescente de coloração de células vivas (verde) adicionando 4 μL de um estoque de 5 mM a 2 mL de LB. - Remova lentamente o meio de cada poço e lave 2x com 200 μL de LB fresco.
- Adicione 200 μL de LB fresco a cada poço e prossiga para o exame por microscopia confocal.
4. Aquisição de imagens por microscopia confocal
NOTA: O procedimento de processamento de imagem e análise COMSTAT é apresentado na Figura 2. Adquira imagens de poços no mesmo dia da coloração do biofilme. Se o atraso na visualização exceder 1 h, refrigere a lamínula com câmara no escuro até processamento posterior.
- Adquira imagens de poços usando um sistema de microscópio confocal (consulte a Tabela de Materiais) com comprimentos de onda de excitação a laser apropriados e conjuntos de filtros para aquisição.
NOTA: Aqui, excite o mAb marcado com fluorescência (vermelho) e a coloração de células vivas (verde) usando comprimentos de onda de excitação de 561 e 491 nm, respectivamente. - Capture imagens de pilha z em camadas (do substrato até o topo de cada segmento de biofilme) em incrementos de 0,3 μm com uma lente de imersão em água de 20 a 25x. Pegue pelo menos 6 pilhas de imagens por poço.
NOTA: Aqui, visualize as imagens usando uma câmera de alta resolução com uma lente de imersão em água de 25x e processadas usando software de análise de imagem (consulte a Tabela de Materiais). Mantenha a configuração do software e os parâmetros de imagem digital (ou seja, brilho e sensibilidade) constantes para todas as aquisições em um único experimento. - Salve imagens como OME-TIFFs para análise COMSTAT.
NOTA: Certifique-se de que os OME-TIFFs sejam salvos separadamente para cada canal (ou seja, vermelho e verde). Esta etapa varia de acordo com o software de análise de imagem usado. - Repita as etapas 2.1 a 4.3 para capturar imagens de um total de 3 réplicas biológicas (ou seja, 3 experimentos independentes) por isolado bacteriano.
5. Análise COMSTAT
NOTA: Analise as imagens quantitativamente usando o programa de computador disponível gratuitamente, COMSTAT 16,17, reescrito como um plug-in (Comstat2) para ImageJ. Leia as instruções gerais para analisar pilhas de imagens de biofilmes dentro do pacote baixado. Esta contribuição fornece um protocolo resumido, com etapas de processamento ImageJ selecionadas e recursos COMSTAT recomendados para quantificar os efeitos dos antimicrobianos na formação de biofilme.
- Baixe o pacote Comstat2 do http://www.comstat.dk/. Na pasta instalada, localize o ImageJ e execute-o.
- Crie uma pasta de origem na área de trabalho e adicione um único OME-TIFF à pasta.
- Abra o OME-TIFF da pasta de origem e exclua todas as camadas vazias que não contenham biomassa. Essas camadas serão a primeira ou as últimas camadas da pilha z.
NOTA: Os limites da pilha z definidos pelo microscópio às vezes são superestimados pelos usuários. A exclusão dessas camadas vazias estabelece um limite de pilha z mais refinado para análise COMSTAT. - Importe o OME-TIFF no ImageJ selecionando Arquivo | Importação | Sequência de imagens. Localize a pasta de origem, realce sem abri-la e clique em Selecionar. Uma janela 'Opções de sequência' aparecerá. Selecione OK.
NOTA: Para importar imagens adicionais no ImageJ, primeiro remova o OME-TIFF anterior da pasta de origem, adicione o novo OME-TIFF à pasta e repita as etapas 5.3 e 5.4. - Inverta a orientação do biofilme selecionando Imagem | Transformar | Vire Z para posicionar o substrato como a primeira pilha (superior).
NOTA: Os algoritmos COMSTAT leem biofilmes na direção z de cima (pilha 1) para baixo. Dependendo do sistema de microscópio confocal usado, a saída OME-TIFF pode ser invertida. Assim, é importante inverter a ordem das fatias posicionando o substrato como pilha de imagens 1 para evitar que os dados de saída se tornem falhos. - Defina as propriedades da imagem selecionando Imagem | Propriedades. Uma janela 'Fonte' aparecerá.
- Especifique 'Unidade de comprimento' como 'mícron'.
- Determine matematicamente 'Largura do pixel' e 'Altura do pixel' usando a seguinte equação:
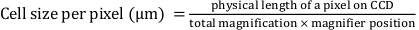 (1)
(1)
Aqui, 'Largura do pixel' e 'Altura do pixel' são definidos como 0,427, onde o comprimento físico de um pixel no dispositivo de carga acoplada (CCD) é de 16 μm; a ampliação total é de 25x; e a posição da lupa é 1.5x.
NOTA: A equação usada para calcular o tamanho da célula por pixel pode variar dependendo do fabricante da câmera do microscópio. Como alternativa, 'Largura do pixel' e 'Altura do pixel' podem ser definidas por calibração espacial (consulte https://imagej.net/Spatial_Calibration). - Defina 'Profundidade do voxel' como 0,3 (ou seja, espaço incremental entre cada camada da pilha z). Selecione OK na janela 'Fonte'.
- Ajuste o limite da imagem selecionando Imagem | Ajustar | Limiar. Uma janela 'Limite' aparecerá.
NOTA: Os objetos na imagem aparecerão em vermelho com um fundo em tons de cinza. Alternativamente, o limite pode ser ajustado em preto e branco, usando o menu suspenso na janela 'Limiar' para alterar 'Vermelho' para 'P&B'.- Na janela de imagem, ajuste o controle deslizante para a extrema direita (ou seja, a camada superior do biofilme). Para remover o ruído de fundo, use a janela 'Threshold', que exibe um histograma da imagem, para definir manualmente os valores de limite máximo e mínimo. Primeiro, defina o valor máximo do limite ajustando o controle deslizante inferior o mais à direita possível. Em segundo lugar, use o controle deslizante superior para ajustar o valor limite mínimo, que segmenta a imagem em duas fases separadas: biomassa vermelha e fundo em tons de cinza (Figura 3).
NOTA: O procedimento de formação de biofilme in vitro e microscopia fluorescente aqui descrito gera OME-TIFFs no caso ideal, permitindo que as imagens sejam segmentadas em duas fases distintas por meio de um método simples de limiar de histograma. No entanto, em alguns casos, a distinção do histograma entre as diferentes fases não é tão clara. Isso pode ser devido à presença de ruído de fundo extenso, intensidades de fundo variadas ou um contraste de baixa intensidade entre a biomassa e o fundo. Nesses casos, os usuários devem adotar um procedimento de segmentação aprimorado 18,19.- Como alternativa, ajuste os limites algoritmicamente para imagens individuais usando o menu suspenso à esquerda definido como 'Padrão' na janela 'Limiar'. Esse recurso fornece 17 opções de limite algorítmico diferentes para escolher (consulte https://imagej.net/Auto_Threshold). Selecione a opção mais aplicável e, em seguida, 'Auto' para definir o limite.
- Quando os valores limite forem ajustados, use o controle deslizante na janela de imagem para rolar por cada camada para garantir que o ruído de fundo seja suficientemente removido.
- Selecione Definir na janela 'Limite' para corrigir primeiro o valor limite inferior. Uma janela 'Definir níveis de limite' aparecerá. Selecione OK. Selecione Definir novamente e repita esta etapa para corrigir o valor limite máximo.
NOTA: Cada vez que 'Definir' é selecionado, o controle deslizante inferior pode ser ajustado automaticamente. Nesses casos, reajuste manualmente o controle deslizante (ou selecione 'Automático' se estiver usando um dos limites algorítmicos) e repita as etapas 5.6.2 a 5.6.3. A ideia principal é que sempre que os controles deslizantes forem reajustados automaticamente, 'Definir' deve ser selecionado mais duas vezes depois para garantir que os limites superior e inferior sejam fixos. - Selecione Aplicar e uma janela 'Converter pilha em binário' aparecerá. Selecione OK e saia da janela 'Limiar'.
- Salve o OME-TIFF recém-ajustado selecionando Plug-ins | Bio-Formatos | Exportador de Bio-Formatos|. Insira um novo nome de arquivo e salve como OME-TIFF na pasta de origem. Uma janela 'Bio-Formats Exporter – Multiple Files' aparecerá. Selecione OK. Uma janela 'Opções do exportador de bioformatos' aparecerá. Selecione OK.
NOTA: Certifique-se de que apenas os OME-TIFFs em preto e branco recém-ajustados sejam salvos na pasta de origem antes de prosseguir para a análise COMSTAT. Remova todos os OME-TIFFs originais da pasta.
- Na janela de imagem, ajuste o controle deslizante para a extrema direita (ou seja, a camada superior do biofilme). Para remover o ruído de fundo, use a janela 'Threshold', que exibe um histograma da imagem, para definir manualmente os valores de limite máximo e mínimo. Primeiro, defina o valor máximo do limite ajustando o controle deslizante inferior o mais à direita possível. Em segundo lugar, use o controle deslizante superior para ajustar o valor limite mínimo, que segmenta a imagem em duas fases separadas: biomassa vermelha e fundo em tons de cinza (Figura 3).
- Execute o COMSTAT selecionando Plug-ins | Comstat2 |. Uma janela 'Sobre' aparecerá. Selecione OK. Três janelas aparecerão.
- Na janela 'Diretórios observados' (canto superior direito), selecione Adicionar. Localize a pasta de origem, realce sem abri-la e selecione Escolher. Uma janela 'Imagens em diretórios' aparecerá (canto superior esquerdo) listando os OME-TIFFs a serem analisados via COMSTAT.
- Na janela 'Comstat 2.1' (canto inferior direito), desmarque 'Limite automático (método de Otsu)' para garantir que o software use valores de limite previamente configurados para OME-TIFFs individuais. Além disso, desmarque o Filtro de Volume Conectado(CVF) para garantir que partes muito finas do biofilme, bem como células flutuantes livres ou biomassa encontradas dentro dos vazios da estrutura do biofilme, sejam incluídas na análise.
NOTA: Aqui, desmarque o CVF porque a análise COMSTAT é realizada em biofilmes muito precoces (crescimento inicial de 24 horas) e células/colônias planctônicas remanescentes após o tratamento antimicrobiano. Para biofilmes maduros, selecione CVF para garantir que apenas a biomassa conectada à estrutura do biofilme seja quantificada. - Na janela 'Comstat 2.1' (canto inferior direito), selecione os recursos desejados para análise quantitativa. Aqui, selecione Biomassa, Distribuição de espessura e Área de superfície. Selecione Ir para executar o programa. Na janela 'Log' (canto inferior esquerdo), os dados de saída são mostrados processando até que 'Concluído com funções/imagens selecionadas!' apareça. Registre as medições COMSTAT. Essas medições também são salvas automaticamente como arquivos TXT na pasta de origem.
Resultados
Um isolado de P. aeruginosa cultivado de um paciente infectado com FC é usado para demonstrar os pontos fortes dessa abordagem na quantificação precisa das alterações induzidas por antimicrobianos na arquitetura do biofilme in vitro . O fluxo de trabalho geral desse modelo é representado na Figura 1. O procedimento de processamento de imagem e análise COMSTAT no ImageJ é mostrado na Figura 2. Uma abordagem simples de limite de histogram...
Discussão
Não existe um método prescrito para comparar quantitativamente imagens tridimensionais de estruturas de biofilme in vitro , e os procedimentos descritos neste contexto são muitas vezes difíceis de padronizar devido à variabilidade interoperadora20. Assim, este protocolo oferece uma estrutura simples e reprodutível para aplicações COMSTAT que buscam quantificar mudanças na arquitetura do biofilme in vitro sob diferentes condições antimicrobianas. Os pontos fortes desta ...
Divulgações
Nenhum
Agradecimentos
Os autores gostariam de agradecer à Cystic Fibrosis Foundation por fornecer financiamento para esta pesquisa.
Materiais
| Name | Company | Catalog Number | Comments |
| Anti-Psl mAb, Psl0096 | Medimmune | ||
| Blood Agar (TSA with 5 % Sheep Blood) Medium | Fisher Scientific | R01200 | |
| Eight-well Chambered Coverglass w/ non-removable wells | Thermo Fisher Scientific | 155411 | |
| Invitrogen SYTO 9 Green Fluorescent Nucleic Acid Stain | Thermo Fisher Scientific | S34854 | |
| LB BROTH (LENNOX), Liquid Autoclave Sterilized | BioShop Canada | LBL666 | |
| Tobramycin, 900 µg/mg | Alfa Aesar by Thermo Fisher Scientific | J66040 | It is recommended to perform a minimal inhibitory concentration (MIC) test for every batch made to ensure quality control of antimicrobial potency |
| Quorum Volocity 6.3 | Quorum Technologies | Image analysis software |
Referências
- Flemming, H. C., Wingender, J. The biofilm matrix. Nature Reviews Microbiology. 8, 623-633 (2010).
- Flemming, H. C., et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology. 14, 563-575 (2016).
- Rybtke, M., Hultqvist, L. D., Givskov, M., Tolker-Nielsen, T. Pseudomonas aeruginosa biofilm infections: community structure, antimicrobial tolerance and immune response. Journal of Molecular Biology. 427, 3628-3645 (2015).
- Wendel, A. F., Ressina, S., Kolbe-Busch, S., Pfeffer, K., MacKenzie, C. R. Species diversity of environmental GIM-1-producing bacteria collected during a long-term outbreak. Applied and Environmental Microbiology. 82, 3605-3610 (2016).
- Costerton, J. W., Stewart, P. S., Greenberg, E. P. Bacterial biofilms: a common cause of persistent infections. Science. 284, 1318-1322 (1999).
- Powell, L. C., et al. Targeted disruption of the extracellular polymeric network of Pseudomonas aeruginosa biofilms by alginate oligosaccharides. NPJ Biofilms and Microbiomes. 4, 1-10 (2018).
- Ciofu, O., Tolker-Nielsen, T., Jensen, P. &. #. 2. 1. 6. ;., Wang, H., Høiby, N. Antimicrobial resistance, respiratory tract infections and role of biofilms in lung infections in cystic fibrosis patients. Advanced Drug Delivery Reviews. 85, 7-23 (2015).
- Landry, R. M., An, D., Hupp, J. T., Singh, P. K., Parsek, M. R. Mucin-Pseudomonas aeruginosa interactions promote biofilm formation and antibiotic resistance. Molecular Microbiology. 59, 142-151 (2006).
- Beaudoin, T., et al. Staphylococcus aureus interaction with Pseudomonas aeruginosa biofilm enhances tobramycin resistance. NPJ Biofilms and Microbiomes. 3, 1-9 (2017).
- Rojo-Molinero, E., et al. Sequential treatment of biofilms with aztreonam and tobramycin is a novel strategy for combating Pseudomonas aeruginosa chronic respiratory infections. Antimicrobial Agents and Chemotherapy. 60, 2912-2922 (2016).
- Hentzer, M., et al. Alginate overproduction affects Pseudomonas aeruginosa biofilm structure and function. Journal of Bacteriology. 183, 5395-5401 (2001).
- Tolker-Nielsen, T., Sternberg, C. Growing and analyzing biofilms in flow chambers. Current Protocols in Microbiology. 21, 1-17 (2011).
- Luo, T. L., et al. A Sensitive thresholding method for confocal laser scanning microscope image stacks of microbial biofilms. Scientific Reports. 8, 1-14 (2018).
- Beaudoin, T., Kennedy, S., Yau, Y., Waters, V. Visualizing the effects of sputum on biofilm development using a chambered coverglass model. Journal of Visualized Experiments. (118), e54819 (2016).
- DiGiandomenico, A., et al. Identification of broadly protective human antibodies to Pseudomonas aeruginosa exopolysaccharide Psl by phenotypic screening. Journal of Experimental Medicine. 209, 1273-1287 (2012).
- Heydorn, A., et al. Quantification of biofilm structures by the novel computer program COMSTAT. Microbiology. 146, 2395-2407 (2000).
- Vorregaard, M. Comstat2 - a modern 3D image analysis environment for biofilms, in Informatics and Mathematical Modelling. Technical University of Denmark. , (2008).
- Hashemi, M. A., Khaddour, G., François, B., Massart, T. J., Salager, S. A tomographic imagery segmentation methodology for three-phase geomaterials based on simultaneous region growing. Acta Geotechnica. 9, 831-846 (2014).
- Rogowska, J. Overview and fundamentals of medical image segmentation. Handbook of Medical Image Processing and Analysis. , 73-90 (2009).
- Webb, D., et al. Assessing technician effects when extracting quantities from microscope images. Journal of Microbiological Methods. 53, 97-106 (2003).
- Azeredo, J., et al. Critical review on biofilm methods. Critical Reviews in Microbiology. 43, 313-351 (2017).
- Xavier, J. B., et al. Objective threshold selection procedure (OTS) for segmentation of scanning laser confocal microscope images. Journal of Microbiological Methods. 47, 169-180 (2001).
- Arena, E. T., et al. Quantitating the cell: turning images into numbers with ImageJ. Wiley Interdisciplinary Reviews: Developmental Biology. 6, 260 (2017).
- Daims, H., Wagner, M. Quantification of uncultured microorganisms by fluorescence microscopy and digital image analysis. Applied Microbiology and Biotechnology. 75, 237-248 (2007).
- Yerly, J., Hu, Y., Jones, S. M., Martinuzzi, R. J. A two-step procedure for automatic and accurate segmentation of volumetric CLSM biofilm images. Journal of Microbiological Methods. 70, 424-433 (2007).
- Lee, B., et al. Heterogeneity of biofilms formed by nonmucoid Pseudomonas aeruginosa isolates from patients with cystic fibrosis. Journal of Clinical Microbiology. 43, 5247-5255 (2005).
- Stapper, A. P., et al. Alginate production affects Pseudomonas aeruginosa biofilm development and architecture, but is not essential for biofilm formation. Journal of Medical Microbiology. 53, 679-690 (2004).
- Reichhardt, C., Parsek, M. Confocal laser scanning microscopy for analysis of Pseudomonas aeruginosa biofilm architecture and matrix localization. Frontiers in Microbiology. 10, 677 (2019).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Quantifying biofilm structure using image analysis. Journal of Microbiological Methods. 39, 109-119 (2000).
- Yang, X., Beyenal, H., Harkin, G., Lewandowski, Z. Evaluation of biofilm image thresholding methods. Water Research. 35, 1149-1158 (2001).
- Ross, S. S., et al. Quantification of confocal images of biofilms grown on irregular surfaces. Journal of Microbiological Methods. 100, 111-120 (2014).
- Ma, L., et al. Assembly and development of the Pseudomonas aeruginosa biofilm matrix. PLoS Pathogens. 5, (2009).
- Mah, T. F., et al. A genetic basis for Pseudomonas aeruginosa biofilm antibiotic resistance. Nature. 426, 306-310 (2003).
- Srinandan, C. S., Jadav, V., Cecilia, D., Nerurkar, A. S. Nutrients determine the spatial architecture of Paracoccus sp. biofilm. Biofouling. 26, 449-459 (2010).
- Ramos, I., Dietrich, L. E., Price-Whelan, A., Newman, D. K. Phenazines affect biofilm formation by Pseudomonas aeruginosa in similar ways at various scales. Research in Microbiology. 161, 187-191 (2010).
- Ma, L., Jackson, K. D., Landry, R. M., Parsek, M. R., Wozniak, D. J. Analysis of Pseudomonas aeruginosa conditional psl variants reveals roles for the psl polysaccharide in adhesion and maintaining biofilm structure postattachment. Journal of Bacteriology. 188, 8213-8221 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados