A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
مقايسة كيميائية أسيتيل كليك لقياس هيستون أسيتيل ترانسفيراز 1 أسيتيل
In This Article
Summary
تعد المقايسات الكيميائية السريعة والدقيقة للكشف عن مثبطات محددة أداة مهمة في ترسانة تطوير الأدوية. هنا ، نقدم مقايسة كيميائية قابلة للتطوير فوق الأسيتيل لقياس تثبيط نشاط أستلة HAT1.
Abstract
يلعب HAT1 ، المعروف أيضا باسم Histone acetyltransferase 1 ، دورا مهما في تخليق الكروماتين عن طريق تثبيت وأستلة H4 الوليدة قبل تجميع النيوكليوسوم. وهو مطلوب لنمو الورم في أنظمة مختلفة ، مما يجعله هدفا محتملا لعلاج السرطان. لتسهيل تحديد المركبات التي يمكن أن تمنع النشاط الأنزيمي HAT1 ، ابتكرنا مقايسة نقرة الأسيتيل للفحص السريع. في هذا الفحص البسيط ، نستخدم HAT1 / Rbap46 المؤتلف ، والذي يتم تنقيته من الخلايا البشرية المنشطة. تستخدم الطريقة الأسيتل-CoA التناظري 4-pentynoyl-CoA (4P) في نهج كيمياء النقر. يتضمن ذلك النقل الأنزيمي لمقبض الألكاين من خلال تفاعل أسيلة يعتمد على HAT1 إلى ببتيد H4 N-terminal البيوتينيل. ثم يتم تجميد الببتيد الذي تم التقاطه على ألواح النيوترفيدين ، متبوعا بوظيفة كيمياء النقر باستخدام البيوتين أزيد. بعد ذلك ، يتم استخدام توظيف streptavidin-peroxidase لأكسدة amplex red ، مما يؤدي إلى ناتج فلوري كمي. من خلال إدخال مثبطات كيميائية أثناء تفاعل الأسيل ، يمكننا تحديد التثبيط الأنزيمي بناء على تقليل إشارة التألق. الأهم من ذلك ، أن هذا التفاعل قابل للتطوير ، مما يسمح بفحص عالي الإنتاجية للمثبطات المحتملة للنشاط الأنزيمي HAT1.
Introduction
من بين العديد من أسيتيل ترانسفيراز حقيقية النواة ، كان HAT1 هو أسيتيل ترانسفيراز هيستون الأولي الذي تم عزله1،2،3. أثبتت التحقيقات اللاحقة دورها المحوري في تكرار الكروماتين ، لا سيما في تخليق نيوكليوسومات جديدة خلال المرحلةS-phase 4. أدت مساعينا البحثية إلى إدراك أن HAT1 يتم تحفيزه بشكل كبير من خلال علاج عامل نمو البشرة (EGF) في الخلايا الثديية5. علاوة على ذلك ، فقد تبين أن HAT1 مطلوب للتكاثر السريع للخلايا وتشكيل الورم في الجسم الحي6،7،8،9. تشير البيانات إلى أن HAT1 أمر بالغ الأهمية في تنسيق العمليات الابتنائية واللاجينية لانقسام الخلايا ، مما يؤدي إلى نمو الورم.
HAT1 ثنائي الأسيتيلات الذيل الأميني الطرفي للهيستون H4 على الليسين 5 و 12 في مركب مع بروتين chaperone Rbap46 ، الذي يربط الهستون ويقدم المحطة الأمينية إلى HAT1. ثم يتم استيراد رباعيات الهستون أو disomes10 ، جنبا إلى جنب مع HAT1 / Rbap46 وغيرها من مرافقي هيستون11 ، إلى النواة. ثم يتم إطلاق الهستونات ليتم إيداعها في شوكة النسخ المتماثل ، أو مواقع أخرى لدعم تنشيط الجينات أو قمعها. وظيفة علامة HAT1 di-acetylation على هيستون H4 ليست مفهومة تماما. من المحتمل إزالته بسرعة في غضون 15-30 دقيقة بفعل هيستون ديسيتيلاز12،13،14،15 بعد إدخال H4 في الكروماتين. وبالتالي ، لا يتم نشر علامة HAT1 di-acetylation في الكروماتين وقد لا تؤدي دورا جينيا حقيقيا ، على الرغم من افتراض دور في تجنيد الإنزيمات المعدلة للكروماتين إلى الكروماتينالناشئ 12. أيضا ، HAT1 لا أسيتيل الكروماتين مباشرة. يقتصر نشاطها على الهستونات القابلة للذوبان.
تم إعاقة تطوير مثبطات هيستون أسيتيل ترانسفيراز ذات الجزيئات الصغيرة بسبب المقايسات غير المحددة ومنخفضة الإنتاجية ، مما يؤدي في كثير من الأحيان إلى توليد مركبات تفاعلية بيولوجيا16،17. يتطلب اختبار المعيار الذهبي لقياس أنشطة أسيتيل ترانسفيراز استخدام 3H-acetyl-CoA ، مما يحد من الإنتاجية ويتطلب الإشعاع. ومع ذلك ، في الآونة الأخيرة ، تم وصف وتأكيد مثبطات أسيتيل ترانسفيراز جزيء صغير محددة وقوية للغاية تستهدف CBP / p30018 و KAT6A / B19،20 من خلال استخدام 3H-acetyl-CoA. للمضي قدما ، يتم وضع فحوصات محسنة لتحقيق إنتاجية أفضل وتجنب المخاطر المختبرية.
استخدمت التطورات الحديثة في مراقبة أستلة21 كيمياء النقر لتمكين مراقبة التفاعل الأنزيمي. هناك مجموعة متنوعة من السلائف التي تدعم النقر والتي يمكن الوصول إليها عن طريق طرق اصطناعية بسيطة أو متاحة للشراء والتي يمكن دمجها في تفاعلات الإنزيم. عادة ما يتم إجراء هذه التفاعلات في الأنظمة المؤتلفة ، على الرغم من أن المقايسات القائمة على الخلايا ممكنة أيضا22. تتمثل ميزة العوامل والركائز التي تدعم النقر في أن الفحص يمكن أن يقيس نشاط الإنزيم مباشرة دون الحاجة إلى أنظمة قراءة مقترنة غالبا ما تزعجها مركبات الفحص وتتطلب خطوات معالجة إضافية. وهذا يسمح بمعالجة المثبطات فقط خلال الخطوة الأنزيمية ، في حين يتم تنفيذ جميع خطوات التشغيل والكشف النهائية بعد الغسيل المكثف لإزالة المركبات ، مما يحد من احتمال حدوث تداخل المقايسة. هذه المزايا تجعل تصميم المقايسات التي تدعم النقر أفضل من المقايسات المقترنة التي تعتمد عادة على اكتشاف أنزيم A الحر.
أحد الاعتبارات المهمة هو قبول العوامل المساعدة التي تدعم النقر في الموقع النشط للإنزيم. قد لا تكون العوامل المشتركة الحالية التي تم تمكين النقر عليها متوافقة تماما مع الموقع النشط المحسن للعامل المساعد الأصلي. يمكن استخدام المعلومات الهيكلية والنمذجة لتصميم بدائل الأحماض الأمينية لتوسيع الموقع النشط لدمج الركائز المعدلة23. قد يتيح ذلك الفحص باستخدام حركية إنزيم محسنة ومستويات أقل من الركيزة والإنزيم. عيب هذا النهج هو أن الجيوب التحفيزية المتغيرة قد لا تحدد المثبطات التي تتفاعل بقوة مع الإنزيم الأصلي. في النهاية ، هناك حاجة إلى مجموعة من الأساليب لتحديد مثبطات الإنزيم المحتملة والتحقق من صحتها.
هنا ، نصف طريقة تم تطويرها لتنقية وفحص نشاط إنزيم HAT1 باستخدام العامل المساعد للنقر 4-pentynoyl-CoA24. يستخدم هذا الفحص (الشكل 1) تسلسل الإنزيم الأصلي في مركب مع البروتين الشريك المطلوب Rbap46 ، والذي ثبت أنه يعزز نشاط الإنزيم. يسمح تنقية الإنزيم من الخلايا البشرية بتنشيط الإنزيم في السليلولو ، مما قد يحافظ على تحفيز تعديلات ما بعد الترجمة المهمة لنشاط الإنزيم الكامل. تم استخدام تصميم وتحسين مقايسات الإنزيم المؤتلف للشاشات الكيميائية عالية الإنتاجية بنجاح لتحديد وتوصيف مثبطات الجزيئات الصغيرة HAT1.
Protocol
1. الطريقة الأولى: إنتاج وتنقية مركب HAT1 / Rbap46 المؤتلف
- إذابة خلايا HEK293f واستعادتها وتوسيعها
- قم بإذابة 1-10 مليون خلية ثديية HEK293f26 إلى وسائط تعبير حرة 293 سعة 30 مل في قارورة سعة 100 مل. احتضان في 8٪ CO2 عند 37 درجة مئوية أثناء الدوران عند 60 دورة في الدقيقة.
- في اليوم التالي ، عد وتحقق من صلاحية الخلية ، ثم اضبط سرعة الدوران على 120 دورة في الدقيقة. قم بالتوسع إلى 300 مل في دورق سعة 1 لتر ، مع الحفاظ على كثافة البذر عند 500000 خلية / مل وتقسيم الخلايا قبل أن تتجاوز الكثافة 3 × 106 خلايا / مل.
- نقل HEK293f
- بذرة 5 × 105 خلايا / مل في مزرعة 300 مل في دورق 1 لتر وثقافة لمدة 24 ساعة. في اليوم التالي ، عد الخلايا للتأكد من أنها تتراوح بين 7.5 × 105 إلى 1.2 × 106 خلايا / مل.
- تحضير خليط من 300 ميكروغرام من pHEK-FLAG-HAT1 و 300 ميكروغرام من الحمض النووي البلازميد pHEK-Rbap46 في 30 مل من PBS. أضف 1.2 مل من البولي إيثيلين (PEI) إلى الحمض النووي / PBS واخلطه. احتضان لمدة 20 دقيقة في RT. أضف مزيج DNA / PBS / PEI إلى مزرعة 300 مل واحتضانها عند 37 درجة مئوية لمدة 48 ساعة عند 120 دورة في الدقيقة.
- حصاد الخلايا
- عد الخلايا للتأكد من أنها تقع بين 1.7 × 106 إلى 2.5 × 106 خلايا / مل. عند كثافة الخلية هذه ، أضف فورسكولين إلى تركيز نهائي يبلغ 12.5 ميكرومتر لتنشيط HAT1 واحتضان الخلايا لمدة 30 دقيقة ، 37 درجة مئوية ، 120 دورة في الدقيقة.
- قم بإذابة الخلايا عند 300 × جم لمدة 5 دقائق ، واغسلها مرة واحدة باستخدام 30 مل من PBS ، وقم بتجميدها في السائل N2. يحفظ في درجة حرارة -80 درجة مئوية حتى تنقية البروتين أو ينتقل مباشرة إلى التنقية.
- HAT1 / Rbap46 مجمع تنقية حبة العلم
- تحلل الخلايا وإعداد مستخلص البروتين.
- تحضير محلول التحلل عن طريق إضافة قرص واحد من كوكتيل مثبطات الأنزيم البروتيني (PIC) إلى 50 مل من RSB-500 (20 مللي متر Tris-HCl pH 7.5 ، 500 mM NaCl ، 25 mM MgCl2).
- قم بإذابة حبيبات الخلية من مزرعة 300 مل على الثلج والتحلل باستخدام 40 مل من محلول التحلل المثلج البارد مع 0.1٪ Triton X-100. قم بالدوران على الجليد ، ثم قم بتدويره لأسفل عند 10000 × جم لمدة 10 دقائق عند 4 درجات مئوية. اجمع كل المواد الطافية في أنبوب واحد سعة 50 مل على الثلج ، وهو الآن مستخلص البروتين.
- إعداد حبات العلم.
- قسم حبات FLAG إلى أربعة أنابيب مخروطية سعة 15 مل عن طريق إضافة 400 ميكرولتر من ملاط حبة الأغاروز M2-FLAG في كل أنبوب يحتوي على 5 مل من 0.1 M Glycine pH 3.5 ، 0.01٪ Triton X-100. رج كل أنبوب بقوة لمدة 5 ثوان باليد.
- أجهزة طرد مركزي نبضية لمدة 30 ثانية عند 1000 × جم عند 4 درجات مئوية ، نضح طاف ، يغسل مرتين ب 5 مل من RSB-500 + 0.1٪ Triton X-100 (Tx-100 ؛ بدون مثبطات الأنزيم البروتيني) ، ويحضن على الجليد.
- أداء الترسيب المناعي FLAG.
- أضف 10 مل من مستخلص البروتين إلى كل أنبوب مخروطي سعة 15 مل يحتوي على حبات مغسولة. احتضان الأنابيب عند 4 درجات مئوية مع دوران مقلوب لمدة 90 دقيقة على الأقل أو طوال الليل.
- اغسل الخرزات المربوطة 5x في 10 مل من RSB-500 + 0.1٪ Tx-100. لكل غسلة ، اقلب الأنبوب 2x-5x لإعادة تعليق الخرز ، ثم الحبيبات عند 1000 × جم لمدة 1 دقيقة عند 4 درجات مئوية. أخيرا ، اغسل 1x في RSB-100 (20 mM Tris-HCl pH 7.5 ، 100 mM NaCl ، 25 mM MgCl2) + 0.1٪ Tx-100.
- شطف الببتيد FLAG: تخلص من مركب HAT1 في 1.5 مل من محلول الشطف (EB) الذي يحتوي على 0.5 مجم / مل من ببتيد FLAG. احتضان في 4 درجة مئوية لمدة 1 ساعة على الأقل أو بين عشية وضحاها. نبض الطرد المركزي حبات مستخلصة عند 1000 × غرام لمدة 30 ثانية عند 4 درجات مئوية وجمع المادة الطافية ، والتي تحتوي على مركب HAT1 / Rbap46 المنقى.
- تركيز البروتين وإزالة الببتيد FLAG.
- أضف الشطف إلى أنبوب مرشح قطع دالتون سعة 20 مل ، 10000 لتر. ارفع مستوى الصوت إلى 15 مل باستخدام EB. قم بالدوران عند 2500 × جم لمدة 15 دقيقة عند 4 درجات مئوية ، مع التكرار مرتين مع 15 مل إضافية من EB في كل مرة.
- استرجع الشطف النهائي من أنبوب المرشح وتحقق من تركيز البروتين بواسطة A260. توقع تركيز 1 ملغ من البروتين الكلي في 6 مل من EB (لكل 300 مل من ثقافة البدء). تحقق من نقاء البروتين بواسطة SDS-PAGE و Coomassie و immunoblotting ، وقم بتخزين القسامات عند -80 درجة مئوية.
- تحلل الخلايا وإعداد مستخلص البروتين.
2. الطريقة الثانية: منحنى قياسي HAT1 أسيتيل انقر
- إعداد المنحنى القياسي
- توليف الببتيد الطرفي H4 N-terminal للتحكم الإيجابي مع التسلسل: SCRG [Pra] GGKGLG [Pra] GGAKRHRKVLRGG [Lys (Biotin)] ، حيث يشير [Pra] إلى Propargylglycine.
- أعد تعليق ببتيد Pra إلى 0.1 مجم / مل في DMSO. امزج ببتيد Pra مع ببتيد هيستون H4 البيوتيني (1-23-GGK-biotin) لإنشاء منحنى قياسي (الشكل 2 ؛ الأحجام الموجودة في الجدول 1). يمكن جعل المنحنيات القياسية طازجة قبل ربط الألواح أو ، إذا تم إجراؤها مسبقا ، يتم خلطها ب 20 ميكرولتر من 8 M من اليوريا وتخزينها عند -20 درجة مئوية حتى الخطوة 3.3.
3. الطريقة الثالثة: مقايسة نقرة الأسيتيل HAT1
- تجميع تفاعلات أستلة مع مثبطات الاختبار
- قم بتجميع تفاعلات الأستيل في نسختين في أنابيب PCR سعة 0.2 مل أو ألواح تفاعل البوليميراز المتسلسل ذات 96 بئرا من المكونات التالية: ببتيد هيستون هيستون H4 بيوتي (1-23-GGK-بيوتين) المعاد تعليقه في DMSO إلى 0.1 مجم / مل (34.8 ميكرومتر) ، إنزيم HAT1 المخفف مسبقا في EB ، 20x buffer (1M Tris pH 8.5 ، 0.1٪ NP40) ، 2 mM DTT ، 4-pentynoyl-CoA المذاب في الماء إلى 1 مجم / مل (1 مللي مول). يتكون تفاعل 20 ميكرولتر من 10 ميكرولتر من الإنزيم ، و 1 ميكرولتر من الببتيد H4 ، و 1 ميكرولتر من المخزن المؤقت 20x ، و 1 ميكرولتر من DTT المخلوط مسبقا والموزع إلى آبار صفيحة PCR ذات 96 بئرا على الجليد.
- أضف 1 ميكرولتر من DMSO (التحكم السلبي) أو مركب الاختبار المذاب عند 1-10 ميكرومتر ، أو H4K12CoA25 (أو مثبط التحكم الإيجابي المناسب) ، لكل بئر. تخلط عن طريق ماصة لطيفة واحتضانها لمدة 10 دقائق على الجليد للسماح للإنزيم: مجمعات مثبطة لتكون.
- استمرار تفاعل الأستيل
- الجمع بين 2 ميكرولتر من 4-pentynoyl-CoA مع 4 ميكرولتر من الماء ، ثم تضاف إلى الآبار. امزج برفق عن طريق السحب ، وأجهزة الطرد المركزي عند 300 × جم لمدة 30 ثانية في RT لجمع المحتويات ، واحتضانها عند 37 درجة مئوية لمدة 1 ساعة في أنابيب أو ألواح مغطاة بورق قابل لإعادة الإغلاق.
- قم بمعالجة المحتويات مباشرة لمنتجات التفاعل أو إخمادها ب 20 ميكرولتر من 8 ميجا يوريا وتخزينها في -20 درجة مئوية حتى المعالجة.
- ربط الببتيد بلوحة النيوترفيدين
- أضف محتويات التفاعل مع أو بدون اليوريا إلى ألبومين مصل الأبقار (BSA) المحظور مسبقا الأسود المطلي بالنيوترافيدين 96 صفيحة تحتوي على 80 ميكرولتر من PBST (PBS + 0.1٪ Tween-20) لكل بئر.
- أضف الببتيدات المنحنية القياسية في نسختين إلى آبارها الخاصة التي تحتوي على 80 ميكرولتر من PBST. ربط الببتيدات مع الاهتزاز المداري لطيف لمدة 1 ساعة في درجة حرارة الغرفة (RT).
- غسل: بعد اكتمال الربط ، قم بشفط السائل من الآبار واغسل الآبار ب 200 ميكرولتر من PBST 3x 15 ضربة (حجم شوط 180 ميكرولتر) باستخدام غسالة ألواح.
- انقر فوق تفاعل الكيمياء
- قم بإعداد الكواشف لتفاعل النقر على النحو التالي: 100 mM Tris (3-hydroxypropyltriazolymethyl) أمين (THPTA) يجند في الماء و 20 mM CuSO4 في الماء. يمنع THPTA التحلل المائي المحفز للنحاس (II) ويروي الجذور والبيروكسيدات المتولدة من O2 / Cu / ascorbate.
- أضف خليط THPTA / Cu إلى 300 mM أسكوربات الصوديوم في الماء و 2.5 mM بيوتين أزيد في DMSO. يحتوي التفاعل بنقرة واحدة على 140 ميكرولتر من PBS ، و 10 ميكرولتر من THPTA ، و 10 ميكرولتر من CuSO4 ، و 10 ميكرولتر من أسكوربات الصوديوم ، و 20 ميكرولتر من البيوتين أزيد. امزج دائما كواشف النقر طازجة ، ثم وزع 190 ميكرولتر على كل بئر ، وأغلق اللوحة ، واحتضنها عند 37 درجة مئوية لمدة 1 ساعة.
- اغسل: نضح السائل من الآبار واغسل اللوحة 3x باستخدام PBST (حجم السكتة الدماغية 180 ميكرولتر ، 15 ضربة لكل غسلة).
- ربط بيروكسيديز الستربتافيدين والفجل: يخفف الستربتافيدين HRP (0.224 مجم / مل) 1:10 في الستربتافيدين (0.224 مجم / مل) ، ثم يخفف 1: 1000 في PBST. أضف Steptavidin-HRP: مزيج الستربتافيدين (100 ميكرولتر لكل بئر) واحتضانه في RT لمدة 1 ساعة مع اهتزاز مداري لطيف.
- اغسل 3x باستخدام PBST كما في الخطوات السابقة.
- أكسدة حمراء أمبلكس
- اجمع بين كواشف الكشف الحمراء Amplex على النحو التالي: 4.45 مل من المخزن المؤقت NaHPO4 (1x = 50 mM التركيز النهائي) ، 50 ميكرولتر amplex red (20 mM مخفف في DMSO) ، 500 ميكرولتر من H2O2 المخفف.
- قم بتخفيف H2O2 من 30٪ مخزون إلى 3٪ في 1x NaHPO4 buffer ، ثم أضف 22.7 ميكرولتر من 3٪ H2O2 إلى 977 ميكرولتر من 1x NaHPO4 buffer (هذا هو H2O2 المستخدم في تفاعل amplex). أضف 100 ميكرولتر من خليط تفاعل amplex الأحمر لكل بئر ، واحتضانه في RT لمدة 30 دقيقة محمية من الضوء ، ثم اكتشف إثارة / انبعاث التألق 571/585 نانومتر باستخدام قارئ لوحة مضان قياسي.
- حساب تثبيط الإنزيم: احسب النسبة المئوية للتثبيط (الشكل 3) وفقا للصيغة التالية:
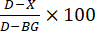 ، حيث D هي القيمة الفلورية لتفاعلات التحكم المعالجة ب DMSO فقط ، X هي قيمة التفاعلات المعالجة بمركبات الاختبار ، و BG هي قيمة الآبار الخلفية (التحكم في الببتيد H4 ، بدون إضافة إنزيم).
، حيث D هي القيمة الفلورية لتفاعلات التحكم المعالجة ب DMSO فقط ، X هي قيمة التفاعلات المعالجة بمركبات الاختبار ، و BG هي قيمة الآبار الخلفية (التحكم في الببتيد H4 ، بدون إضافة إنزيم). - منحنى الجرعة: حدد مركبات الاختبار التي وجد أنها تمنع نشاط إنزيم HAT1 للمقايسات المتكررة ، مع تخفيف المركب بشكل متسلسل. تحديد التخفيفات المركبة تجريبيا. على سبيل المثال ، ابدأ بتركيز مخزون 2 مللي متر ، ثم قم بتخفيف 1: 3 بشكل متسلسل لثمانية تخفيفات. ثم خفف 1:20 إلى مقايسات إنزيمية تنتج جرعة أعلى 100 ميكرومتر.
- رسم المنحنى: استخدم انحدار المربعات الصغرى لتناسب منحنيات تثبيط الجرعة والاستجابة (باستخدام قيم تثبيط النسبة المئوية من الخطوة 3.10) في برنامج تحليل البيانات (GraphPad Prism) واشتقاق قيم IC50 لكل مركب (الشكل 4).
- تفاعل الأستيل مع أسيتيل-CoA:
- استخدم هذا لتأكيد نشاط الإنزيم مع العامل المساعد الأصلي acetyl-CoA بدلا من 4-pentynoyl-CoA. قم بإجراء مقايسات أستلة HAT1 كما هو موضح في الخطوات 3.1-3.2 باستخدام acetyl-CoA بدلا من 4-pentynoyl-CoA.
- اكتشف منتجات التفاعل على أغشية النيتروسليلوز (1-2 ميكرولتر) ، واتركها تجف ، ثم لطخة نقطية بالأجسام المضادة ل H4-lysine-12-acetyl أو الأجسام المضادة ل H4-lysine-5-acetyl. تحديد إشارة اللطخة المناعية عن طريق قياس الكثافة.
النتائج
يجب تضمين المنحنيات القياسية في نسختين (16 بئرا) على كل لوحة لضمان أداء الفحص المناسب. يجب إعداد بيانات المنحنى القياسي في شكل جدول ، مع نطاق من 100٪ إلى 0٪ وفقا لنسبة الببتيد المحتوي على Pra إلى الببتيد H4 الأصلي في المحلول (الجدول 1). ستكون إشارة Amplex الحمراء هي الأعلى في آبار الببتيد H4 ا?...
Discussion
في العقد الماضي ، أصبحت كيمياء النقر بارزة20 ، مما يتيح التصميم الدقيق للهياكل الكيميائية المتفاعلة. في هذا السياق ، ظهرت العديد من الاتصالات التساهمية المتعامدةالحيوية 21 كخيارات واعدة لتشكيل مجمعات في بيئتها الطبيعية. تستخدم كيمياء النقرات أزواجا من المجموعات ...
Disclosures
لقد قدمنا براءات اختراع تصف مقايسة نقرة الأسيتيل HAT1 (PCT / US20/29395). تقارير JG اتفاقيات استشارية مع Sharma Therapeutics، LLC و Guidepoint ودعم البحث (لمؤسسته) من Hummingbird Biosciences.
Acknowledgements
نشكر جورج تشنغ على توفير H4K12CoA. نشكر أعضاء Gruber Lab على المناقشات والتعليقات المفيدة. نشكر الدعم المقدم من المعاهد الوطنية للصحة / NCI (1K08CA245024) و CPRIT (RR200090) ومؤسسة V (V2022-022).
Materials
| Name | Company | Catalog Number | Comments |
| 4P CoA | Cayman Chemical | 10547 | Click chemistry co-factor |
| Amplex Red | Fisher Sci | A12222 | Fluorescence substrate |
| Biotin-PEG-Azide | Alfa Aesar | J64996MC | Click chemistry |
| Copper Sulfate | Sigma-aldrich | 7758-98-7 | Click chemistry |
| DMSO | Fisher Scientific | 67-68-5 | diluent |
| DTT | Acros Organics | 03-12-3483 | reducting agent |
| Forskolin | VWR | 102987-310 | Protein expression |
| Freestyle 293 Expression Medium | Thermo Fisher | 12338018 | Media |
| Freestyle 293-F cells | Thermo Fisher | R790-07 | Protein expression |
| H4-peptide/1-23-GGK-biotin | Anaspec | AS65097 | peptide substrate |
| HEPES | Sigma-aldrich | 7365-45-9 | EB buffer |
| Hydrogen peroxide 30% solution | Sigma-aldrich | Z00183-99-0 | initiator |
| M2 FLAG antibody slurry | Millipore-Sigma | A2220 | Protein purification |
| Macrosep 10K Filter (Pall Lab) | VWR | 89131-980 | Protein purification |
| Neutravidin Plate | Thermo Sci | 15127 | BSA-pre-blocked |
| NP40 (IGEPAL) | MP Biomedical | 198596 | 20x buffer |
| pHEK-293 plasmid | Takara Bio | 3390 | Protein expression |
| Phosphate Buffered Saline 10x | Alfa Aesar | Z00082-33-6 | wash buffer |
| Pra peptide | Genscript | Custom synthesis | biotinylated |
| Sodium Ascorbate | Sigma-aldrich | 134-03-2 | Click chemistry |
| Sodium chloride | Sigma-aldrich | 7647-14-5 | EB buffer |
| Sodium phosphate | VWR International | 7558-80-7 | buffer |
| Streptavidin | EMD Millipore | 189730 | competitor |
| Streptavidin-HRP | Cell Signaling | 3999S | enzyme |
| THPTA ligand | Fisher Sci | 1010-500 | Click chemistry |
| Tris base | Sigma-aldrich | 77-86-1 | 20x buffer |
| Triton-X 100 | VWR International | 9002-93-1 | EB buffer |
| Tween-20 | Sigma-aldrich | 9005-64-5 | Wash buffer |
| Urea | Sigma-Aldrich | 57-13-6 | quencher |
References
- Kleff, S., et al. Identification of a gene encoding a yeast histone H4 acetyltransferase. J Biol Chem. 270 (42), 24674-24677 (1995).
- Parthun, M. R., Widom, J., Gottschling, D. E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism. Cell. 87 (1), 85-94 (1996).
- Verreault, A., et al. Nucleosomal DNA regulates the core-histone-binding subunit of the human Hat1 acetyltransferase. Curr Biol. 8 (2), 96-108 (1998).
- Parthun, M. R. Histone acetyltransferase 1: more than just an enzyme. Biochim Biophys Acta. 1819 (3-4), 256-263 (2013).
- Gruber, J. J., et al. HAT1 coordinates histone production and acetylation via H4 promoter binding. Mol Cell. 75 (4), 711-724 e5 (2019).
- Fan, P., et al. Overexpressed histone acetyltransferase 1 regulates cancer immunity by increasing programmed death-ligand 1 expression in pancreatic cancer. J Exp Clin Cancer Res. 38 (1), 47 (2019).
- Xia, P., et al. MicroRNA-377 exerts a potent suppressive role in osteosarcoma through the involvement of the histone acetyltransferase 1-mediated Wnt axis. J Cell Physiol. 234 (12), 22787-22798 (2019).
- Yang, G., et al. Histone acetyltransferase 1 is a succinyltransferase for histones and non-histones and promotes tumorigenesis. EMBO Rep. 22 (2), e50967 (2021).
- Xue, L., et al. RNAi screening identifies HAT1 as a potential drug target in esophageal squamous cell carcinoma. Int J Clin Exp Pathol. 7 (7), 3898-3907 (2014).
- Zhang, W., et al. Structural plasticity of histones H3-H4 facilitates their allosteric exchange between RbAp48 and ASF1. Nat Struct Mol Biol. 20 (1), 29-35 (2013).
- Campos, E. I., et al. The program for processing newly synthesized histones H3.1 and H4. Nat Struct Mol Biol. 17 (11), 1343-1351 (2010).
- Agudelo Garcia, P. A., et al. Identification of multiple roles for histone acetyltransferase 1 in replication-coupled chromatin assembly. Nucleic Acids Res. 45 (16), 9319-9335 (2017).
- Nagarajan, P., et al. Histone acetyl transferase 1 is essential for mammalian development, genome stability, and the processing of newly synthesized histones H3 and H4. PLoS Genet. 9 (6), e1003518 (2013).
- Annunziato, A. T. Assembling chromatin: the long and winding road. Biochim Biophys Acta. 1819 (3-4), 196-210 (2013).
- Annunziato, A. T., Seale, R. L. Histone deacetylation is required for the maturation of newly replicated chromatin. J Biol Chem. 258 (20), 12675-12684 (1983).
- Dahlin, J. L., et al. Assay interference and off-target liabilities of reported histone acetyltransferase inhibitors. Nat Commun. 8 (1), 1527 (2017).
- Baell, J. B., Miao, W. Histone acetyltransferase inhibitors: where art thou. Future Med Chem. 8 (13), 1525-1528 (2016).
- Lasko, L. M., et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours. Nature. 550 (7674), 128-132 (2017).
- Baell, J. B., et al. Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. Nature. 560 (7717), 253-257 (2018).
- Falk, H., et al. An efficient high-throughput screening method for MYST family acetyltransferases, a new class of epigenetic drug targets. J Biomol Screen. 16 (10), 1196-1205 (2011).
- He, M., et al. Chemical biology approaches for investigating the functions of lysine acetyltransferases. Angew Chem Int Ed Engl. 57 (5), 1162-1184 (2018).
- Lipchik, A. M., et al. A peptide-based biosensor assay to detect intracellular Syk kinase activation and inhibition. Biochemistry. 51 (38), 7515-7524 (2012).
- Song, J., et al. Chemoproteomic profiling of protein substrates of a major lysine acetyltransferase in the native cellular context. ACS Chem Biol. 17 (5), 1092-1102 (2022).
- Gaddameedi, J. D., et al. Acetyl-click screening platform identifies small-molecule inhibitors of histone acetyltransferase 1 (HAT1). J Med Chem. 66 (8), 5774-5801 (2023).
- Ngo, L., Brown, T., Zheng, Y. G. Bisubstrate inhibitors to target histone acetyltransferase 1 (HAT1). Chem Biol Drug Des. 93 (5), 865-873 (2019).
- Parker, C. G., Pratt, M. R. Click chemistry in proteomic investigations. Cell. 180 (4), 605-632 (2020).
- Islam, K. The bump-and-hole tactic: Expanding the scope of chemical genetics. Cell Chem Biol. 25 (10), 1171-1184 (2018).
- Radziwon, K., Weeks, A. M. Protein engineering for selective proteomics. Curr Opin Chem Biol. 60, 10-19 (2021).
- Rich, R. L., Myszka, D. G. Survey of the year 2007 commercial optical biosensor literature. J Mol Recognit. 21 (6), 355-400 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved