Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Un test de chimie acétyl-clic pour mesurer l’acétylation de l’histone acétyltransférase 1
Dans cet article
Résumé
Des tests chimiques rapides et précis pour dépister des inhibiteurs spécifiques sont un outil important dans l’arsenal de développement de médicaments. Ici, nous présentons un test de chimie acétyl-click évolutif pour mesurer l’inhibition de l’activité d’acétylation de HAT1.
Résumé
HAT1, également connue sous le nom d’histone acétyltransférase 1, joue un rôle crucial dans la synthèse de la chromatine en stabilisant et en acétylant le H4 naissant avant l’assemblage des nucléosomes. Il est nécessaire à la croissance tumorale dans divers systèmes, ce qui en fait une cible potentielle pour le traitement du cancer. Pour faciliter l’identification des composés capables d’inhiber l’activité enzymatique de HAT1, nous avons mis au point un test acétyl-click pour un criblage rapide. Dans ce test simple, nous utilisons HAT1/Rbap46 recombinant, qui est purifié à partir de cellules humaines activées. La méthode utilise l’analogue de l’acétyl-CoA 4-pentynoyl-CoA (4P) dans une approche de chimie click. Il s’agit du transfert enzymatique d’une poignée d’alcyne par une réaction d’acylation dépendante de HAT1 à un peptide N-terminal H4 biotinylé. Le peptide capturé est ensuite immobilisé sur des plaques de neutravidines, suivi d’une fonctionnalisation par chimie click avec de l’azoture de biotine. Par la suite, le recrutement de la streptavidine-peroxydase est utilisé pour oxyder le rouge d’amplex, ce qui donne une sortie fluorescente quantitative. En introduisant des inhibiteurs chimiques pendant la réaction d’acylation, nous pouvons quantifier l’inhibition enzymatique en fonction d’une réduction du signal de fluorescence. Il est important de noter que cette réaction est évolutive, ce qui permet un criblage à haut débit d’inhibiteurs potentiels de l’activité enzymatique HAT1.
Introduction
Parmi les nombreuses acétyltransférases eucaryotes, HAT1 était l’histone acétyltransférase initiale à être isolée 1,2,3. Des recherches ultérieures ont fermement établi son rôle central dans la réplication de la chromatine, en particulier dans la synthèse de nouveaux nucléosomes au cours de la phaseS 4. Nos efforts de recherche ont permis de reconnaître que HAT1 est fortement stimulé par le traitement par facteur de croissance épidermique (EGF) dans les cellules mammaires5. De plus, il est apparu que HAT1 est nécessaire à la prolifération cellulaire rapide et à la formation de tumeurs in vivo 6,7,8,9. Les données indiquent que HAT1 est essentiel à la coordination des processus anaboliques et épigénétiques pour la division cellulaire, conduisant la croissance tumorale.
HAT1 di-acétyle la queue amino-terminale de l’histone H4 sur les lysines 5 et 12 en complexe avec la protéine chaperon Rbap46, qui se lie à l’histone et présente l’amino-terminal à HAT1. Les tétramères d’histones ou disomes10, ainsi que HAT1/Rbap46 et d’autres chaperons d’histones11, sont ensuite importés dans le noyau. Les histones sont ensuite libérées pour être déposées à la fourche de réplication ou à d’autres sites pour soutenir l’activation ou la répression des gènes. La fonction de la marque de di-acétylation HAT1 sur l’histone H4 n’est pas entièrement comprise. Il est probable qu’il est rapidement éliminé en l’espace de 15 à 30 minutes par l’action des histones désacétylases 12,13,14,15 après l’insertion de H4 dans la chromatine. Ainsi, la marque de di-acétylation HAT1 n’est pas propagée dans la chromatine et peut ne pas jouer un véritable rôle épigénétique, bien qu’un rôle dans le recrutement d’enzymes modifiant la chromatine dans la chromatine naissante ait été postulé12. De plus, HAT1 n’acétyle pas directement la chromatine ; Son activité est limitée aux histones solubles.
Le développement de petites molécules inhibitrices de l’histone acétyltransférase a été entravé par des essais non spécifiques et à faible débit, ce qui a souvent entraîné la génération de composés biologiquement réactifs16,17. Le test de référence pour mesurer les activités de l’acétyltransférase nécessite l’utilisation de 3H-acétyl-coA, ce qui limite le débit et nécessite des radiations. Néanmoins, récemment, des inhibiteurs spécifiques et très puissants de l’acétyltransférase à petites molécules ciblant CBP/p30018 et KAT6A/B19,20 ont été décrits et confirmés par l’utilisation de 3H-acétyl-CoA. À l’avenir, des tests améliorés pour obtenir un meilleur débit et éviter les dangers en laboratoire sont en cours de conception.
Les progrès récents dans la surveillance de l’acétylation21 ont utilisé la chimie click pour permettre la surveillance des réactions enzymatiques. Il existe une variété de précurseurs à clic qui sont accessibles par de simples voies synthétiques ou disponibles à l’achat et qui peuvent être incorporés dans des réactions enzymatiques. Ces réactions sont généralement effectuées dans des systèmes recombinants, bien que des essais cellulaires soient également réalisables22. L’avantage des cofacteurs et des substrats à clic est que le criblage peut mesurer directement l’activité enzymatique sans avoir besoin de systèmes de lecture couplés qui sont souvent perturbés par les composés de criblage et nécessitent des étapes de manipulation supplémentaires. Cela permet des traitements inhibiteurs uniquement pendant l’étape enzymatique, alors que toutes les étapes de fonctionnalisation et de détection en aval sont effectuées après un lavage approfondi pour éliminer les composés, limitant ainsi le risque d’interférence avec le test. Ces avantages rendent la conception des tests par clic préférable aux tests couplés qui reposent généralement sur la détection de la coenzyme A libre.
Une considération importante est l’acceptation des cofacteurs cliquables dans le site actif de l’enzyme. Les cofacteurs à clic existants peuvent ne pas être entièrement compatibles avec le site actif optimisé pour le cofacteur natif. Les informations structurelles et la modélisation peuvent être utilisées pour concevoir des substitutions d’acides aminés afin d’élargir le site actif pour incorporer des substrats modifiés23. Cela peut permettre un criblage avec une cinétique enzymatique améliorée et des niveaux de substrat et d’enzymes plus faibles. L’inconvénient de cette approche est que les poches catalytiques altérées peuvent ne pas identifier les inhibiteurs qui interagissent fortement avec l’enzyme native. En fin de compte, une combinaison d’approches est nécessaire pour identifier et valider les inhibiteurs enzymatiques potentiels.
Nous décrivons ici une méthode développée pour purifier et doser l’activité enzymatique HAT1 en utilisant le cofacteur clic 4-pentynoyl-CoA24. Ce test (Figure 1) utilise la séquence enzymatique native en complexe avec sa protéine partenaire requise Rbap46, qui s’est avérée stimuler l’activité enzymatique. La purification de l’enzyme des cellules humaines permet l’activation enzymatique dans le cellulo, ce qui peut préserver des modifications post-traductionnelles stimulantes importantes pour une activité enzymatique complète. La conception et l’optimisation de tests d’enzymes recombinantes pour les criblages chimiques à haut débit ont été utilisées avec succès pour identifier et caractériser les inhibiteurs de petites molécules HAT1.
Protocole
1. Méthode 1 : Production et purification du complexe recombinant HAT1/Rbap46
- Décongélation, récupération et expansion des cellules HEK293f
- Décongeler 1 à 10 millions de cellules de mammifères HEK293f26 dans des milieux d’expression libres 293 de 30 mL dans une fiole de 100 mL. Incuber dans 8% de CO2 à 37 °C en tournant à 60 tr/min.
- Le lendemain, comptez et vérifiez la viabilité des cellules, puis ajustez la vitesse de rotation à 120 tr/min. Étendre à 300 mL de culture dans une fiole de 1 L, en maintenant la densité d’ensemencement à 500 000 cellules/mL et en divisant les cellules avant que la densité ne dépasse 3 x 106 cellules/mL.
- Transfection HEK293f
- Semez 5 x 105 cellules/mL dans une culture de 300 mL dans une fiole de 1 L et cultivez pendant 24 h. Le lendemain, comptez les cellules pour vous assurer qu’elles se situent entre 7,5 x 105 et 1,2 x 106 cellules/ml.
- Préparez un mélange de 300 μg de pHEK-FLAG-HAT1 et de 300 μg d’ADN plasmidique pHEK-Rbap46 dans 30 mL de PBS. Ajouter 1,2 mL de polyéthylénimine (PEI) à l’ADN/PBS et mélanger. Incuber pendant 20 min à RT. Ajouter le mélange ADN/PBS/PEI à la culture de 300 mL et incuber à 37 °C pendant 48 h à 120 tr/min.
- Cellules de prélèvement
- Comptez les cellules pour vous assurer qu’elles se situent entre 1,7 x 106 et 2,5 x 106 cellules/ml. À cette densité cellulaire, ajouter de la forskoline à une concentration finale de 12,5 μM pour activer HAT1 et incuber les cellules pendant 30 min, 37 °C, 120 tr/min.
- Granulez les cellules à 300 x g pendant 5 min, lavez une fois avec 30 mL de PBS et congelez-les dans du liquide N2. Conserver à -80 °C jusqu’à la purification des protéines ou procéder directement à la purification.
- Purification complexe HAT1/Rbap46 de billes FLAG
- Lyser les cellules et préparer l’extrait de protéines.
- Préparez le tampon de lyse en ajoutant un comprimé de cocktail d’inhibiteurs de protéase (PIC) à 50 mL de RSB-500 (20 mM de Tris-HCl pH 7,5, 500 mM de NaCl, 25 mM de MgCl2).
- Décongeler la pastille cellulaire de 300 mL de culture sur de la glace et la lyser avec 40 mL de tampon de lyse glacé avec 0,1 % de Triton X-100. Sonicate sur glace, puis essorage à 10 000 x g pendant 10 min à 4°C. Rassemblez tous les surnageants dans un tube de 50 mL sur de la glace, qui est maintenant l’extrait protéique.
- Préparez les perles FLAG.
- Divisez les billes FLAG en quatre tubes coniques de 15 mL en ajoutant 400 μL de boue de billes d’agarose M2-FLAG dans chaque tube contenant 5 mL de glycine à 0,1 M pH 3,5, 0,01 % Triton X-100. Agiter vigoureusement chaque tube pendant 5 s à la main.
- Centrifuger par impulsions pendant 30 s à 1000 x g à 4 °C, aspirer le surnageant, laver deux fois avec 5 mL de RSB-500 + 0,1 % de Triton X-100 (Tx-100 ; sans inhibiteur de protéase) et incuber sur glace.
- Effectuer l’immunoprécipitation FLAG.
- Ajouter 10 ml d’extrait de protéines dans chaque tube conique de 15 ml contenant des billes lavées. Incuber les tubes à 4 °C avec une rotation inversée pendant au moins 90 minutes ou toute la nuit.
- Lavez les perles liées 5 fois dans 10 mL de RSB-500 + 0,1 % Tx-100. Pour chaque lavage, retournez le tube 2x-5x pour remettre les billes en suspension, puis granulez à 1000 x g pendant 1 min à 4 °C. Enfin, laver 1x dans RSB-100 (20 mM Tris-HCl pH 7,5, 100 mM NaCl, 25 mM MgCl2) + 0,1% Tx-100.
- élution du peptide FLAG : Éluer le complexe HAT1 dans 1,5 mL de tampon d’élution (EB) contenant 0,5 mg/mL de peptide FLAG. Incuber à 4 °C pendant au moins 1 h ou toute la nuit. Centrifuger les billes éluées à 1000 x g pendant 30 s à 4 °C et recueillir le surnageant, qui contient le complexe HAT1/Rbap46 purifié.
- Concentrer les protéines et éliminer le peptide FLAG.
- Ajouter l’éluat dans un tube filtrant de 20 mL et 10 000 Dalton. Porter le volume à 15 mL avec EB. Essorer à 2500 x g pendant 15 min à 4 °C, en répétant deux fois avec 15 mL supplémentaires d’EB à chaque fois.
- Récupérez l’éluat final du tube filtrant et vérifiez la concentration en protéines par A260. Attendez-vous à une concentration de 1 mg de protéines totales dans 6 mL d’EB (par 300 mL de culture initiale). Vérifier la pureté des protéines par SDS-PAGE, Coomassie et immunobuvard, et stocker les aliquotes à -80 °C.
- Lyser les cellules et préparer l’extrait de protéines.
2. Méthode 2 : Courbe standard acétyl-clic HAT1
- Préparation de la courbe standard
- Synthétiser un peptide N-terminal H4 témoin positif avec la séquence : SCRG[Pra]GGKGLG[Pra]GGAKRHRKVLRGG[Lys(Biotine)], où [Pra] désigne Propargylglycine.
- Remettre en suspension le peptide Pra à 0,1 mg/mL dans du DMSO. Mélanger le peptide Pra avec le peptide histones H4 biotinylé (1-23-GGK-biotine) pour créer une courbe standard (Figure 2 ; volumes trouvés dans le tableau 1). Les courbes standard peuvent être fraîches avant la fixation de la plaque ou, si elles sont faites à l’avance, mélangées à 20 μL d’urée 8 M et stockées à -20 °C jusqu’à l’étape 3.3.
3. Méthode 3 : test acétyl-clic HAT1
- Assemblage des réactions d’acétylation avec des inhibiteurs de test
- Assembler les réactions d’acétylation en double dans des tubes PCR de 0,2 mL ou des plaques PCR à 96 puits à partir des composants suivants : peptide d’histone H4 biotinylé (1-23-GGK-biotine) remis en suspension dans du DMSO à 0,1 mg/mL (34,8 μM), enzyme HAT1 pré-diluée dans de l’EB, tampon 20x (1M Tris pH 8,5, 0,1% NP40), 2 mM de DTT, 4-pentynoyl-CoA dissous dans l’eau à 1 mg/mL (1 mM). Une réaction de 20 μL comprend 10 μL d’enzyme, 1 μL de peptide H4, 1 μL de tampon 20x, 1 μL de DTT pré-mélangé et aliquote aux puits d’une plaque PCR de 96 puits sur glace.
- Ajouter 1 μL de DMSO (témoin négatif) ou de composé d’essai dissous à 1-10 μM, ou H4K12CoA25 (ou inhibiteur de contrôle positif approprié), par puits. Mélanger par pipetage doux et incuber pendant 10 min sur de la glace pour permettre la formation de complexes enzymes-inhibiteurs.
- Poursuite de la réaction d’acétylation
- Mélanger 2 μL de 4-pentynoyl-CoA avec 4 μL d’eau, puis ajouter dans les puits. Mélanger délicatement par pipetage, centrifuger à 300 x g pendant 30 s à RT pour recueillir le contenu, et incuber à 37 °C pendant 1 h dans des tubes ou des plaques bouchés avec une feuille refermable.
- Traiter le contenu directement pour les produits de réaction ou tremper avec 20 μL d’urée 8 M et stocker à -20 °C jusqu’au traitement.
- Liaison peptidique à la plaque de neutravidine
- Ajouter le contenu réactionnel avec ou sans urée à des plaques noires prébloquées de 96 puits recouvertes de neutravidine contenant 80 μL de PBST (PBS + 0,1 % Tween-20) par puits.
- Ajouter des peptides courbes standard en double dans leurs propres puits contenant 80 μL de PBST. Lier les peptides par une légère agitation orbitale pendant 1 h à température ambiante (RT).
- Lavage : Une fois le liage terminé, aspirer le liquide des puits et laver les puits avec 200 μL de PBST 3x 15 coups (volume de course de 180 μL) à l’aide d’une laveuse à plaques.
- Réaction de chimie click
- Préparez les réactifs pour la réaction click comme suit : 100 mM de ligand Tris(3-hydroxypropyltriazolyméthyl)amine (THPTA) dans l’eau et 20 mM CuSO4 dans l’eau. Le THPTA empêche l’hydrolyse catalysée par le Cu(II) et éteint les radicaux et les peroxydes générés par l’O2/Cu/ascorbate.
- Ajouter le mélange THPTA/Cu à 300 mM d’ascorbate de sodium dans l’eau et à 2,5 mM d’azoture de biotine dans du DMSO. La réaction en un clic contient 140 μL de PBS, 10 μL de THPTA, 10 μL de CuSO4, 10 μL d’ascorbate de sodium, 20 μL de biotine-azoture. Mélangez toujours les réactifs de clic frais, puis distribuez 190 μL dans chaque puits, scellez la plaque et incubez à 37°C pendant 1 h.
- Lavage : Aspirer le liquide des puits et laver la plaque 3 fois avec du PBST (volume de course de 180 μL, 15 coups pour chaque lavage).
- Liaison streptavidine-peroxydase de raifort : Diluer la streptavidine-HRP (0,224 mg/mL) 1:10 dans la streptavidine (0,224 mg/mL), puis diluer davantage 1:1000 dans le PBST. Ajouter Steptavidin-HRP : mélange de streptavidine (100 μL par puits) et incuber à RT pendant 1 h avec une légère agitation orbitale.
- Lavez 3 fois avec PBST comme dans les étapes précédentes.
- Oxydation rouge Amplex
- Combinez les réactifs de détection Amplex red comme suit : 4,45 mL de tampon NaHPO4 (1x = 50 mM de concentration finale), 50 μL de rouge Amplex (20 mM dilué dans du DMSO), 500 μL de H2O2 dilué.
- Diluer H2O2 de 30 % de stock à 3 % dans 1 tampon NaHPO4 , puis ajouter 22,7 μL de 3 % H2O2 dans 977 μL de 1 tampon NaHPO4 (c’est le H2O2 utilisé pour la réaction d’amplex). Ajouter 100 μL du mélange réactionnel Amplex Red par puits, incuber à RT pendant 30 min à l’abri de la lumière, puis détecter l’excitation/émission de fluorescence 571/585 nm à l’aide d’un lecteur de plaques de fluorescence standard.
- Calcul de l’inhibition enzymatique : Calculer le pourcentage d’inhibition (figure 3) selon la formule suivante :
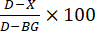 , où D est la valeur de fluorescence des réactions témoins traitées avec du DMSO uniquement, X est la valeur des réactions traitées avec les composés d’essai et BG est la valeur des puits de fond (contrôle peptidique H4, aucune enzyme ajoutée).
, où D est la valeur de fluorescence des réactions témoins traitées avec du DMSO uniquement, X est la valeur des réactions traitées avec les composés d’essai et BG est la valeur des puits de fond (contrôle peptidique H4, aucune enzyme ajoutée). - Courbe de dose : Sélectionner les composés d’essai qui inhibent l’activité enzymatique HAT1 pour des essais répétés, avec le composé dilué en série. Déterminer les dilutions des composés de manière empirique. Par exemple, commencez avec une concentration de stock de 2 mM, puis diluez en série 1:3 pour huit dilutions. Diluer ensuite 1:20 dans des dosages enzymatiques donnant une dose maximale de 100 μM.
- Tracer la courbe : Utiliser la régression des moindres carrés pour ajuster les courbes d’inhibition dose-réponse (en utilisant les valeurs d’inhibition en pourcentage de l’étape 3.10) dans un logiciel d’analyse de données (GraphPad Prism) et dériver les valeurs de la CI50 pour chaque composé (figure 4).
- Réaction d’acétylation avec l’acétyl-CoA :
- Utilisez-le pour confirmer l’activité enzymatique avec le cofacteur natif acétyl-CoA au lieu du 4-pentynoyl-CoA. Effectuer les tests d’acétylation HAT1 comme décrit aux étapes 3.1-3.2 avec de l’acétyl-CoA à la place du 4-pentynoyl-CoA.
- Repérez les produits de réaction sur des membranes de nitrocellulose (1-2 μL), laissez-les sécher, puis épongez avec des anticorps anti-H4-lysine-12-acétyle ou anti-H4-lysine-5-acétyle. Quantifier le signal immunoblot par densitométrie.
Résultats
Des courbes standard en double (16 puits) doivent être incluses sur chaque plaque pour garantir une bonne performance d’analyse. Les données de la courbe standard doivent être présentées sous forme de tableau, avec une fourchette de 100 % à 0 % selon le rapport entre le peptide contenant du pra et le peptide H4 natif en solution (tableau 1). Le signal rouge Amplex sera le plus élevé dans les puits de peptides H4 100 % pra/0 % natif, et le plus bas dans les puits peptidiques H4 0 % pra/100 % nat...
Discussion
Au cours de la dernière décennie, la chimie click a pris de l’importance20, permettant la conception précise de structures chimiques en interaction. Dans ce contexte, diverses connexions covalentes bioorthogonales21 sont apparues comme des options prometteuses pour former des complexes dans leur environnement naturel. La chimie click utilise des paires de groupes fonctionnels qui présentent des réactions rapides et sélectives, communément appelées « réactions cl...
Déclarations de divulgation
Nous avons déposé des brevets décrivant le test HAT1 acétyl-clic (PCT/US20/29395). J.J.G. rapporte des accords de consultation avec Sharma Therapeutics, LLC et Guidepoint et un soutien à la recherche (à son institution) de Hummingbird Biosciences.
Remerciements
Nous remercions George Zheng pour avoir fourni H4K12CoA. Nous remercions les membres du Gruber Lab pour leurs discussions et leurs commentaires utiles. Nous remercions le soutien du NIH/NCI (1K08CA245024), du CPRIT (RR200090) et de la V Foundation (V2022-022).
matériels
| Name | Company | Catalog Number | Comments |
| 4P CoA | Cayman Chemical | 10547 | Click chemistry co-factor |
| Amplex Red | Fisher Sci | A12222 | Fluorescence substrate |
| Biotin-PEG-Azide | Alfa Aesar | J64996MC | Click chemistry |
| Copper Sulfate | Sigma-aldrich | 7758-98-7 | Click chemistry |
| DMSO | Fisher Scientific | 67-68-5 | diluent |
| DTT | Acros Organics | 03-12-3483 | reducting agent |
| Forskolin | VWR | 102987-310 | Protein expression |
| Freestyle 293 Expression Medium | Thermo Fisher | 12338018 | Media |
| Freestyle 293-F cells | Thermo Fisher | R790-07 | Protein expression |
| H4-peptide/1-23-GGK-biotin | Anaspec | AS65097 | peptide substrate |
| HEPES | Sigma-aldrich | 7365-45-9 | EB buffer |
| Hydrogen peroxide 30% solution | Sigma-aldrich | Z00183-99-0 | initiator |
| M2 FLAG antibody slurry | Millipore-Sigma | A2220 | Protein purification |
| Macrosep 10K Filter (Pall Lab) | VWR | 89131-980 | Protein purification |
| Neutravidin Plate | Thermo Sci | 15127 | BSA-pre-blocked |
| NP40 (IGEPAL) | MP Biomedical | 198596 | 20x buffer |
| pHEK-293 plasmid | Takara Bio | 3390 | Protein expression |
| Phosphate Buffered Saline 10x | Alfa Aesar | Z00082-33-6 | wash buffer |
| Pra peptide | Genscript | Custom synthesis | biotinylated |
| Sodium Ascorbate | Sigma-aldrich | 134-03-2 | Click chemistry |
| Sodium chloride | Sigma-aldrich | 7647-14-5 | EB buffer |
| Sodium phosphate | VWR International | 7558-80-7 | buffer |
| Streptavidin | EMD Millipore | 189730 | competitor |
| Streptavidin-HRP | Cell Signaling | 3999S | enzyme |
| THPTA ligand | Fisher Sci | 1010-500 | Click chemistry |
| Tris base | Sigma-aldrich | 77-86-1 | 20x buffer |
| Triton-X 100 | VWR International | 9002-93-1 | EB buffer |
| Tween-20 | Sigma-aldrich | 9005-64-5 | Wash buffer |
| Urea | Sigma-Aldrich | 57-13-6 | quencher |
Références
- Kleff, S., et al. Identification of a gene encoding a yeast histone H4 acetyltransferase. J Biol Chem. 270 (42), 24674-24677 (1995).
- Parthun, M. R., Widom, J., Gottschling, D. E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism. Cell. 87 (1), 85-94 (1996).
- Verreault, A., et al. Nucleosomal DNA regulates the core-histone-binding subunit of the human Hat1 acetyltransferase. Curr Biol. 8 (2), 96-108 (1998).
- Parthun, M. R. Histone acetyltransferase 1: more than just an enzyme. Biochim Biophys Acta. 1819 (3-4), 256-263 (2013).
- Gruber, J. J., et al. HAT1 coordinates histone production and acetylation via H4 promoter binding. Mol Cell. 75 (4), 711-724 e5 (2019).
- Fan, P., et al. Overexpressed histone acetyltransferase 1 regulates cancer immunity by increasing programmed death-ligand 1 expression in pancreatic cancer. J Exp Clin Cancer Res. 38 (1), 47 (2019).
- Xia, P., et al. MicroRNA-377 exerts a potent suppressive role in osteosarcoma through the involvement of the histone acetyltransferase 1-mediated Wnt axis. J Cell Physiol. 234 (12), 22787-22798 (2019).
- Yang, G., et al. Histone acetyltransferase 1 is a succinyltransferase for histones and non-histones and promotes tumorigenesis. EMBO Rep. 22 (2), e50967 (2021).
- Xue, L., et al. RNAi screening identifies HAT1 as a potential drug target in esophageal squamous cell carcinoma. Int J Clin Exp Pathol. 7 (7), 3898-3907 (2014).
- Zhang, W., et al. Structural plasticity of histones H3-H4 facilitates their allosteric exchange between RbAp48 and ASF1. Nat Struct Mol Biol. 20 (1), 29-35 (2013).
- Campos, E. I., et al. The program for processing newly synthesized histones H3.1 and H4. Nat Struct Mol Biol. 17 (11), 1343-1351 (2010).
- Agudelo Garcia, P. A., et al. Identification of multiple roles for histone acetyltransferase 1 in replication-coupled chromatin assembly. Nucleic Acids Res. 45 (16), 9319-9335 (2017).
- Nagarajan, P., et al. Histone acetyl transferase 1 is essential for mammalian development, genome stability, and the processing of newly synthesized histones H3 and H4. PLoS Genet. 9 (6), e1003518 (2013).
- Annunziato, A. T. Assembling chromatin: the long and winding road. Biochim Biophys Acta. 1819 (3-4), 196-210 (2013).
- Annunziato, A. T., Seale, R. L. Histone deacetylation is required for the maturation of newly replicated chromatin. J Biol Chem. 258 (20), 12675-12684 (1983).
- Dahlin, J. L., et al. Assay interference and off-target liabilities of reported histone acetyltransferase inhibitors. Nat Commun. 8 (1), 1527 (2017).
- Baell, J. B., Miao, W. Histone acetyltransferase inhibitors: where art thou. Future Med Chem. 8 (13), 1525-1528 (2016).
- Lasko, L. M., et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours. Nature. 550 (7674), 128-132 (2017).
- Baell, J. B., et al. Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. Nature. 560 (7717), 253-257 (2018).
- Falk, H., et al. An efficient high-throughput screening method for MYST family acetyltransferases, a new class of epigenetic drug targets. J Biomol Screen. 16 (10), 1196-1205 (2011).
- He, M., et al. Chemical biology approaches for investigating the functions of lysine acetyltransferases. Angew Chem Int Ed Engl. 57 (5), 1162-1184 (2018).
- Lipchik, A. M., et al. A peptide-based biosensor assay to detect intracellular Syk kinase activation and inhibition. Biochemistry. 51 (38), 7515-7524 (2012).
- Song, J., et al. Chemoproteomic profiling of protein substrates of a major lysine acetyltransferase in the native cellular context. ACS Chem Biol. 17 (5), 1092-1102 (2022).
- Gaddameedi, J. D., et al. Acetyl-click screening platform identifies small-molecule inhibitors of histone acetyltransferase 1 (HAT1). J Med Chem. 66 (8), 5774-5801 (2023).
- Ngo, L., Brown, T., Zheng, Y. G. Bisubstrate inhibitors to target histone acetyltransferase 1 (HAT1). Chem Biol Drug Des. 93 (5), 865-873 (2019).
- Parker, C. G., Pratt, M. R. Click chemistry in proteomic investigations. Cell. 180 (4), 605-632 (2020).
- Islam, K. The bump-and-hole tactic: Expanding the scope of chemical genetics. Cell Chem Biol. 25 (10), 1171-1184 (2018).
- Radziwon, K., Weeks, A. M. Protein engineering for selective proteomics. Curr Opin Chem Biol. 60, 10-19 (2021).
- Rich, R. L., Myszka, D. G. Survey of the year 2007 commercial optical biosensor literature. J Mol Recognit. 21 (6), 355-400 (2008).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon