Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Ацетил-клик-химический анализ для измерения ацетилирования гистон-ацетилтрансферазы 1
В этой статье
Резюме
Быстрые и точные химические анализы для скрининга специфических ингибиторов являются важным инструментом в арсенале разработки лекарств. Здесь мы представляем масштабируемый химический анализ с ацетиловым щелком для измерения ингибирования активности ацетилирования HAT1.
Аннотация
HAT1, также известный как гистон-ацетилтрансфераза 1, играет решающую роль в синтезе хроматина, стабилизируя и ацетилируя зарождающийся H4 перед сборкой нуклеосом. Он необходим для роста опухолей в различных системах, что делает его потенциальной мишенью для лечения рака. Чтобы облегчить идентификацию соединений, которые могут ингибировать ферментативную активность HAT1, мы разработали ацетил-клик-анализ для быстрого скрининга. В этом простом анализе мы используем рекомбинантный HAT1/Rbap46, который очищается от активированных клеток человека. В методе используется аналог ацетил-КоА 4-пентиноил-КоА (4P) в клик-химическом подходе. Это включает в себя ферментативный перенос алкинового ручки через HAT1-зависимую реакцию ацилирования на биотинилированный N4-концевой пептид. Затем захваченный пептид иммобилизуют на нейтравидиновых планшетах с последующей клик-химическим функционализированием с помощью биотина-азида. Затем рекрутирование стрептавидин-пероксидазы используется для окисления амплексного красного, что приводит к количественному выходу флуоресцентного сигнала. Вводя химические ингибиторы во время реакции ацилирования, мы можем количественно оценить ферментативное ингибирование на основе снижения сигнала флуоресценции. Важно отметить, что эта реакция является масштабируемой, что позволяет проводить высокопроизводительный скрининг потенциальных ингибиторов ферментативной активности HAT1.
Введение
Среди многочисленных эукариотических ацетилтрансфераз HAT1 был исходной гистон-ацетилтрансферазой, которая была выделена 1,2,3. Последующие исследования твердо установили его ключевую роль в репликации хроматина, особенно в синтезе новых нуклеосом во время S-фазы4. Наши исследования привели к признанию того, что HAT1 сильно стимулируется лечением эпидермального фактора роста (EGF) в клетках молочной железы5. Кроме того, выяснилось, что HAT1 необходим для быстрой пролиферации клеток и образования опухолей in vivo 6,7,8,9. Данные указывают на то, что HAT1 имеет решающее значение в координации анаболических и эпигенетических процессов деления клеток, стимулируя рост опухоли.
HAT1 диацетилирует аминоконцевой хвост гистона H4 на лизинах 5 и 12 в комплексе с белком-шапероном Rbap46, который связывает гистон и представляет аминоконцевой конец HAT1. Затем тетрамеры или дисомыгистонов 10 вместе с HAT1/Rbap46 и другими шаперонамигистонов 11 импортируются в ядро. Затем гистоны высвобождаются, чтобы отложиться в репликационной вилке или других местах для поддержки активации или репрессии генов. Функция метки диацетилирования HAT1 на гистон H4 до конца не изучена. Вероятно, он быстро удаляется в течение 15-30 мин под действием гистондеацетилаз 12,13,14,15 после вставки H4 в хроматин. Таким образом, метка диацетилирования HAT1 не размножается в хроматине и может не играть истинной эпигенетической роли, хотя постулируется роль в рекрутировании хроматин-модифицирующих ферментов в зарождающийся хроматин12. Кроме того, HAT1 не ацетилирует хроматин напрямую; Его активность ограничена растворимыми гистонами.
Разработка низкомолекулярных ингибиторов гистон-ацетилтрансферазы затрудняется неспецифическими и малопроизводительными анализами, часто приводящими к образованию биологически реакционноспособных соединений16,17. Золотой стандарт анализа для измерения активности ацетилтрансферазы требует использования 3H-ацетил-коА, что ограничивает пропускную способность и требует облучения. Тем не менее, в последнее время были описаны и подтверждены специфические и высокоэффективные низкомолекулярные ингибиторы ацетилтрансферазы, нацеленные на CBP/p30018 и KAT6A/B19,20 с использованием 3-H-ацетил-КоА. В дальнейшем разрабатываются более совершенные анализы для достижения большей пропускной способности и предотвращения лабораторных опасностей.
Недавние достижения в области мониторинга ацетилирования21 позволили использовать клик-химию для обеспечения мониторинга ферментативных реакций. Существует множество прекурсоров с поддержкой щелчков, которые доступны простыми синтетическими путями или доступны для покупки, которые могут быть включены в ферментативные реакции. Эти реакции, как правило, проводятся в рекомбинантных системах, хотя клеточные анализы также возможны22. Преимущество кофакторов и субстратов с поддержкой щелчков заключается в том, что скрининг может напрямую измерять активность ферментов без необходимости использования связанных систем считывания, которые часто нарушаются просеивающими соединениями и требуют дополнительных действий по обработке. Это позволяет проводить обработку ингибиторами только на ферментативной стадии, в то время как все последующие этапы функционализации и детектирования выполняются после обширной промывки для удаления соединений, что ограничивает возможность возникновения интерференции при анализе. Эти преимущества делают разработку анализов с поддержкой щелчков предпочтительнее связанных анализов, которые обычно полагаются на обнаружение свободного коэнзима А.
Одним из важных соображений является принятие кофакторов, включенных щелчками, в активный центр фермента. Существующие кофакторы с поддержкой кликов могут быть не полностью совместимы с активным сайтом, оптимизированным для собственного кофактора. Структурная информация и моделирование могут быть использованы для разработки аминокислотных замен для увеличения активного центра для включения измененных субстратов23. Это может позволить проводить скрининг с улучшенной кинетикой ферментов и более низкими уровнями субстратов и ферментов. Недостаток этого подхода заключается в том, что измененные каталитические карманы могут не идентифицировать ингибиторы, которые сильно взаимодействуют с нативным ферментом. В конечном счете, для выявления и валидации потенциальных ингибиторов ферментов требуется комбинация подходов.
В данной статье мы опишем метод, разработанный для очистки и анализа активности фермента HAT1 с использованием клик-кофактора 4-пентиноил-КоА24. В этом анализе (рис. 1) последовательность нативного фермента используется в комплексе с его необходимым партнерским белком Rbap46, который, как было показано, повышает активность фермента. Очистка фермента из клеток человека позволяет активировать фермент в целлюло, что может сохранить стимулирующие посттрансляционные модификации, важные для полной активности фермента. Разработка и оптимизация анализов рекомбинантных ферментов для высокопроизводительных химических скринингов были успешно использованы для идентификации и характеристики низкомолекулярных ингибиторов HAT1.
протокол
1. Способ 1: Получение и очистка рекомбинантного комплекса HAT1/Rbap46
- Размораживание, восстановление и расширение клеток HEK293f
- Разморозьте 1-10 миллионов клеток млекопитающих HEK293f26 в 30 мл среды для экспрессии Freestyle 293 в колбе объемом 100 мл. Инкубировать в 8%СО2 при 37 °C при вращении при 60 об/мин.
- На следующий день пересчитайте и проверьте жизнеспособность ячейки, затем отрегулируйте скорость вращения до 120 об/мин. Расширьте до 300 мл культуры в колбе объемом 1 л, поддерживая плотность посева на уровне 500 000 клеток/мл и расщепляя клетки до того, как плотность превысит 3 x 106 клеток/мл.
- Трансфекция HEK293f
- Высевают 5 x 105 клеток/мл в культуре 300 мл в колбе объемом 1 л и культивируют в течение 24 ч. На следующий день подсчитайте количество клеток, чтобы убедиться, что они находятся в диапазоне от 7,5 x 105 до 1,2 x 106 клеток/мл.
- Приготовьте смесь из 300 мкг pHEK-FLAG-HAT1 и 300 мкг плазмидных ДНК pHEK-Rbap46 в 30 мл PBS. Добавьте 1,2 мл полиэтиленимина (PEI) к ДНК/PBS и перемешайте. Инкубируйте в течение 20 мин при RT. Добавьте смесь ДНК/PBS/PEI в культуру объемом 300 мл и инкубируйте при 37 °C в течение 48 ч при 120 об/мин.
- Заготовка клеток
- Подсчитайте количество клеток, чтобы убедиться, что они находятся в диапазоне от 1,7 x 106 до 2,5 x 106 клеток/мл. При такой плотности клеток добавляют форсколин до конечной концентрации 12,5 мкМ для активации HAT1 и инкубируют клетки в течение 30 мин, 37 °C, 120 об/мин.
- Гранулируйте клетки при концентрации 300 x g в течение 5 минут, промойте один раз 30 мл PBS и мгновенно заморозьте в жидкости N2. Хранить при температуре -80 °C до очистки белка или приступать непосредственно к очистке.
- HAT1/Rbap46 комплекс очистки ФЛАГ-шарик
- Лизируйте клетки и приготовьте белковый экстракт.
- Приготовьте буфер для лизиса, добавив одну таблетку коктейля ингибитора протеазы (PIC) к 50 мл RSB-500 (20 мМ Tris-HCl pH 7,5, 500 мМ NaCl, 25 мМ MgCl2).
- Разморозьте клеточную гранулу из культуры объемом 300 мл на льду и лизируйте 40 мл ледяного лизисного буфера с 0,1% Triton X-100. Нанесите ультразвук на лед, затем отжмите при 10 000 x g в течение 10 минут при 4°C. Соберите всю надосадочную жидкость в одну пробирку объемом 50 мл на льду, которая теперь является белковым экстрактом.
- Подготовьте бусины ФЛАЖОК.
- Разделите шарики FLAG на четыре конические пробирки по 15 мл, добавив 400 мкл суспензии агарозных шариков M2-FLAG в каждую пробирку, содержащую 5 мл 0,1 М глицина pH 3,5, 0,01% Triton X-100. Энергично встряхивайте каждую пробирку в течение 5 секунд вручную.
- Импульсную центрифугу в течение 30 с при 1000 x g при 4 °C, отсасывают надосадочную жидкость, дважды промывают 5 мл RSB-500 + 0,1% Triton X-100 (Tx-100; без ингибитора протеазы) и инкубируют на льду.
- Проведите иммунопреципитацию FLAG.
- Добавьте 10 мл белкового экстракта в каждую коническую пробирку объемом 15 мл, содержащую промытые шарики. Инкубируйте пробирки при температуре 4 °C при перевернутом вращении не менее 90 мин или в течение ночи.
- Промыть связанные бусины 5х раз в 10 мл RSB-500 + 0,1% Tx-100. При каждой стирке переворачивайте тюбик 2-5 раз, чтобы снова взвесить шарики, затем гранулируйте при 1000 x g в течение 1 минуты при 4 °C. Наконец, промыть 1x в RSB-100 (20 мМ Tris-HCl pH 7,5, 100 мМ NaCl, 25 мМ MgCl2) + 0,1% Tx-100.
- Элюирование пептида FLAG: Элюируют комплекс HAT1 в 1,5 мл элюирующего буфера (EB), содержащего 0,5 мг/мл пептида FLAG. Инкубировать при 4 °C не менее 1 ч или в течение ночи. Импульсную центрифугу элюируют гранулы при 1000 x g в течение 30 с при 4 °C и собирают надосадочную жидкость, содержащую очищенный комплекс HAT1/Rbap46.
- Сконцентрируйте белок и удалите пептид FLAG.
- Добавьте элюат в отсечную фильтрующую трубку объемом 20 мл 10 000 Дальтон. Доведите объем до 15 мл с помощью EB. Отжим при 2500 x g в течение 15 минут при 4 °C, повторяя дважды с добавлением 15 мл EB каждый раз.
- Извлеките окончательный элюат из фильтрующей трубки и проверьте концентрацию белка с помощью A260. Следует ожидать концентрации 1 мг общего белка в 6 мл БЭ (на 300 мл исходной культуры). Проверяйте чистоту белка с помощью SDS-PAGE, Coomassie и иммуноблоттинга и храните аликвоты при -80 °C.
- Лизируйте клетки и приготовьте белковый экстракт.
2. Способ 2: стандартная кривая HAT1 с ацетиловым щелчком
- Подготовка стандартной кривой
- Синтезируют положительный контрольный N4-концевой пептид с последовательностью: SCRG[Pra]GGKGLG[Pra]GGAKRHRKVLRGG[Lys(Biotin)], где [Pra] обозначает пропаргилглицин.
- Ресуспендируйте пептид Пра до 0,1 мг/мл в ДМСО. Смешайте пептид Pra с биотинилированным пептидом гистонов H4 (1-23-GGK-биотин) для создания стандартной кривой (рис. 2; объемы см. в таблице 1). Стандартные кривые могут быть сделаны свежими перед обвязкой пластин или, если они сделаны заранее, смешаны с 20 мкл 8 М мочевины и хранятся при -20 °C до этапа 3.3.
3. Метод 3: HAT1 ацетил-кликовый анализ
- Сборка реакций ацетилирования с тест-ингибиторами
- Собирают реакции ацетилирования в двух экземплярах в 0,2 мл ПЦР-пробирок или 96-луночных ПЦР-планшетах из следующих компонентов: биотинилированный пептид гистонов H4 (1-23-GGK-биотин), ресуспендированный в ДМСО до 0,1 мг/мл (34,8 мкМ), фермент HAT1, предварительно разбавленный в EB, 20-кратный буфер (1M Tris pH 8,5, 0,1% NP40), 2 мМ DTT, 4-пентиноил-КоА, растворенный в воде до 1 мг/мл (1 мМ). Реакция объемом 20 мкл состоит из 10 мкл фермента, 1 мкл пептида H4, 1 мкл 20-кратного буфера, 1 мкл ДТТ, предварительно смешанного и аликвотированного в лунки 96-луночного ПЦР-планшета на льду.
- Добавьте 1 мкл ДМСО (отрицательный контроль) или тестовое соединение, растворенное в концентрации 1-10 мкМ, или H4K12CoA25 (или подходящий ингибитор положительного контроля) на лунку. Перемешайте с помощью осторожного пипетирования и инкубируйте в течение 10 минут на льду, чтобы образовались комплексы ферментов: ингибиторов.
- Продолжение реакции ацетилирования
- Смешайте 2 мкл 4-пентиноил-КоА с 4 мкл воды, затем добавьте в лунки. Осторожно перемешайте с помощью пипетирования, центрифугируйте при 300 x g в течение 30 с при RT для сбора содержимого и инкубируйте при 37 °C в течение 1 ч в закрытых пробирках или планшетах с закрывающейся фольгой.
- Обработайте содержимое непосредственно продуктами реакции или закалите 20 мкл мочевины 8 М и храните при -20 °C до переработки.
- Связывание пептида с нейтравидиновой пластиной
- Добавляют реакционное содержимое с мочевиной или без нее в предварительно заблокированный черный нейтравидин с 96-луночным покрытием бычий сывороточный альбумин (BSA), содержащий 80 мкл PBST (PBS + 0,1% Tween-20) на лунку.
- Добавляют стандартные кривые пептиды в двух экземплярах в их собственные лунки, содержащие 80 мкл PBST. Связывают пептиды легким орбитальным встряхиванием в течение 1 ч при комнатной температуре (RT).
- Промывка: После завершения связывания отсосите жидкость из лунок и промойте лунки 200 мкл PBST 3 x 15 ходов (объем хода 180 мкл) с помощью пластинчатой шайбы.
- Реакция клик-химии
- Готовят реактивы для клик-реакции следующим образом: 100 мМ лиганда трис(3-гидроксипропилтриазолиметил)амина (THPTA) в воде и 20 мМCuSO4 в воде. THPTA предотвращает катализируемый Cu(II) гидролиз и гасит радикалы и пероксиды, образующиеся из O2 / Cu / аскорбата.
- Добавьте смесь THPTA/Cu к 300 мМ аскорбата натрия в воде и 2,5 мМ азида биотина в ДМСО. Реакция в один клик содержит 140 мкл PBS, 10 мкл THPTA, 10 мклCuSO4, 10 мкл аскорбата натрия, 20 мкл биотин-азида. Всегда смешивайте свежие реагенты click, затем дозируйте по 190 мкл в каждую лунку, закройте планшет и инкубируйте при 37°C в течение 1 ч.
- Промывка: Аспирируйте жидкость из лунок и промойте планшет 3 раза PBST (объем хода 180 мкл, 15 ходов для каждой промывки).
- Связывание стрептавидина с пероксидазой хрена: разбавить стрептавидин-HRP (0,224 мг/мл) 1:10 в стрептавидине (0,224 мг/мл), затем дополнительно разбавить 1:1000 в PBST. Добавляют Стептавидин-HRP: смесь стрептавидина (100 мкл на лунку) и инкубируют при РТ в течение 1 ч при легком орбитальном встряхивании.
- Вымойте 3 раза PBST, как и на предыдущих шагах.
- Амплекс красное окисление
- Комбинируйте реагенты для обнаружения красного цвета Амплекс следующим образом: 4,45 мл буфера NaHPO4 (1x = конечная концентрация 50 мМ), 50 мкл красного амплекса (20 мМ, разбавленного в ДМСО), 500 мкл разбавленного H2O2.
- РазбавьтеH2O2 с 30% до 3% в 1x буфере NaHPO4 , затем добавьте 22,7 мкл 3%H2O2 в 977 мкл 1x буфера NaHPO4 (этоH2O2 , используемый для амплексной реакции). Добавьте 100 мкл реакционной смеси амплексного красного цвета на лунку, инкубируйте при RT в течение 30 мин в защищенном от света месте, затем детектируйте возбуждение/излучение флуоресценции 571/585 нм с помощью стандартного флуоресцентного планшетного считывателя.
- Расчет ингибирования ферментов: Рассчитайте процент ингибирования (рис. 3) по следующей формуле:
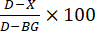 , где D — значение флуоресценции контрольных реакций, обработанных только ДМСО, X — значение реакций, обработанных исследуемыми соединениями, а BG — значение фоновых лунок (контроль пептида H4 без добавления фермента).
, где D — значение флуоресценции контрольных реакций, обработанных только ДМСО, X — значение реакций, обработанных исследуемыми соединениями, а BG — значение фоновых лунок (контроль пептида H4 без добавления фермента). - Кривая дозы: Выберите исследуемые соединения, которые, как установлено, ингибируют активность фермента HAT1 для повторных анализов, с последовательным разбавлением соединения. Определите разведения соединений опытным путем. Например, начните с концентрации 2 мМ, затем последовательно разбавляйте 1:3 в течение восьми разведений. Затем разбавляют 1:20 в ферментных анализах, получая верхнюю дозу 100 мкМ.
- Построение кривой: Используйте регрессию наименьших квадратов для аппроксимации кривых ингибирования «доза-реакция» (используя процентные значения ингибирования из шага 3.10) в программном обеспечении для анализа данных (GraphPad Prism) и получите значения IC50 для каждого соединения (рис. 4).
- Реакция ацетилирования с ацетил-КоА:
- Используйте это для подтверждения активности фермента с нативным кофактором ацетил-КоА вместо 4-пентиноил-КоА. Проведите анализ ацетилирования HAT1, как описано в шагах 3.1-3.2, с ацетил-КоА вместо 4-пентиноил-КоА.
- Нанесите продукты реакции на нитроцеллюлозные мембраны (1-2 мкл), дайте им высохнуть, затем промокните анти-H4-лизин-12-ацетилом или анти-H4-лизин-5-ацетильными антителами. Количественная оценка сигнала иммуноблоттинга методом денситометрии.
Результаты
Стандартные кривые в двух экземплярах (16 лунок) должны быть включены в каждую пластину, чтобы обеспечить надлежащее выполнение анализа. Стандартные данные кривой должны быть представлены в табличной форме с диапазоном от 100% до 0% в зависимости от соотношения Pra-содержащего пептида к нат...
Обсуждение
В последнее десятилетие клик-химиястала широко распространенной, позволив точно проектировать взаимодействующие химические структуры. В этом контексте различные биоортогональные ковалентные связи21 стали перспективными вариантами формирования комплексов...
Раскрытие информации
Мы подали заявки на патенты, описывающие ацетил-клик-анализ HAT1 (PCT/US20/29395). J.J.G. сообщает о консультационных соглашениях с Sharma Therapeutics, LLC и Guidepoint, а также о поддержке исследований (для своего учреждения) со стороны Hummingbird Biosciences.
Благодарности
Мы благодарим Джорджа Чжэна (George Zheng) за предоставление H4K12CoA. Мы благодарим сотрудников Gruber Lab за полезные дискуссии и отзывы. Мы благодарим за поддержку NIH/NCI (1K08CA245024), CPRIT (RR200090) и V Foundation (V2022-022).
Материалы
| Name | Company | Catalog Number | Comments |
| 4P CoA | Cayman Chemical | 10547 | Click chemistry co-factor |
| Amplex Red | Fisher Sci | A12222 | Fluorescence substrate |
| Biotin-PEG-Azide | Alfa Aesar | J64996MC | Click chemistry |
| Copper Sulfate | Sigma-aldrich | 7758-98-7 | Click chemistry |
| DMSO | Fisher Scientific | 67-68-5 | diluent |
| DTT | Acros Organics | 03-12-3483 | reducting agent |
| Forskolin | VWR | 102987-310 | Protein expression |
| Freestyle 293 Expression Medium | Thermo Fisher | 12338018 | Media |
| Freestyle 293-F cells | Thermo Fisher | R790-07 | Protein expression |
| H4-peptide/1-23-GGK-biotin | Anaspec | AS65097 | peptide substrate |
| HEPES | Sigma-aldrich | 7365-45-9 | EB buffer |
| Hydrogen peroxide 30% solution | Sigma-aldrich | Z00183-99-0 | initiator |
| M2 FLAG antibody slurry | Millipore-Sigma | A2220 | Protein purification |
| Macrosep 10K Filter (Pall Lab) | VWR | 89131-980 | Protein purification |
| Neutravidin Plate | Thermo Sci | 15127 | BSA-pre-blocked |
| NP40 (IGEPAL) | MP Biomedical | 198596 | 20x buffer |
| pHEK-293 plasmid | Takara Bio | 3390 | Protein expression |
| Phosphate Buffered Saline 10x | Alfa Aesar | Z00082-33-6 | wash buffer |
| Pra peptide | Genscript | Custom synthesis | biotinylated |
| Sodium Ascorbate | Sigma-aldrich | 134-03-2 | Click chemistry |
| Sodium chloride | Sigma-aldrich | 7647-14-5 | EB buffer |
| Sodium phosphate | VWR International | 7558-80-7 | buffer |
| Streptavidin | EMD Millipore | 189730 | competitor |
| Streptavidin-HRP | Cell Signaling | 3999S | enzyme |
| THPTA ligand | Fisher Sci | 1010-500 | Click chemistry |
| Tris base | Sigma-aldrich | 77-86-1 | 20x buffer |
| Triton-X 100 | VWR International | 9002-93-1 | EB buffer |
| Tween-20 | Sigma-aldrich | 9005-64-5 | Wash buffer |
| Urea | Sigma-Aldrich | 57-13-6 | quencher |
Ссылки
- Kleff, S., et al. Identification of a gene encoding a yeast histone H4 acetyltransferase. J Biol Chem. 270 (42), 24674-24677 (1995).
- Parthun, M. R., Widom, J., Gottschling, D. E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism. Cell. 87 (1), 85-94 (1996).
- Verreault, A., et al. Nucleosomal DNA regulates the core-histone-binding subunit of the human Hat1 acetyltransferase. Curr Biol. 8 (2), 96-108 (1998).
- Parthun, M. R. Histone acetyltransferase 1: more than just an enzyme. Biochim Biophys Acta. 1819 (3-4), 256-263 (2013).
- Gruber, J. J., et al. HAT1 coordinates histone production and acetylation via H4 promoter binding. Mol Cell. 75 (4), 711-724 e5 (2019).
- Fan, P., et al. Overexpressed histone acetyltransferase 1 regulates cancer immunity by increasing programmed death-ligand 1 expression in pancreatic cancer. J Exp Clin Cancer Res. 38 (1), 47 (2019).
- Xia, P., et al. MicroRNA-377 exerts a potent suppressive role in osteosarcoma through the involvement of the histone acetyltransferase 1-mediated Wnt axis. J Cell Physiol. 234 (12), 22787-22798 (2019).
- Yang, G., et al. Histone acetyltransferase 1 is a succinyltransferase for histones and non-histones and promotes tumorigenesis. EMBO Rep. 22 (2), e50967 (2021).
- Xue, L., et al. RNAi screening identifies HAT1 as a potential drug target in esophageal squamous cell carcinoma. Int J Clin Exp Pathol. 7 (7), 3898-3907 (2014).
- Zhang, W., et al. Structural plasticity of histones H3-H4 facilitates their allosteric exchange between RbAp48 and ASF1. Nat Struct Mol Biol. 20 (1), 29-35 (2013).
- Campos, E. I., et al. The program for processing newly synthesized histones H3.1 and H4. Nat Struct Mol Biol. 17 (11), 1343-1351 (2010).
- Agudelo Garcia, P. A., et al. Identification of multiple roles for histone acetyltransferase 1 in replication-coupled chromatin assembly. Nucleic Acids Res. 45 (16), 9319-9335 (2017).
- Nagarajan, P., et al. Histone acetyl transferase 1 is essential for mammalian development, genome stability, and the processing of newly synthesized histones H3 and H4. PLoS Genet. 9 (6), e1003518 (2013).
- Annunziato, A. T. Assembling chromatin: the long and winding road. Biochim Biophys Acta. 1819 (3-4), 196-210 (2013).
- Annunziato, A. T., Seale, R. L. Histone deacetylation is required for the maturation of newly replicated chromatin. J Biol Chem. 258 (20), 12675-12684 (1983).
- Dahlin, J. L., et al. Assay interference and off-target liabilities of reported histone acetyltransferase inhibitors. Nat Commun. 8 (1), 1527 (2017).
- Baell, J. B., Miao, W. Histone acetyltransferase inhibitors: where art thou. Future Med Chem. 8 (13), 1525-1528 (2016).
- Lasko, L. M., et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours. Nature. 550 (7674), 128-132 (2017).
- Baell, J. B., et al. Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. Nature. 560 (7717), 253-257 (2018).
- Falk, H., et al. An efficient high-throughput screening method for MYST family acetyltransferases, a new class of epigenetic drug targets. J Biomol Screen. 16 (10), 1196-1205 (2011).
- He, M., et al. Chemical biology approaches for investigating the functions of lysine acetyltransferases. Angew Chem Int Ed Engl. 57 (5), 1162-1184 (2018).
- Lipchik, A. M., et al. A peptide-based biosensor assay to detect intracellular Syk kinase activation and inhibition. Biochemistry. 51 (38), 7515-7524 (2012).
- Song, J., et al. Chemoproteomic profiling of protein substrates of a major lysine acetyltransferase in the native cellular context. ACS Chem Biol. 17 (5), 1092-1102 (2022).
- Gaddameedi, J. D., et al. Acetyl-click screening platform identifies small-molecule inhibitors of histone acetyltransferase 1 (HAT1). J Med Chem. 66 (8), 5774-5801 (2023).
- Ngo, L., Brown, T., Zheng, Y. G. Bisubstrate inhibitors to target histone acetyltransferase 1 (HAT1). Chem Biol Drug Des. 93 (5), 865-873 (2019).
- Parker, C. G., Pratt, M. R. Click chemistry in proteomic investigations. Cell. 180 (4), 605-632 (2020).
- Islam, K. The bump-and-hole tactic: Expanding the scope of chemical genetics. Cell Chem Biol. 25 (10), 1171-1184 (2018).
- Radziwon, K., Weeks, A. M. Protein engineering for selective proteomics. Curr Opin Chem Biol. 60, 10-19 (2021).
- Rich, R. L., Myszka, D. G. Survey of the year 2007 commercial optical biosensor literature. J Mol Recognit. 21 (6), 355-400 (2008).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены