需要订阅 JoVE 才能查看此. 登录或开始免费试用。
Method Article
用于测量组蛋白乙酰转移酶 1 乙酰化的乙酰基点击化学测定法
摘要
快速准确的化学检测以筛选特定抑制剂是药物开发武器库中的重要工具。在这里,我们提出了一种可扩展的乙酰点击化学测定法来测量HAT1乙酰化活性的抑制。
摘要
HAT1,也称为组蛋白乙酰转移酶 1,通过在核小体组装前稳定和乙酰化新生 H4,在染色质合成中起着至关重要的作用。它是各种系统中肿瘤生长所必需的,使其成为癌症治疗的潜在靶点。为了便于鉴定可抑制HAT1酶活性的化合物,我们设计了一种用于快速筛选的乙酰点击测定法。在这个简单的测定中,我们采用重组HAT1 / Rbap46,它是从活化的人细胞中纯化的。该方法以点击化学方法利用乙酰辅酶A类似物4-戊酰辅酶A(4P)。这涉及通过 HAT1 依赖性酰化反应将炔烃手柄酶促转移到生物素化的 H4 N 末端肽上。然后将捕获的肽固定在中性亲和素平板上,然后用生物素叠氮化物进行点击化学官能团化。随后,采用链霉亲和素-过氧化物酶募集来氧化 amplex 红,从而产生定量荧光输出。通过在酰化反应过程中引入化学抑制剂,我们可以根据荧光信号的还原来量化酶抑制。重要的是,该反应具有可扩展性,可以高通量筛选HAT1酶活性的潜在抑制剂。
引言
在众多真核乙酰转移酶中,HAT1 是最初分离的组蛋白乙酰转移酶 1,2,3。随后的研究已经牢固地确立了它在染色质复制中的关键作用,特别是在 S 期4 期间合成新核小体中。我们的研究努力使人们认识到 HAT1 在乳腺细胞中受到表皮生长因子 (EGF) 处理的高度刺激5。此外,已经发现 HAT1 是体内快速细胞增殖和肿瘤形成所必需的 6,7,8,9。数据表明,HAT1在协调细胞分裂的合成代谢和表观遗传过程,推动肿瘤生长方面至关重要。
HAT1 与伴侣蛋白 Rbap46 复合物将赖氨酸 5 和 12 上组蛋白 H4 的氨基末端尾部二乙酰化,后者结合组蛋白并将氨基末端呈递给 HAT1。然后将组蛋白四聚体或二聚体10与HAT1 / Rbap46和其他组蛋白伴侣11一起导入细胞核。然后组蛋白被释放以沉积在复制叉或其他位点以支持基因激活或抑制。HAT1 二乙酰化标记对组蛋白 H4 的功能尚不完全清楚。在将H4插入染色质后,它可能在15-30分钟内通过组蛋白脱乙酰酶12,13,14,15的作用迅速去除。因此,HAT1 二乙酰化标记不会在染色质中传播,并且可能不具有真正的表观遗传作用,尽管已经假设在染色质修饰酶向新生染色质募集中发挥作用12。此外,HAT1 不直接乙酰化染色质;其活性仅限于可溶性组蛋白。
小分子组蛋白乙酰转移酶抑制剂的开发受到非特异性和低通量测定的阻碍,通常会导致生物反应性化合物的产生16,17。测量乙酰转移酶活性的金标准测定需要使用 3H-乙酰辅酶 A,这限制了通量并需要辐射。尽管如此,最近,通过使用 3H-乙酰辅酶 A 描述并证实了靶向 CBP/p30018 和 KAT6A/B 19,20 的特异性和高效小分子乙酰转移酶抑制剂。展望未来,正在设计改进的检测方法,以实现更好的通量并避免实验室危害。
乙酰化监测21 的最新进展已使用点击化学来实现酶促反应监测。有多种支持点击的前体,可以通过简单的合成途径获得,也可以购买可以掺入酶反应中。这些反应通常在重组系统中进行,尽管基于细胞的测定也是可行的22。支持点击的辅助因子和底物的优点是,筛选可以直接测量酶活性,而无需偶联读出系统,而耦合读出系统通常会受到筛选化合物的干扰,并且需要额外的处理步骤。这允许仅在酶促步骤中进行抑制剂处理,而所有下游功能化和检测步骤均在大量洗涤后进行,以去除化合物,从而限制了发生检测干扰的可能性。这些优点使得支持点击的检测设计比通常依赖于游离辅酶 A 检测的偶联检测更可取。
一个重要的考虑因素是接受启用点击的辅助因子进入酶活性位点。现有的启用点击的辅助因子可能与针对原生辅因子优化的活动站点不完全兼容。结构信息和建模可用于设计氨基酸取代,以扩大活性位点以掺入改变的底物23。这可以通过改进的酶动力学和更低的底物和酶水平进行筛选。这种方法的缺点是改变的催化口袋可能无法识别与天然酶强烈相互作用的抑制剂。最终,需要结合多种方法来识别和验证潜在的酶抑制剂。
在这里,我们描述了一种使用点击辅因子 4-戊酰辅酶A 24 纯化和测定 HAT1 酶活性的方法。该测定(图1)使用天然酶序列与其所需的伴侣蛋白Rbap46复合物,Rbap46已被证明可以提高酶活性。从人体细胞中纯化酶允许 纤维素中的酶活化,这可以保留对完全酶活性很重要的刺激性翻译后修饰。用于高通量化学筛选的重组酶检测的设计和优化已成功用于鉴定和表征 HAT1 小分子抑制剂。
研究方案
1. 方法一:生产纯化重组HAT1/Rbap46复合物
- 解冻、回收和扩增 HEK293f 细胞
- 在 100 mL 烧瓶中将 1-1000 万个 HEK293f 哺乳动物细胞 26 解冻到 30 mL freestyle 293 表达培养基中。在37°C下在8%CO2 中孵育,同时以60rpm旋转。
- 第二天,计数并检查细胞活力,然后将转速调整为 120 rpm。在 1 L 烧瓶中扩增至 300 mL 培养物,将接种密度保持在 500,000 个细胞/mL,并在密度超过 3 x 106 个细胞/mL 之前分裂细胞。
- HEK293f 转染
- 将 5 x 105 个细胞/mL 接种在 300 mL 培养物中,放入 1 L 烧瓶中并培养 24 小时。第二天,对细胞进行计数,确保它们在 7.5 x 105 至 1.2 x 106 个细胞/mL 之间。
- 在 30 mL PBS 中制备 300 μg pHEK-FLAG-HAT1 和 300 μg pHEK-Rbap46 质粒 DNA 的混合物。向 DNA/PBS 中加入 1.2 mL 聚乙烯亚胺 (PEI) 并混合。将DNA / PBS / PEI混合物加入300mL培养物中,并在37°C下以120rpm孵育48小时。
- 收获细胞
- 对细胞进行计数,确保它们介于 1.7 x 106 至 2.5 x 106 个细胞/mL 之间。在该细胞密度下,加入毛喉素至终浓度为12.5μM以激活HAT1并孵育细胞30分钟,37°C,120rpm。
- 将细胞以 300 x g 沉淀 5 分钟,用 30 mL PBS 洗涤一次,并在液体 N2 中快速冷冻。储存在-80°C直至蛋白质纯化或直接进行纯化。
- HAT1/Rbap46复合物FLAG微珠纯化
- 裂解细胞并制备蛋白质提取物。
- 通过向 50 mL RSB-500(20 mM Tris-HCl pH 7.5、500 mM NaCl、25 mM MgCl2)中加入一片蛋白酶抑制剂混合物 (PIC) 来制备裂解缓冲液。
- 在冰上解冻来自 300 mL 培养物的细胞沉淀,并用 40 mL 含有 0.1% Triton X-100 的冰冷裂解缓冲液裂解。在冰上超声处理,然后在 4°C 下以 10,000 x g 旋转 10 分钟。 将所有上清液收集到一个 50 mL 的冰管中,现在是蛋白质提取物。
- 准备FLAG珠子。
- 将 400 μL M2-FLAG 琼脂糖珠浆液加入每个含有 5 mL 0.1 M 甘氨酸 pH 3.5、0.01% Triton X-100 的试管中,将 FLAG 微珠分成四个 15 mL 锥形管。用手用力摇晃每根管子 5 秒。
- 脉冲离心机在4°C下以1000× g 离心30秒,吸入上清液,用5mLRSB-500 + 0.1%Triton X-100(Tx-100;无蛋白酶抑制剂)洗涤两次,并在冰上孵育。
- 进行 FLAG 免疫沉淀。
- 将 10 mL 蛋白质提取物加入每个 15 mL 含有洗涤珠子的锥形管中。将管在4°C下倒置旋转孵育至少90分钟或过夜。
- 在 10 mL RSB-500 + 0.1% Tx-100 中洗涤结合的珠子 5 次。对于每次洗涤,将管倒置2x-5x以重悬珠子,然后在4°C下以1000× g 沉淀1分钟。 最后,在RSB-100(20mM Tris-HCl pH 7.5,100mM NaCl,25mM MgCl2)+ 0.1%Tx-100中洗涤1x。
- FLAG 肽洗脱:在含有 0.5 mg/mL FLAG 肽的 1.5 mL 洗脱缓冲液 (EB) 中洗脱 HAT1 复合物。在4°C孵育至少1小时或过夜。在4°C下以1000× g 脉冲离心洗脱的珠子30秒,并收集上清液,其中含有纯化的HAT 1 / Rbap46复合物。
- 浓缩蛋白质并去除FLAG肽。
- 将洗脱液加入 20 mL、10,000 道尔顿截止过滤管中。使用 EB 将体积提高到 15 mL。在4°C下以2500× g 旋转15分钟,每次用额外的15mLEB重复两次。
- 从过滤管中回收最终洗脱液,并用 A260 检查蛋白质浓度。预计 6 mL EB 中总蛋白的浓度为 1 mg(每 300 mL 起始培养物)。通过SDS-PAGE,考马斯和免疫印迹检查蛋白质纯度,并将等分试样储存在-80°C。
- 裂解细胞并制备蛋白质提取物。
2. 方法二:HAT1乙酰点击标准曲线
- 准备标准曲线
- 合成阳性对照 H4 N 末端肽,序列如下:SCRG[Pra]GGKGLG[Pra]GGAKRHRKVLRGG[Lys(Biotin)],其中 [Pra] 表示炔丙基甘氨酸。
- 将 Pra 肽重悬至 0.1 mg/mL 的 DMSO。将Pra肽与生物素化组蛋白H4肽(1-23-GGK-生物素)混合以创建标准曲线(图2; 体积见表1)。标准曲线可以在板结合之前新鲜制备,或者如果提前制备,则与20μL8M尿素混合,并在-20°C下储存至步骤3.3。
3. 方法3:HAT1乙酰点击法
- 用测试抑制剂组装乙酰化反应
- 在0.2 mL PCR管或96孔PCR板中从以下组分组装乙酰化反应一式两份:生物素化组蛋白H4肽(1-23-GGK-生物素)重悬于DMSO中至0.1mg / mL(34.8μM),HAT1酶在EB中预稀释,20x缓冲液(1M Tris pH 8.5,0.1%NP40),2mM DTT,4-戊酰辅酶A溶于水至1mg / mL(1mM)。20 μL 反应包括 10 μL 酶、1 μL H4 肽、1 μL 20x 缓冲液、1 μL 预混合的 DTT,并分装到冰上 96 孔 PCR 板的孔中。
- 每孔加入 1 μL DMSO(阴性对照)或溶解在 1-10 μM 的测试化合物或 H4K12CoA25 (或合适的阳性对照抑制剂)。通过轻轻移液混合并在冰上孵育 10 分钟,以形成酶:抑制剂复合物。
- 乙酰化反应继续
- 将 2 μL 4-戊酰辅酶 A 与 4 μL 水混合,然后加入孔中。通过移液轻轻混合,在室温下以300× g 离心30秒以收集内容物,并在37°C下用可重新密封的箔在带盖的管或板中孵育1小时。
- 直接处理反应产物的内容物或用20μL8M尿素淬灭,并在-20°C下储存直至加工。
- 肽与中性亲和素平板结合
- 将反应内容物加入牛血清白蛋白 (BSA) 预封闭的黑色中性亲和素包被的 96 孔板中,每孔含有 80 μL PBST(PBS + 0.1% 吐温-20)。
- 将标准曲线肽一式两份加入到含有 80 μL PBST 的孔中。在室温(RT)下用轻轻的轨道振荡结合肽1小时。
- 洗涤:结合完成后,从孔中吸出液体,并使用洗板机用 200 μL PBST 3x 15 冲程(180 μL 冲程体积)洗涤孔。
- 点击化学反应
- 按如下方式制备点击反应的试剂:100 mM Tris(3-羟丙基三唑基甲基)胺 (THPTA) 配体在水中和 20 mM CuSO4 在水中。THPTA 可防止 Cu(II) 催化水解并淬灭由 O2/Cu/抗坏血酸产生的自由基和过氧化物。
- 将THPTA/Cu混合物加入300mM抗坏血酸钠的水溶液和2.5mM生物素叠氮化物的DMSO溶液中。一键反应含有 140 μL PBS、10 μL THPTA、10 μL CuSO4、10 μL 抗坏血酸钠、20 μL 生物素叠氮化物。始终将点击试剂新鲜混合,然后向每个孔中分配 190 μL,密封板,并在 37°C 下孵育 1 小时。
- 洗涤:从孔中吸出液体,并用PBST洗涤板3次(180μL冲程体积,每次洗涤15次)。
- 链霉亲和素-辣根过氧化物酶结合:在链霉亲和素 (0.224 mg/mL) 中以 1:10 的比例稀释链霉亲和素 (0.224 mg/mL),然后在 PBST 中以 1:1000 的比例进一步稀释。加入Steptavidin-HRP:链霉亲和素混合物(每孔100μL),并在室温下孵育1小时,轻轻摇动轨道。
- 如上所述,用PBST洗涤3次。
- Amplex 红色氧化
- 按如下方式组合 Amplex 红检测试剂:4.45 mL NaHPO4 缓冲液(1x = 50 mM 终浓度)、50 μL amplex 红(20 mM 在 DMSO 中稀释)、500 μL 稀释的 H2O2。
- 在 1x NaHPO4 缓冲液中将 H2O2 从 30% 的原液稀释至 3%,然后将 22.7 μL 的 3% H2O2 加入到 977 μL 的 1x NaHPO4 缓冲液中(这是用于 amplex 反应的 H2O2)。每孔加入 100 μL amplex red 反应混合物,在室温下避光孵育 30 分钟,然后使用标准荧光酶标仪检测荧光激发/发射 571/585 nm。
- 酶抑制的计算:根据以下公式计算抑制百分比(图3):
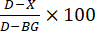 其中 D 是仅用DMSO处理的对照反应的荧光值, X 是用测试化合物处理的反应的值, BG 是背景孔的值(H4肽对照,不添加酶)。
其中 D 是仅用DMSO处理的对照反应的荧光值, X 是用测试化合物处理的反应的值, BG 是背景孔的值(H4肽对照,不添加酶)。 - 剂量曲线:选择发现可抑制 HAT1 酶活性的测试化合物进行重复测定,并连续稀释该化合物。根据经验测定化合物稀释度。例如,从 2 mM 原液浓度开始,然后以 1:3 的比例连续稀释 8 次。然后以 1:20 的比例稀释到酶测定中,产生 100 μM 最高剂量。
- 绘制曲线:在数据分析软件(GraphPad Prism)中使用最小二乘回归拟合剂量反应抑制曲线(使用步骤3.10中的抑制百分比值),并得出每种化合物的IC50 值(图4)。
- 与乙酰辅酶A的乙酰化反应:
- 用它来确认天然辅因子乙酰辅酶A而不是4-戊酰辅酶A的酶活性。按照步骤3.1-3.2中所述进行HAT1乙酰化测定,用乙酰辅酶A代替4-戊酰辅酶A。
- 将反应产物点在硝酸纤维素膜(1-2μL)上,使其干燥,然后用抗H4-赖氨酸-12-乙酰或抗H4-赖氨酸-5-乙酰抗体点印。通过密度测定法定量免疫印迹信号。
结果
每个板上应包含一式两份(16 孔)的标准曲线,以确保适当的测定性能。标准曲线数据应以表格形式设置,根据溶液中含 Pra 肽与天然 H4 肽的比例,范围为 100% 至 0%(表 1)。Amplex 红色信号在 100% pra/0% 天然 H4 肽孔中最高,在 0% pra/100% 天然 H4 肽孔中最低。在检测到荧光并对孔进行平均后,得到的标准品图应拟合一条直线(图2),尽管可以观察到浓度高于50%Pra的...
讨论
在过去的十年中,点击化学变得突出20,使相互作用的化学结构的精确设计成为可能。在此背景下,各种生物正交共价连接21 已成为在其自然环境中形成复合物的有希望的选择。点击化学采用表现出快速和选择性反应的官能团对,通常称为"点击反应"。这些反应在环保、温和的水性条件下有效发生。在这里,我们介绍了一种旨在鉴定和表征 HAT1 乙酰转移酶活性
披露声明
我们已经申请了描述HAT1乙酰点击测定法的专利(PCT/US20/29395)。J.J.G. 报告了与 Sharma Therapeutics, LLC 和 Guidepoint 的咨询协议,以及 Hummingbird Biosciences 的研究支持(对他的机构)。
致谢
我们感谢 George Zheng 提供 H4K12CoA。我们感谢格鲁伯实验室的成员进行有益的讨论和反馈。我们感谢 NIH/NCI (1K08CA245024)、CPRIT (RR200090) 和 V 基金会 (V2022-022) 的支持。
材料
| Name | Company | Catalog Number | Comments |
| 4P CoA | Cayman Chemical | 10547 | Click chemistry co-factor |
| Amplex Red | Fisher Sci | A12222 | Fluorescence substrate |
| Biotin-PEG-Azide | Alfa Aesar | J64996MC | Click chemistry |
| Copper Sulfate | Sigma-aldrich | 7758-98-7 | Click chemistry |
| DMSO | Fisher Scientific | 67-68-5 | diluent |
| DTT | Acros Organics | 03-12-3483 | reducting agent |
| Forskolin | VWR | 102987-310 | Protein expression |
| Freestyle 293 Expression Medium | Thermo Fisher | 12338018 | Media |
| Freestyle 293-F cells | Thermo Fisher | R790-07 | Protein expression |
| H4-peptide/1-23-GGK-biotin | Anaspec | AS65097 | peptide substrate |
| HEPES | Sigma-aldrich | 7365-45-9 | EB buffer |
| Hydrogen peroxide 30% solution | Sigma-aldrich | Z00183-99-0 | initiator |
| M2 FLAG antibody slurry | Millipore-Sigma | A2220 | Protein purification |
| Macrosep 10K Filter (Pall Lab) | VWR | 89131-980 | Protein purification |
| Neutravidin Plate | Thermo Sci | 15127 | BSA-pre-blocked |
| NP40 (IGEPAL) | MP Biomedical | 198596 | 20x buffer |
| pHEK-293 plasmid | Takara Bio | 3390 | Protein expression |
| Phosphate Buffered Saline 10x | Alfa Aesar | Z00082-33-6 | wash buffer |
| Pra peptide | Genscript | Custom synthesis | biotinylated |
| Sodium Ascorbate | Sigma-aldrich | 134-03-2 | Click chemistry |
| Sodium chloride | Sigma-aldrich | 7647-14-5 | EB buffer |
| Sodium phosphate | VWR International | 7558-80-7 | buffer |
| Streptavidin | EMD Millipore | 189730 | competitor |
| Streptavidin-HRP | Cell Signaling | 3999S | enzyme |
| THPTA ligand | Fisher Sci | 1010-500 | Click chemistry |
| Tris base | Sigma-aldrich | 77-86-1 | 20x buffer |
| Triton-X 100 | VWR International | 9002-93-1 | EB buffer |
| Tween-20 | Sigma-aldrich | 9005-64-5 | Wash buffer |
| Urea | Sigma-Aldrich | 57-13-6 | quencher |
参考文献
- Kleff, S., et al. Identification of a gene encoding a yeast histone H4 acetyltransferase. J Biol Chem. 270 (42), 24674-24677 (1995).
- Parthun, M. R., Widom, J., Gottschling, D. E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism. Cell. 87 (1), 85-94 (1996).
- Verreault, A., et al. Nucleosomal DNA regulates the core-histone-binding subunit of the human Hat1 acetyltransferase. Curr Biol. 8 (2), 96-108 (1998).
- Parthun, M. R. Histone acetyltransferase 1: more than just an enzyme. Biochim Biophys Acta. 1819 (3-4), 256-263 (2013).
- Gruber, J. J., et al. HAT1 coordinates histone production and acetylation via H4 promoter binding. Mol Cell. 75 (4), 711-724 e5 (2019).
- Fan, P., et al. Overexpressed histone acetyltransferase 1 regulates cancer immunity by increasing programmed death-ligand 1 expression in pancreatic cancer. J Exp Clin Cancer Res. 38 (1), 47 (2019).
- Xia, P., et al. MicroRNA-377 exerts a potent suppressive role in osteosarcoma through the involvement of the histone acetyltransferase 1-mediated Wnt axis. J Cell Physiol. 234 (12), 22787-22798 (2019).
- Yang, G., et al. Histone acetyltransferase 1 is a succinyltransferase for histones and non-histones and promotes tumorigenesis. EMBO Rep. 22 (2), e50967 (2021).
- Xue, L., et al. RNAi screening identifies HAT1 as a potential drug target in esophageal squamous cell carcinoma. Int J Clin Exp Pathol. 7 (7), 3898-3907 (2014).
- Zhang, W., et al. Structural plasticity of histones H3-H4 facilitates their allosteric exchange between RbAp48 and ASF1. Nat Struct Mol Biol. 20 (1), 29-35 (2013).
- Campos, E. I., et al. The program for processing newly synthesized histones H3.1 and H4. Nat Struct Mol Biol. 17 (11), 1343-1351 (2010).
- Agudelo Garcia, P. A., et al. Identification of multiple roles for histone acetyltransferase 1 in replication-coupled chromatin assembly. Nucleic Acids Res. 45 (16), 9319-9335 (2017).
- Nagarajan, P., et al. Histone acetyl transferase 1 is essential for mammalian development, genome stability, and the processing of newly synthesized histones H3 and H4. PLoS Genet. 9 (6), e1003518 (2013).
- Annunziato, A. T. Assembling chromatin: the long and winding road. Biochim Biophys Acta. 1819 (3-4), 196-210 (2013).
- Annunziato, A. T., Seale, R. L. Histone deacetylation is required for the maturation of newly replicated chromatin. J Biol Chem. 258 (20), 12675-12684 (1983).
- Dahlin, J. L., et al. Assay interference and off-target liabilities of reported histone acetyltransferase inhibitors. Nat Commun. 8 (1), 1527 (2017).
- Baell, J. B., Miao, W. Histone acetyltransferase inhibitors: where art thou. Future Med Chem. 8 (13), 1525-1528 (2016).
- Lasko, L. M., et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours. Nature. 550 (7674), 128-132 (2017).
- Baell, J. B., et al. Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. Nature. 560 (7717), 253-257 (2018).
- Falk, H., et al. An efficient high-throughput screening method for MYST family acetyltransferases, a new class of epigenetic drug targets. J Biomol Screen. 16 (10), 1196-1205 (2011).
- He, M., et al. Chemical biology approaches for investigating the functions of lysine acetyltransferases. Angew Chem Int Ed Engl. 57 (5), 1162-1184 (2018).
- Lipchik, A. M., et al. A peptide-based biosensor assay to detect intracellular Syk kinase activation and inhibition. Biochemistry. 51 (38), 7515-7524 (2012).
- Song, J., et al. Chemoproteomic profiling of protein substrates of a major lysine acetyltransferase in the native cellular context. ACS Chem Biol. 17 (5), 1092-1102 (2022).
- Gaddameedi, J. D., et al. Acetyl-click screening platform identifies small-molecule inhibitors of histone acetyltransferase 1 (HAT1). J Med Chem. 66 (8), 5774-5801 (2023).
- Ngo, L., Brown, T., Zheng, Y. G. Bisubstrate inhibitors to target histone acetyltransferase 1 (HAT1). Chem Biol Drug Des. 93 (5), 865-873 (2019).
- Parker, C. G., Pratt, M. R. Click chemistry in proteomic investigations. Cell. 180 (4), 605-632 (2020).
- Islam, K. The bump-and-hole tactic: Expanding the scope of chemical genetics. Cell Chem Biol. 25 (10), 1171-1184 (2018).
- Radziwon, K., Weeks, A. M. Protein engineering for selective proteomics. Curr Opin Chem Biol. 60, 10-19 (2021).
- Rich, R. L., Myszka, D. G. Survey of the year 2007 commercial optical biosensor literature. J Mol Recognit. 21 (6), 355-400 (2008).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。