È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Un test chimico acetil-click per misurare l'acetiltransferasi istonica 1
In questo articolo
Riepilogo
Saggi chimici rapidi e accurati per lo screening di inibitori specifici sono uno strumento importante nell'arsenale di sviluppo dei farmaci. Qui, presentiamo un saggio chimico scalabile acetil-click per misurare l'inibizione dell'attività di acetilazione di HAT1.
Abstract
HAT1, noto anche come istone acetiltransferasi 1, svolge un ruolo cruciale nella sintesi della cromatina stabilizzando e acetilando l'H4 nascente prima dell'assemblaggio del nucleosoma. È necessario per la crescita del tumore in vari sistemi, il che lo rende un potenziale bersaglio per il trattamento del cancro. Per facilitare l'identificazione di composti in grado di inibire l'attività enzimatica di HAT1, abbiamo ideato un saggio acetil-click per uno screening rapido. In questo semplice saggio, impieghiamo HAT1/Rbap46 ricombinante, che viene purificato da cellule umane attivate. Il metodo utilizza l'analogo dell'acetil-CoA 4-pentinoil-CoA (4P) in un approccio click-chemistry. Ciò comporta il trasferimento enzimatico di un manico alchino attraverso una reazione di acilazione HAT1-dipendente a un peptide N-terminale H4 biotinilato. Il peptide catturato viene quindi immobilizzato su piastre di neutravidina, seguita da una funzionalizzazione click-chemistry con biotina-azide. Successivamente, il reclutamento della streptavidina-perossidasi viene impiegato per ossidare il rosso amplex, con conseguente emissione fluorescente quantitativa. Introducendo inibitori chimici durante la reazione di acilazione, possiamo quantificare l'inibizione enzimatica in base ad una riduzione del segnale di fluorescenza. È importante sottolineare che questa reazione è scalabile, consentendo lo screening ad alto rendimento di potenziali inibitori per l'attività enzimatica di HAT1.
Introduzione
Tra le numerose acetiltransferasi eucariotiche, HAT1 è stata la prima istone acetiltransferasi ad essere isolata 1,2,3. Indagini successive hanno stabilito il suo ruolo fondamentale nella replicazione della cromatina, in particolare nella sintesi di nuovi nucleosomi durante la faseS 4. I nostri sforzi di ricerca hanno portato al riconoscimento che HAT1 è altamente stimolato dal trattamento con fattore di crescita epidermico (EGF) nelle cellule mammarie5. Inoltre, è venuto alla luce che HAT1 è necessario per una rapida proliferazione cellulare e formazione tumorale in vivo 6,7,8,9. I dati indicano che HAT1 è fondamentale nel coordinare i processi anabolici ed epigenetici per la divisione cellulare, guidando la crescita del tumore.
HAT1 di-acetila la coda amino-terminale dell'istone H4 sulle lisine 5 e 12 in complesso con la proteina chaperone Rbap46, che lega l'istone e presenta l'amino-terminale a HAT1. I tetrameri istonici o disomi10, insieme a HAT1/Rbap46 e altri chaperoni istonici11, vengono quindi importati nel nucleo. Gli istoni vengono quindi rilasciati per essere depositati sulla forcella di replicazione o in altri siti per supportare l'attivazione o la repressione genica. La funzione del segno di diacetilazione HAT1 sull'istone H4 non è completamente compresa. È probabile che venga rapidamente rimosso entro un intervallo di 15-30 minuti dall'azione delle deacetilasi istoniche 12,13,14,15 dopo che H4 è stato inserito nella cromatina. Pertanto, il marchio di diacetilazione HAT1 non si propaga nella cromatina e potrebbe non svolgere un vero ruolo epigenetico, sebbene sia stato ipotizzato un ruolo nel reclutamento di enzimi modificanti la cromatina nella cromatina nascente12. Inoltre, HAT1 non acetila direttamente la cromatina; La sua attività è limitata agli istoni solubili.
Lo sviluppo di inibitori dell'acetiltransferasi istonica a piccole molecole è stato ostacolato da saggi aspecifici e a basso rendimento, che spesso hanno portato alla generazione di composti biologicamente reattivi 16,17. Il test gold standard per misurare le attività dell'acetiltransferasi richiede l'uso di 3H-acetil-coA, che limita la produttività e richiede radiazioni. Ciononostante, recentemente sono stati descritti e confermati inibitori specifici e altamente potenti dell'acetiltransferasi a piccole molecole che hanno come bersaglio CBP/p30018 e KAT6A/B19,20 attraverso l'uso di 3H-acetil-CoA. In futuro, si stanno studiando saggi migliorati per ottenere una migliore produttività ed evitare i rischi di laboratorio.
I recenti progressi nel monitoraggio dell'acetilazione21 hanno utilizzato la click chemistry per consentire il monitoraggio delle reazioni enzimatiche. Esiste una varietà di precursori click-enabled che sono accessibili tramite semplici vie sintetiche o disponibili per l'acquisto che possono essere incorporati nelle reazioni enzimatiche. Queste reazioni sono tipicamente condotte in sistemi ricombinanti, sebbene siano possibili anche saggi basati su cellule22. Il vantaggio dei cofattori e dei substrati abilitati al clic è che lo screening può misurare direttamente l'attività enzimatica senza la necessità di sistemi di lettura accoppiati che sono spesso perturbati dai composti di screening e richiedono ulteriori passaggi di manipolazione. Ciò consente trattamenti con inibitori solo durante la fase enzimatica, mentre tutte le fasi di funzionalizzazione e rilevamento a valle vengono eseguite dopo un lavaggio approfondito per rimuovere i composti, limitando così il potenziale di interferenza del saggio. Questi vantaggi rendono la progettazione di saggi click-enabled preferibile ai saggi accoppiati che comunemente si basano sulla rilevazione del coenzima A libero.
Una considerazione importante è l'accettazione di cofattori abilitati al clic nel sito attivo dell'enzima. I cofattori abilitati per i clic esistenti potrebbero non essere completamente compatibili con il sito attivo ottimizzato per il cofattore nativo. Le informazioni strutturali e la modellazione possono essere utilizzate per progettare sostituzioni di amminoacidi per allargare il sito attivo per incorporare substrati alterati23. Ciò può consentire lo screening con una migliore cinetica enzimatica e livelli più bassi di substrato ed enzimi. Lo svantaggio di questo approccio è che le tasche catalitiche alterate potrebbero non identificare gli inibitori che interagiscono fortemente con l'enzima nativo. In definitiva, è necessaria una combinazione di approcci per identificare e convalidare potenziali inibitori enzimatici.
Qui, descriviamo un metodo sviluppato per purificare e saggiare l'attività dell'enzima HAT1 utilizzando il cofattore click 4-penninoil-CoA24. Questo test (Figura 1) utilizza la sequenza enzimatica nativa in complesso con la sua proteina partner richiesta Rbap46, che ha dimostrato di aumentare l'attività enzimatica. La purificazione dell'enzima dalle cellule umane consente l'attivazione enzimatica nella cellulo, che può preservare le stimolanti modificazioni post-traduzionali importanti per la piena attività enzimatica. La progettazione e l'ottimizzazione di saggi enzimatici ricombinanti per schermi chimici ad alto rendimento sono state utilizzate con successo per identificare e caratterizzare gli inibitori delle piccole molecole HAT1.
Protocollo
1. Metodo 1: produzione e purificazione del complesso ricombinante HAT1/Rbap46
- Scongelamento, recupero ed espansione delle cellule HEK293f
- Scongelare 1-10 milioni di cellule di mammifero HEK293f26 in terreni di espressione freestyle 293 da 30 mL in un pallone da 100 mL. Incubare in CO2 all'8% a 37 °C ruotando a 60 giri/min.
- Il giorno successivo, contare e controllare la vitalità delle celle, quindi regolare la velocità di rotazione a 120 giri/min. Espandere la coltura a 300 mL in un matraccio da 1 L, mantenendo la densità di semina a 500.000 cellule/mL e dividendo le cellule prima che la densità superi 3 x 106 cellule/mL.
- Trasfezione HEK293f
- Seminare 5 x 105 cellule/mL in coltura da 300 mL in un matraccio da 1 L e coltivare per 24 ore. Il giorno successivo, contare le cellule per assicurarsi che siano comprese tra 7,5 x 105 e 1,2 x 106 cellule/mL.
- Preparare una miscela di 300 μg di pHEK-FLAG-HAT1 e 300 μg di DNA plasmidico pHEK-Rbap46 in 30 mL di PBS. Aggiungere 1,2 mL di polietilenimmina (PEI) al DNA/PBS e mescolare. Incubare per 20 minuti a RT. Aggiungere la miscela DNA/PBS/PEI alla coltura da 300 mL e incubare a 37 °C per 48 ore a 120 giri/min.
- Cellule di raccolta
- Contare le cellule per assicurarsi che rientrino tra 1,7 x 106 e 2,5 x 106 cellule/mL. A questa densità cellulare, aggiungere forskolina a una concentrazione finale di 12,5 μM per attivare HAT1 e incubare le cellule per 30 minuti, 37 °C, 120 giri/min.
- Pellettare le cellule a 300 x g per 5 minuti, lavare una volta con 30 ml di PBS e congelare in liquido N2 . Conservare a -80 °C fino alla purificazione delle proteine o procedere direttamente alla purificazione.
- Purificazione del complesso FLAG-bead HAT1/Rbap46
- Lisare le cellule e preparare l'estratto proteico.
- Preparare il tampone di lisi aggiungendo una compressa di cocktail di inibitori della proteasi (PIC) a 50 mL di RSB-500 (20 mM Tris-HCl pH 7,5, 500 mM NaCl, 25 mM MgCl2).
- Scongelare il pellet cellulare da 300 mL di coltura su ghiaccio e lisare con 40 mL di tampone di lisi ghiacciato con Triton X-100 allo 0,1%. Sonicare su ghiaccio, quindi centrifugare a 10.000 x g per 10 minuti a 4°C. Raccogli tutto il surnatante in una provetta da 50 ml su ghiaccio, che ora è l'estratto proteico.
- Prepara le perline FLAG.
- Dividere le perle FLAG in quattro provette coniche da 15 mL aggiungendo 400 μL di impasto di perle di agarosio M2-FLAG in ciascuna provetta contenente 5 mL di glicina 0,1 M pH 3,5, 0,01% Triton X-100. Agitare energicamente ogni tubo per 5 secondi a mano.
- Centrifugare a impulsi per 30 s a 1000 x g a 4 °C, aspirare il surnatante, lavare due volte con 5 mL di RSB-500 + 0,1% Triton X-100 (Tx-100; nessun inibitore della proteasi) e incubare su ghiaccio.
- Eseguire l'immunoprecipitazione FLAG.
- Aggiungere 10 ml di estratto proteico a ciascuna provetta conica da 15 ml contenente le perle lavate. Incubare le provette a 4 °C con rotazione invertita per almeno 90 minuti o per tutta la notte.
- Lavare le perline rilegate 5 volte in 10 ml di RSB-500 + 0,1% Tx-100. Per ogni lavaggio, capovolgere il tubo 2x-5x per risospendere le perle, quindi pellet a 1000 x g per 1 min a 4 °C. Infine, lavare 1 volta in RSB-100 (20 mM Tris-HCl pH 7,5, 100 mM NaCl, 25 mM MgCl2) + 0,1% Tx-100.
- Eluizione del peptide FLAG: eluire il complesso HAT1 in 1,5 mL di tampone di eluizione (EB) contenente 0,5 mg/mL di peptide FLAG. Incubare a 4 °C per almeno 1 ora o per tutta la notte. Centrifugare a impulsi le perle eluite a 1000 x g per 30 s a 4 °C e raccogliere il surnatante, che contiene il complesso HAT1/Rbap46 purificato.
- Concentrare le proteine e rimuovere il peptide FLAG.
- Aggiungere l'eluato a un tubo filtrante da 20 mL e 10.000 Dalton. Portare il volume a 15 mL con EB. Centrifugare a 2500 x g per 15 minuti a 4 °C, ripetendo due volte con altri 15 ml di EB ogni volta.
- Recuperare l'eluato finale dal tubo del filtro e controllare la concentrazione proteica con A260. Aspettatevi una concentrazione di 1 mg di proteine totali in 6 mL di EB (per 300 mL di coltura iniziale). Controllare la purezza delle proteine mediante SDS-PAGE, Coomassie e immunoblotting e conservare le aliquote a -80 °C.
- Lisare le cellule e preparare l'estratto proteico.
2. Metodo 2: curva standard HAT1 acetil-click
- Preparazione della curva standard
- Sintetizzare un peptide N-terminale H4 di controllo positivo con la sequenza: SCRG[Pra]GGKGLG[Pra]GGAKRHRKVLRGG[Lys(Biotina)], dove [Pra] indica la Proparglilglicina.
- Sospendere nuovamente il peptide Pra a 0,1 mg/mL in DMSO. Miscelare il peptide Pra con il peptide H4 dell'istone biotinilato (1-23-GGK-biotina) per creare una curva standard (Figura 2; volumi riportati nella Tabella 1). Le curve standard possono essere fatte fresche prima della legatura su piastra o, se fatte in anticipo, miscelate con 20 μL di urea 8 M e conservate a -20 °C fino alla fase 3.3.
3. Metodo 3: test dell'acetil-click HAT1
- Assemblaggio delle reazioni di acetilazione con gli inibitori del test
- Assemblare le reazioni di acetilazione in duplicato in provette PCR da 0,2 mL o piastre PCR a 96 pozzetti dai seguenti componenti: peptide H4 dell'istone biotinilato (1-23-GGK-biotina) risospeso in DMSO a 0,1 mg/mL (34,8 μM), enzima HAT1 pre-diluito in EB, tampone 20x (1M Tris pH 8,5, 0,1% NP40), DTT 2 mM, 4-pentinoil-CoA disciolto in acqua a 1 mg/mL (1 mM). Una reazione di 20 μL comprende 10 μL di enzima, 1 μL di peptide H4, 1 μL di tampone 20x, 1 μL di DTT premiscelato e aliquotato ai pozzetti di una piastra PCR a 96 pozzetti su ghiaccio.
- Aggiungere 1 μL di DMSO (controllo negativo) o composto in esame disciolto a 1-10 μM, o H4K12CoA25 (o inibitore del controllo positivo adatto), per pozzetto. Miscelare con pipettaggio delicato e incubare per 10 minuti su ghiaccio per consentire la formazione di complessi enzima-inibitore.
- Continuazione della reazione di acetilazione
- Combinare 2 μL di 4-pentinoil-CoA con 4 μL di acqua, quindi aggiungere ai pozzetti. Miscelare delicatamente mediante pipettaggio, centrifugare a 300 x g per 30 s a RT per raccogliere il contenuto e incubare a 37 °C per 1 h in provette o piastre tappate con pellicola richiudibile.
- Trattare il contenuto direttamente per i prodotti di reazione o temprare con 20 μL di urea 8 M e conservare a -20 °C fino alla lavorazione.
- Legame del peptide alla piastra della neutravidina
- Aggiungere il contenuto della reazione con o senza urea a piastre a 96 pozzetti rivestite di Neutravidina nera pre-bloccata con albumina sierica bovina (BSA) contenenti 80 μL di PBST (PBS + 0,1% Tween-20) per pozzetto.
- Aggiungere peptidi a curva standard in duplicato ai propri pozzetti contenenti 80 μL di PBST. Legare i peptidi con un leggero scuotimento orbitale per 1 ora a temperatura ambiente (RT).
- Lavaggio: Al termine del legame, aspirare il liquido dai pozzetti e lavare i pozzetti con 200 μL di PBST 3x 15 colpi (volume di corsa 180 μL) utilizzando una rondella per piastre.
- Fare clic sulla reazione chimica
- Preparare i reagenti per la reazione di clic come segue: 100 mM di tris(3-idrossipropiltriazolimetil)ammina (THPTA) ligando in acqua e 20 mM CuSO4 in acqua. Il THPTA previene l'idrolisi catalizzata da Cu(II) e spegne i radicali e i perossidi generati da O2/Cu/ascorbato.
- Aggiungere la miscela THPTA/Cu a 300 mM di ascorbato di sodio in acqua e 2,5 mM di biotina azide in DMSO. La reazione con un clic contiene 140 μL di PBS, 10 μL di THPTA, 10 μL di CuSO4, 10 μL di ascorbato di sodio, 20 μL di biotina-azide. Miscelare sempre i reagenti a scatto freschi, quindi erogare 190 μL in ciascun pozzetto, sigillare la piastra e incubare a 37°C per 1 ora.
- Lavaggio: Aspirare il liquido dai pozzetti e lavare la piastra 3 volte con PBST (volume di corsa di 180 μL, 15 colpi per ogni lavaggio).
- Legame streptavidina-perossidasi di rafano: diluire streptavidina-HRP (0,224 mg/mL) 1:10 in streptavidina (0,224 mg/mL), quindi diluire ulteriormente 1:1000 in PBST. Aggiungere Steptavidin-HRP: miscela di streptavidina (100 μL per pozzetto) e incubare a RT per 1 ora con un leggero agitazione orbitale.
- Lavare 3 volte con PBST come nei passaggi precedenti.
- Ossidazione rossa Amplex
- Combinare i reagenti per la rilevazione del rosso Amplex come segue: 4,45 mL di tampone NaHPO4 (1x = 50 mM di concentrazione finale), 50 μL di amplex rosso (20 mM diluiti in DMSO), 500 μL di H2O2 diluito.
- Diluire H2O2 dal 30% al 3% in 1x tampone NaHPO4 , quindi aggiungere 22,7 μL di H2O2 al 3% in 977 μL di 1x tampone NaHPO4 (questo è l'H2O2 utilizzato per la reazione amplex). Aggiungere 100 μL della miscela di reazione rossa amplex per pozzetto, incubare a RT per 30 minuti al riparo dalla luce, quindi rilevare l'eccitazione/emissione di fluorescenza 571/585 nm utilizzando un lettore di piastre a fluorescenza standard.
- Calcolo dell'inibizione enzimatica: calcolare l'inibizione percentuale (Figura 3) secondo la seguente formula:
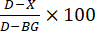 , dove D è il valore di fluorescenza delle reazioni di controllo trattate solo con DMSO, X è il valore delle reazioni trattate con composti in esame e BG è il valore dei pozzetti di fondo (controllo del peptide H4, nessun enzima aggiunto).
, dove D è il valore di fluorescenza delle reazioni di controllo trattate solo con DMSO, X è il valore delle reazioni trattate con composti in esame e BG è il valore dei pozzetti di fondo (controllo del peptide H4, nessun enzima aggiunto). - Curva di dose: selezionare i composti in esame che inibiscono l'attività dell'enzima HAT1 per saggi ripetuti, con il composto diluito in serie. Determinare empiricamente le diluizioni del composto. Ad esempio, iniziare con una concentrazione di 2 mM, quindi diluire in serie 1:3 per otto diluizioni. Quindi diluire 1:20 in saggi enzimatici che producono una dose massima di 100 μM.
- Rappresentazione grafica della curva: utilizzare la regressione dei minimi quadrati per adattare le curve di inibizione dose-risposta (utilizzando i valori di inibizione percentuale del passaggio 3.10) nel software di analisi dei dati (GraphPad Prism) e derivare i valori IC50 per ciascun composto (Figura 4).
- Reazione di acetilazione con acetil-CoA:
- Usalo per confermare l'attività enzimatica con il cofattore nativo acetil-CoA invece di 4-penninoil-CoA. Eseguire saggi di acetilazione di HAT1 come descritto nei passaggi 3.1-3.2 con acetil-CoA al posto di 4-pentinoil-CoA.
- Individuare i prodotti di reazione su membrane di nitrocellulosa (1-2 μL), lasciarli asciugare, quindi tamponare con anticorpi anti-H4-lisina-12-acetil o anti-H4-lisina-5-acetile. Quantificare il segnale immunoblot mediante densitometria.
Risultati
Le curve standard in duplicato (16 pozzetti) devono essere incluse su ogni piastra per garantire una corretta esecuzione del saggio. I dati della curva standard devono essere impostati sotto forma di tabella, con un intervallo compreso tra il 100% e lo 0% in base al rapporto tra il peptide contenente Pra e il peptide H4 nativo in soluzione (Tabella 1). Il segnale rosso di Amplex sarà il più alto nei pozzetti peptidici H4 nativi al 100% pra/0% e il più basso nei pozzetti peptidici H4 nativi allo 0% pra...
Discussione
Nell'ultimo decennio, la click chemistry è diventata importante20, consentendo la progettazione precisa di strutture chimiche interagenti. In questo contesto, varie connessioni covalenti bioortogonali21 sono emerse come opzioni promettenti per la formazione di complessi nel loro ambiente naturale. La chimica dei clic impiega coppie di gruppi funzionali che mostrano reazioni rapide e selettive, comunemente note come "reazioni di clic". Queste reazioni si verificano in modo ...
Divulgazioni
Abbiamo depositato brevetti che descrivono il test acetil-click HAT1 (PCT/US20/29395). J.J.G. riferisce di accordi di consulenza con Sharma Therapeutics, LLC e Guidepoint e di supporto alla ricerca (alla sua istituzione) da parte di Hummingbird Biosciences.
Riconoscimenti
Ringraziamo George Zheng per aver fornito H4K12CoA. Ringraziamo i membri del Gruber Lab per le utili discussioni e feedback. Ringraziamo il supporto di NIH/NCI (1K08CA245024), CPRIT (RR200090) e V Foundation (V2022-022).
Materiali
| Name | Company | Catalog Number | Comments |
| 4P CoA | Cayman Chemical | 10547 | Click chemistry co-factor |
| Amplex Red | Fisher Sci | A12222 | Fluorescence substrate |
| Biotin-PEG-Azide | Alfa Aesar | J64996MC | Click chemistry |
| Copper Sulfate | Sigma-aldrich | 7758-98-7 | Click chemistry |
| DMSO | Fisher Scientific | 67-68-5 | diluent |
| DTT | Acros Organics | 03-12-3483 | reducting agent |
| Forskolin | VWR | 102987-310 | Protein expression |
| Freestyle 293 Expression Medium | Thermo Fisher | 12338018 | Media |
| Freestyle 293-F cells | Thermo Fisher | R790-07 | Protein expression |
| H4-peptide/1-23-GGK-biotin | Anaspec | AS65097 | peptide substrate |
| HEPES | Sigma-aldrich | 7365-45-9 | EB buffer |
| Hydrogen peroxide 30% solution | Sigma-aldrich | Z00183-99-0 | initiator |
| M2 FLAG antibody slurry | Millipore-Sigma | A2220 | Protein purification |
| Macrosep 10K Filter (Pall Lab) | VWR | 89131-980 | Protein purification |
| Neutravidin Plate | Thermo Sci | 15127 | BSA-pre-blocked |
| NP40 (IGEPAL) | MP Biomedical | 198596 | 20x buffer |
| pHEK-293 plasmid | Takara Bio | 3390 | Protein expression |
| Phosphate Buffered Saline 10x | Alfa Aesar | Z00082-33-6 | wash buffer |
| Pra peptide | Genscript | Custom synthesis | biotinylated |
| Sodium Ascorbate | Sigma-aldrich | 134-03-2 | Click chemistry |
| Sodium chloride | Sigma-aldrich | 7647-14-5 | EB buffer |
| Sodium phosphate | VWR International | 7558-80-7 | buffer |
| Streptavidin | EMD Millipore | 189730 | competitor |
| Streptavidin-HRP | Cell Signaling | 3999S | enzyme |
| THPTA ligand | Fisher Sci | 1010-500 | Click chemistry |
| Tris base | Sigma-aldrich | 77-86-1 | 20x buffer |
| Triton-X 100 | VWR International | 9002-93-1 | EB buffer |
| Tween-20 | Sigma-aldrich | 9005-64-5 | Wash buffer |
| Urea | Sigma-Aldrich | 57-13-6 | quencher |
Riferimenti
- Kleff, S., et al. Identification of a gene encoding a yeast histone H4 acetyltransferase. J Biol Chem. 270 (42), 24674-24677 (1995).
- Parthun, M. R., Widom, J., Gottschling, D. E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism. Cell. 87 (1), 85-94 (1996).
- Verreault, A., et al. Nucleosomal DNA regulates the core-histone-binding subunit of the human Hat1 acetyltransferase. Curr Biol. 8 (2), 96-108 (1998).
- Parthun, M. R. Histone acetyltransferase 1: more than just an enzyme. Biochim Biophys Acta. 1819 (3-4), 256-263 (2013).
- Gruber, J. J., et al. HAT1 coordinates histone production and acetylation via H4 promoter binding. Mol Cell. 75 (4), 711-724 e5 (2019).
- Fan, P., et al. Overexpressed histone acetyltransferase 1 regulates cancer immunity by increasing programmed death-ligand 1 expression in pancreatic cancer. J Exp Clin Cancer Res. 38 (1), 47 (2019).
- Xia, P., et al. MicroRNA-377 exerts a potent suppressive role in osteosarcoma through the involvement of the histone acetyltransferase 1-mediated Wnt axis. J Cell Physiol. 234 (12), 22787-22798 (2019).
- Yang, G., et al. Histone acetyltransferase 1 is a succinyltransferase for histones and non-histones and promotes tumorigenesis. EMBO Rep. 22 (2), e50967 (2021).
- Xue, L., et al. RNAi screening identifies HAT1 as a potential drug target in esophageal squamous cell carcinoma. Int J Clin Exp Pathol. 7 (7), 3898-3907 (2014).
- Zhang, W., et al. Structural plasticity of histones H3-H4 facilitates their allosteric exchange between RbAp48 and ASF1. Nat Struct Mol Biol. 20 (1), 29-35 (2013).
- Campos, E. I., et al. The program for processing newly synthesized histones H3.1 and H4. Nat Struct Mol Biol. 17 (11), 1343-1351 (2010).
- Agudelo Garcia, P. A., et al. Identification of multiple roles for histone acetyltransferase 1 in replication-coupled chromatin assembly. Nucleic Acids Res. 45 (16), 9319-9335 (2017).
- Nagarajan, P., et al. Histone acetyl transferase 1 is essential for mammalian development, genome stability, and the processing of newly synthesized histones H3 and H4. PLoS Genet. 9 (6), e1003518 (2013).
- Annunziato, A. T. Assembling chromatin: the long and winding road. Biochim Biophys Acta. 1819 (3-4), 196-210 (2013).
- Annunziato, A. T., Seale, R. L. Histone deacetylation is required for the maturation of newly replicated chromatin. J Biol Chem. 258 (20), 12675-12684 (1983).
- Dahlin, J. L., et al. Assay interference and off-target liabilities of reported histone acetyltransferase inhibitors. Nat Commun. 8 (1), 1527 (2017).
- Baell, J. B., Miao, W. Histone acetyltransferase inhibitors: where art thou. Future Med Chem. 8 (13), 1525-1528 (2016).
- Lasko, L. M., et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours. Nature. 550 (7674), 128-132 (2017).
- Baell, J. B., et al. Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. Nature. 560 (7717), 253-257 (2018).
- Falk, H., et al. An efficient high-throughput screening method for MYST family acetyltransferases, a new class of epigenetic drug targets. J Biomol Screen. 16 (10), 1196-1205 (2011).
- He, M., et al. Chemical biology approaches for investigating the functions of lysine acetyltransferases. Angew Chem Int Ed Engl. 57 (5), 1162-1184 (2018).
- Lipchik, A. M., et al. A peptide-based biosensor assay to detect intracellular Syk kinase activation and inhibition. Biochemistry. 51 (38), 7515-7524 (2012).
- Song, J., et al. Chemoproteomic profiling of protein substrates of a major lysine acetyltransferase in the native cellular context. ACS Chem Biol. 17 (5), 1092-1102 (2022).
- Gaddameedi, J. D., et al. Acetyl-click screening platform identifies small-molecule inhibitors of histone acetyltransferase 1 (HAT1). J Med Chem. 66 (8), 5774-5801 (2023).
- Ngo, L., Brown, T., Zheng, Y. G. Bisubstrate inhibitors to target histone acetyltransferase 1 (HAT1). Chem Biol Drug Des. 93 (5), 865-873 (2019).
- Parker, C. G., Pratt, M. R. Click chemistry in proteomic investigations. Cell. 180 (4), 605-632 (2020).
- Islam, K. The bump-and-hole tactic: Expanding the scope of chemical genetics. Cell Chem Biol. 25 (10), 1171-1184 (2018).
- Radziwon, K., Weeks, A. M. Protein engineering for selective proteomics. Curr Opin Chem Biol. 60, 10-19 (2021).
- Rich, R. L., Myszka, D. G. Survey of the year 2007 commercial optical biosensor literature. J Mol Recognit. 21 (6), 355-400 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon