Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Ein Acetyl-Click-Chemie-Assay zur Messung der Histon-Acetyltransferase-1-Acetylierung
In diesem Artikel
Zusammenfassung
Schnelle und genaue chemische Assays zum Screening auf bestimmte Inhibitoren sind ein wichtiges Instrument in der Arzneimittelentwicklung. Hier präsentieren wir einen skalierbaren Acetyl-Click-Chemie-Assay zur Messung der Hemmung der HAT1-Acetylierungsaktivität.
Zusammenfassung
HAT1, auch bekannt als Histon-Acetyltransferase 1, spielt eine entscheidende Rolle bei der Chromatinsynthese, indem es naszierendes H4 vor dem Nukleosomenaufbau stabilisiert und acetyliert. Es wird für das Tumorwachstum in verschiedenen Systemen benötigt, was es zu einem potenziellen Ziel für die Krebsbehandlung macht. Um die Identifizierung von Verbindungen zu erleichtern, die die enzymatische Aktivität von HAT1 hemmen können, haben wir einen Acetyl-Click-Assay für ein schnelles Screening entwickelt. In diesem einfachen Assay verwenden wir rekombinantes HAT1/Rbap46, das aus aktivierten menschlichen Zellen aufgereinigt wird. Das Verfahren nutzt das Acetyl-CoA-Analogon 4-Pentynoyl-CoA (4P) in einem Click-Chemie-Ansatz. Dabei handelt es sich um den enzymatischen Transfer eines Alkingriffs durch eine HAT1-abhängige Acylierungsreaktion auf ein biotinyliertes H4 N-terminales Peptid. Das eingefangene Peptid wird dann auf Neutravidinplatten immobilisiert, gefolgt von einer Click-Chemie-Funktionalisierung mit Biotin-Azid. Anschließend wird Amplexrot durch Streptavidin-Peroxidase-Rekrutierung oxidiert, was zu einer quantitativen Fluoreszenzleistung führt. Durch das Einbringen chemischer Inhibitoren während der Acylierungsreaktion können wir die enzymatische Hemmung basierend auf einer Verringerung des Fluoreszenzsignals quantifizieren. Wichtig ist, dass diese Reaktion skalierbar ist und ein Hochdurchsatz-Screening potenzieller Inhibitoren auf enzymatische Aktivität von HAT1 ermöglicht.
Einleitung
Unter den zahlreichen eukaryotischen Acetyltransferasen war HAT1 die erste Histonacetyltransferase, die isoliert wurde 1,2,3. Nachfolgende Untersuchungen haben seine zentrale Rolle bei der Chromatinreplikation, insbesondere bei der Synthese neuer Nukleosomen während der S-Phase4, fest etabliert. Unsere Forschungsbemühungen führten zu der Erkenntnis, dass HAT1 durch die Behandlung mit dem epidermalen Wachstumsfaktor (EGF) in Brustzellen stark stimuliert wird5. Darüber hinaus hat sich herausgestellt, dass HAT1 für die schnelle Zellproliferation und Tumorbildung in vivo benötigt wird 6,7,8,9. Die Daten deuten darauf hin, dass HAT1 entscheidend für die Koordination anaboler und epigenetischer Prozesse für die Zellteilung ist und das Tumorwachstum vorantreibt.
HAT1 diacetyliert den aminoterminalen Schwanz des Histons H4 an den Lysinen 5 und 12 im Komplex mit dem Chaperonprotein Rbap46, das das Histon bindet und den Aminoterminus an HAT1 präsentiert. Histontetramere oder Disome10 werden dann zusammen mit HAT1/Rbap46 und anderen Histon-Chaperonen11 in den Zellkern importiert. Histone werden dann freigesetzt, um an der Replikationsgabel oder an anderen Stellen abgelagert zu werden, um die Genaktivierung oder -unterdrückung zu unterstützen. Die Funktion der HAT1-Diacetylierungsmarkierung auf Histon H4 ist nicht vollständig verstanden. Es wird wahrscheinlich innerhalb einer Spanne von 15-30 Minuten durch die Wirkung der Histon-Deacetylasen 12,13,14,15 schnell entfernt, nachdem H4 in das Chromatin eingefügt wurde. Daher wird die HAT1-Diacetylierungsmarkierung im Chromatin nicht propagiert und spielt möglicherweise keine echte epigenetische Rolle, obwohl eine Rolle bei der Rekrutierung von Chromatin-modifizierenden Enzymen für naszierendes Chromatin postuliert wurde12. Außerdem acetyliert HAT1 Chromatin nicht direkt; seine Aktivität ist auf lösliche Histone beschränkt.
Die Entwicklung von niedermolekularen Histon-Acetyltransferase-Inhibitoren wurde durch unspezifische Assays und Assays mit niedrigem Durchsatz behindert, was häufig zur Erzeugung biologisch reaktiver Verbindungen führte16,17. Der Goldstandard-Assay zur Messung der Acetyltransferase-Aktivitäten erfordert die Verwendung von 3-H-Acetyl-coA, was den Durchsatz begrenzt und Strahlung erfordert. Nichtsdestotrotz wurden kürzlich spezifische und hochwirksame niedermolekulare Acetyltransferase-Inhibitoren beschrieben, die auf CBP/p30018 und KAT6A/B19,20 abzielen und durch die Verwendung von 3-H-Acetyl-CoA bestätigt wurden. In Zukunft werden verbesserte Assays entwickelt, um einen besseren Durchsatz zu erzielen und Laborgefahren zu vermeiden.
Jüngste Fortschritte in der Acetylierungsüberwachung21 haben die Click-Chemie verwendet, um die Überwachung enzymatischer Reaktionen zu ermöglichen. Es gibt eine Vielzahl von klickfähigen Vorläufern, die über einfache Synthesewege zugänglich oder käuflich zu erwerben sind und in Enzymreaktionen eingebaut werden können. Diese Reaktionen werden typischerweise in rekombinanten Systemen durchgeführt, obwohl auch zellbasierte Assays möglich sind22. Der Vorteil von klickfähigen Co-Faktoren und Substraten besteht darin, dass das Screening die Enzymaktivität direkt messen kann, ohne dass gekoppelte Auslesesysteme erforderlich sind, die oft durch das Screening von Verbindungen gestört werden und zusätzliche Handhabungsschritte erfordern. Dies ermöglicht Inhibitorbehandlungen nur während des enzymatischen Schritts, während alle nachgeschalteten Funktionalisierungs- und Detektionsschritte nach umfangreichem Waschen durchgeführt werden, um Verbindungen zu entfernen, wodurch das Potenzial für Assay-Interferenzen begrenzt wird. Diese Vorteile machen das Design von klickfähigen Assays gegenüber gekoppelten Assays vorzuziehen, die üblicherweise auf dem Nachweis von freiem Coenzym A beruhen.
Eine wichtige Überlegung ist die Akzeptanz von klickfähigen Co-Faktoren im aktiven Zentrum des Enzyms. Vorhandene Co-Faktoren mit aktivierten Klicks sind möglicherweise nicht vollständig mit der aktiven Website kompatibel, die für den nativen Co-Faktor optimiert ist. Strukturelle Informationen und Modellierung können verwendet werden, um Aminosäuresubstitutionen zu entwerfen, um das aktive Zentrum zu vergrößern und veränderte Substrate aufzunehmen23. Dies kann ein Screening mit verbesserter Enzymkinetik und niedrigeren Substrat- und Enzymspiegeln ermöglichen. Der Nachteil dieses Ansatzes besteht darin, dass veränderte katalytische Taschen möglicherweise keine Inhibitoren identifizieren, die stark mit dem nativen Enzym interagieren. Letztendlich ist eine Kombination von Ansätzen erforderlich, um potenzielle Enzyminhibitoren zu identifizieren und zu validieren.
Hier beschreiben wir eine Methode, die entwickelt wurde, um die HAT1-Enzymaktivität mit dem Click-Co-Faktor 4-Pentynoyl-CoA24 zu reinigen und zu testen. Dieser Assay (Abbildung 1) verwendet die native Enzymsequenz im Komplex mit dem erforderlichen Partnerprotein Rbap46, das nachweislich die Enzymaktivität steigert. Die Reinigung des Enzyms aus menschlichen Zellen ermöglicht eine Enzymaktivierung in Cellulo, wodurch stimulierende posttranslationale Modifikationen erhalten bleiben können, die für die volle Enzymaktivität wichtig sind. Das Design und die Optimierung rekombinanter Enzymassays für chemische Hochdurchsatz-Screenings wurden erfolgreich zur Identifizierung und Charakterisierung von niedermolekularen HAT1-Inhibitoren eingesetzt.
Protokoll
1. Methode 1: Herstellung und Aufreinigung des rekombinanten HAT1/Rbap46-Komplexes
- Auftauen, Wiederherstellen und Expandieren von HEK293f-Zellen
- Auftauen von 1-10 Millionen HEK293f-Säugetierzellen26 in 30-ml-Freestyle-293-Expressionsmedien in einem 100-ml-Kolben. Inkubieren Sie in 8 % CO2 bei 37 °C und drehen Sie bei 60 U/min.
- Zählen und überprüfen Sie am nächsten Tag die Lebensfähigkeit der Zellen und stellen Sie dann die Rotationsgeschwindigkeit auf 120 U/min ein. In einem 1-Liter-Kolben auf 300 ml Kultur erweitern, die Aussaatdichte bei 500.000 Zellen/ml beibehalten und die Zellen teilen, bevor die Dichte 3 x 106 Zellen/ml überschreitet.
- HEK293f-Transfektion
- 5 x 105 Zellen/ml in 300-ml-Kultur in einem 1-Liter-Kolben aussäen und 24 h lang kultivieren. Zählen Sie am nächsten Tag die Zellen, um sicherzustellen, dass sie zwischen 7,5 x 105 und 1,2 x 106 Zellen/ml liegen.
- Bereiten Sie eine Mischung aus 300 μg pHEK-FLAG-HAT1 und 300 μg pHEK-Rbap46-Plasmid-DNA in 30 ml PBS vor. Fügen Sie 1,2 ml Polyethylenimin (PEI) zur DNA/PBS hinzu und mischen Sie. 20 Minuten bei RT inkubieren. Die DNA/PBS/PEI-Mischung in die 300-ml-Kultur geben und 48 Stunden bei 37 °C bei 120 U/min inkubieren.
- Zellen ernten
- Zählen Sie die Zellen, um sicherzustellen, dass sie zwischen 1,7 x 106 und 2,5 x 106 Zellen/ml liegen. Bei dieser Zelldichte Forskolin bis zu einer Endkonzentration von 12,5 μM zugeben, um HAT1 zu aktivieren und die Zellen 30 min, 37 °C, 120 U/min zu inkubieren.
- Pelletieren Sie die Zellen 5 Minuten lang bei 300 x g , waschen Sie sie einmal mit 30 ml PBS und frieren Sie sie in flüssigem N2 ein. Bis zur Proteinreinigung bei -80 °C lagern oder direkt mit der Reinigung fortfahren.
- HAT1/Rbap46 komplexe FLAG-Bead-Aufreinigung
- Zellen lysieren und Proteinextrakt herstellen.
- Bereiten Sie den Lysepuffer vor, indem Sie eine Tablette Proteaseinhibitor-Cocktail (PIC) zu 50 ml RSB-500 (20 mM Tris-HCl pH 7,5, 500 mM NaCl, 25 mM MgCl2) geben.
- Das Zellpellet aus 300 ml Kultur auf Eis auftauen und mit 40 ml eiskaltem Lysepuffer mit 0,1 % Triton X-100 lysieren. Auf Eis beschallen, dann 10 Minuten lang bei 4 °C bei 10.000 x g herunterschleudern. Sammeln Sie den gesamten Überstand in einem 50-ml-Röhrchen auf Eis, das jetzt der Proteinextrakt ist.
- Bereiten Sie FLAG-Perlen vor.
- Teilen Sie die FLAG-Beads in vier konische 15-ml-Röhrchen auf, indem Sie 400 μl M2-FLAG-Agarose-Bead-Aufschlämmung in jedes Röhrchen mit 5 ml 0,1 M Glycin pH 3,5, 0,01 % Triton X-100 geben. Schütteln Sie jedes Röhrchen 5 s lang kräftig mit der Hand.
- Pulszentrifuge für 30 s bei 1000 x g bei 4 °C, Überstand ansaugen, zweimal mit 5 ml RSB-500 + 0,1 % Triton X-100 (Tx-100; kein Proteaseinhibitor) waschen und auf Eis inkubieren.
- Führen Sie eine FLAG-Immunpräzipitation durch.
- Geben Sie 10 ml Proteinextrakt in jedes konische 15-ml-Röhrchen mit gewaschenen Kügelchen. Inkubieren Sie die Röhrchen bei 4 °C mit umgekehrter Rotation für mindestens 90 min oder über Nacht.
- Waschen Sie die gebundenen Perlen 5x in 10 ml RSB-500 + 0,1% Tx-100. Für jede Wäsche das Röhrchen 2x-5x umdrehen, um die Perlen zu resuspendieren, dann 1 Minute bei 4 °C bei 1000 x g pelletieren. Zum Schluss 1x in RSB-100 (20 mM Tris-HCl pH 7,5, 100 mM NaCl, 25 mM MgCl2) + 0,1% Tx-100 waschen.
- FLAG-Peptid-Elution: Eluieren Sie den HAT1-Komplex in 1,5 ml Elutionspuffer (EB), der 0,5 mg/ml FLAG-Peptid enthält. Bei 4 °C mindestens 1 h oder über Nacht inkubieren. Die eluierten Kügelchen werden bei 1000 x g für 30 s bei 4 °C pulszentrifugiert und der Überstand gesammelt, der den gereinigten HAT1/Rbap46-Komplex enthält.
- Konzentrieren Sie Protein und entfernen Sie das FLAG-Peptid.
- Geben Sie Eluat in ein 20 ml, 10.000 Dalton Cutoff-Filterröhrchen. Bringen Sie das Volumen mit EB auf 15 ml. Bei 2500 x g für 15 min bei 4 °C schleudern und jedes Mal zweimal mit zusätzlichen 15 ml EB wiederholen.
- Das endgültige Eluat wird aus dem Filterrohr gewonnen und die Proteinkonzentration mit A260 überprüft. Erwarten Sie eine Konzentration von 1 mg Gesamtprotein in 6 ml EB (pro 300 ml Startkultur). Überprüfen Sie die Proteinreinheit durch SDS-PAGE, Coomassie und Immunblotting und lagern Sie Aliquots bei -80 °C.
- Zellen lysieren und Proteinextrakt herstellen.
2. Methode 2: HAT1-Acetyl-Klick-Standardkurve
- Standardkurve vorbereiten
- Synthetisieren Sie ein H4-N-terminales Peptid mit positiver Kontrolle mit der Sequenz: SCRG[Pra]GGKGLG[Pra]GGAKRHRKVLRGG[Lys(Biotin)], wobei [Pra] Propargylglycin bezeichnet.
- Resuspendieren Sie das Pra-Peptid auf 0,1 mg/ml in DMSO. Mischen Sie das Pra-Peptid mit biotinyliertem Histon-H4-Peptid (1-23-GGK-Biotin), um eine Standardkurve zu erstellen (Abbildung 2; Volumina in Tabelle 1). Standardkurven können vor dem Binden der Platte frisch hergestellt oder, falls im Voraus erstellt, mit 20 μl 8 M Harnstoff gemischt und bis Schritt 3.3 bei -20 °C gelagert werden.
3. Methode 3: HAT1-Acetyl-Click-Assay
- Assemblierung von Acetylierungsreaktionen mit Testinhibitoren
- Assemblieren Sie Acetylierungsreaktionen in doppelter Ausführung in 0,2-ml-PCR-Röhrchen oder 96-Well-PCR-Platten aus den folgenden Komponenten: biotinyliertes Histon-H4-Peptid (1-23-GGK-Biotin), resuspendiert in DMSO auf 0,1 mg/ml (34,8 μM), HAT1-Enzym in EB vorverdünnt, 20x-Puffer (1 M Tris pH 8,5, 0,1 % NP40), 2 mM DTT, 4-Pentynoyl-CoA gelöst in Wasser auf 1 mg/ml (1 mM). Eine 20-μl-Reaktion umfasst 10 μl Enzym, 1 μl H4-Peptid, 1 μl 20x-Puffer, 1 μl DTT, vorgemischt und in Vertiefungen einer 96-Well-PCR-Platte auf Eis aliquotiert.
- Pro Vertiefung wird 1 μl DMSO (Negativkontrolle) oder eine bei 1-10 μM gelöste Testverbindung oder H4K12CoA25 (oder ein geeigneter Positivkontrollinhibitor) zugegeben. Durch sanftes Pipettieren mischen und 10 Minuten auf Eis inkubieren, damit sich Enzym-Inhibitor-Komplexe bilden können.
- Fortsetzung der Acetylierungsreaktion
- Kombinieren Sie 2 μl 4-Pentynoyl-CoA mit 4 μl Wasser und geben Sie es dann in die Vertiefungen. Durch Pipettieren vorsichtig mischen, bei 300 x g für 30 s bei RT zentrifugieren, um den Inhalt aufzufangen, und 1 h bei 37 °C in verschlossenen Röhrchen oder Platten mit wiederverschließbarer Folie inkubieren.
- Verarbeiten Sie den Inhalt direkt für Reaktionsprodukte oder schrecken Sie ihn mit 20 μL 8 M Harnstoff ab und lagern Sie ihn bis zur Verarbeitung bei -20 °C.
- Peptidbindung an Neutravidin-Platte
- Geben Sie Reaktionsinhalte mit oder ohne Harnstoff zu Rinderserumalbumin (BSA) vorblockierten schwarzen Neutravidin-beschichteten 96-Well-Platten mit 80 μl PBST (PBS + 0,1 % Tween-20) pro Well.
- Fügen Sie Standardkurvenpeptide in doppelter Ausführung in ihre eigenen Vertiefungen hinzu, die 80 μl PBST enthalten. Binden Sie Peptide mit sanftem Orbitalschütteln für 1 h bei Raumtemperatur (RT).
- Waschen: Nach Abschluss der Bindung die Flüssigkeit aus den Wells ansaugen und die Wells mit 200 μl PBST 3x 15 Hübe (180 μl Hubvolumen) mit einer Plattenscheibe waschen.
- Click-Chemie-Reaktion
- Bereiten Sie die Reagenzien für die Click-Reaktion wie folgt vor: 100 mM Tris(3-hydroxypropyltriazolymethyl)amin (THPTA)-Ligand in Wasser und 20 mM CuSO4 in Wasser. THPTA verhindert die Cu(II)-katalysierte Hydrolyse und löscht Radikale und Peroxide, die ausO2/Cu/Ascorbat erzeugt werden.
- Geben Sie das THPTA/Cu-Gemisch zu 300 mM Natriumascorbat in Wasser und 2,5 mM Biotinazid in DMSO. Die Ein-Klick-Reaktion enthält 140 μl PBS, 10 μl THPTA, 10 μl CuSO4, 10 μl Natriumascorbat, 20 μl Biotinazid. Mischen Sie die Click-Reagenzien immer frisch, geben Sie dann 190 μl in jede Vertiefung, verschließen Sie die Platte und inkubieren Sie sie 1 h lang bei 37 °C.
- Waschen: Saugen Sie die Flüssigkeit aus den Wells an und waschen Sie die Platte 3x mit PBST (180 μL Hubvolumen, 15 Hübe für jeden Waschgang).
- Streptavidin-Meerrettich-Peroxidase-Bindung: Verdünnen Sie Streptavidin-HRP (0,224 mg/ml) 1:10 in Streptavidin (0,224 mg/ml) und verdünnen Sie es dann weiter 1:1000 in PBST. Steptavidin-HRP: Streptavidin-Mischung (100 μl pro Well) zugeben und 1 h bei RT unter leichtem Orbitalschütteln inkubieren.
- 3x mit PBST waschen wie in den vorherigen Schritten.
- Amplex rote Oxidation
- Kombinieren Sie die Amplex-Rot-Detektionsreagenzien wie folgt: 4,45 ml NaHPO4-Puffer (1x = 50 mM Endkonzentration), 50 μl Amplexrot (20 mM in DMSO verdünnt), 500 μl verdünntes H2O2.
- Verdünnen Sie H2O2 von 30 % Stamm auf 3 % in 1x NaHPO4 Puffer und fügen Sie dann 22,7 μl 3 % H2O2 in 977 μl 1x NaHPO4 Puffer hinzu (dies ist das H2O2 , das für die Amplex-Reaktion verwendet wird). 100 μl des amplex red Reaktionsgemisches pro Well zugeben, 30 min lichtgeschützt bei RT inkubieren und dann die Fluoreszenzanregung/-emission 571/585 nm mit einem Standard-Fluoreszenzplatten-Reader detektieren.
- Berechnung der Enzymhemmung: Berechnen Sie die prozentuale Hemmung (Abbildung 3) nach der folgenden Formel:
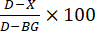 , wobei D der Fluoreszenzwert von Kontrollreaktionen ist, die nur mit DMSO behandelt wurden, X der Wert von Reaktionen, die mit Testverbindungen behandelt wurden, und BG der Wert der Hintergrundvertiefungen (H4-Peptidkontrolle, kein Enzym hinzugefügt).
, wobei D der Fluoreszenzwert von Kontrollreaktionen ist, die nur mit DMSO behandelt wurden, X der Wert von Reaktionen, die mit Testverbindungen behandelt wurden, und BG der Wert der Hintergrundvertiefungen (H4-Peptidkontrolle, kein Enzym hinzugefügt). - Dosiskurve: Wählen Sie Testverbindungen aus, die die HAT1-Enzymaktivität für Wiederholungstests hemmen, wobei die Verbindung seriell verdünnt wird. Bestimmen Sie die Verdünnungen der Verbindung empirisch. Beginnen Sie beispielsweise mit einer Stammkonzentration von 2 mM und verdünnen Sie sie dann seriell 1:3 für acht Verdünnungen. Dann 1:20 zu Enzymassays verdünnen, die eine Spitzendosis von 100 μM ergeben.
- Zeichnen der Kurve: Verwenden Sie die Regression der kleinsten Quadrate, um Dosis-Wirkungs-Hemmkurven (unter Verwendung von prozentualen Hemmwerten aus Schritt 3.10) in der Datenanalysesoftware (GraphPad Prism) anzupassen und IC50-Werte für jede Verbindung abzuleiten (Abbildung 4).
- Acetylierungsreaktion mit Acetyl-CoA:
- Verwenden Sie dies, um die Enzymaktivität mit dem nativen Co-Faktor Acetyl-CoA anstelle von 4-Pentynoyl-CoA zu bestätigen. HAT1-Acetylierungsassays wie in den Schritten 3.1-3.2 beschrieben mit Acetyl-CoA anstelle von 4-Pentynoyl-CoA durchführen.
- Die Reaktionsprodukte werden auf Nitrozellulosemembranen (1-2 μl) aufgebracht, trocknen gelassen und dann mit Anti-H4-Lysin-12-Acetyl- oder Anti-H4-Lysin-5-Acetyl-Antikörpern getupft. Quantifizieren Sie das Immunoblot-Signal durch Densitometrie.
Ergebnisse
Standardkurven in doppelter Ausführung (16 Wells) sollten auf jeder Platte enthalten sein, um eine ordnungsgemäße Testleistung zu gewährleisten. Die Standardkurvendaten sollten in Tabellenform mit einem Bereich von 100 % bis 0 % entsprechend dem Verhältnis von Pra-haltigem Peptid zu nativem H4-Peptid in Lösung erstellt werden (Tabelle 1). Das rote Signal von Amplex ist das höchste in 100 % pra/0 % nativen H4-Peptid-Wells und das niedrigste in 0 % pra/100 % nativen H4-Peptid-Wells. Nachdem die Fluo...
Diskussion
In den letzten zehn Jahren wurde die Klickchemie prominent20 und ermöglichte das präzise Design wechselwirkender chemischer Strukturen. In diesem Zusammenhang haben sich verschiedene bioorthogonale kovalente Verbindungen21 als vielversprechende Optionen für die Bildung von Komplexen in ihrer natürlichen Umgebung herausgestellt. Die Click-Chemie verwendet Paare funktioneller Gruppen, die schnelle und selektive Reaktionen zeigen, die allgemein als "Click-Reaktionen" bekan...
Offenlegungen
Wir haben Patente angemeldet, die den HAT1-Acetyl-Click-Assay (PCT/US20/29395) beschreiben. J.J.G. berichtet über Beratungsverträge mit Sharma Therapeutics, LLC und Guidepoint sowie Forschungsunterstützung (für seine Institution) von Hummingbird Biosciences.
Danksagungen
Wir danken George Zheng für die Bereitstellung von H4K12CoA. Wir danken den Mitgliedern des Gruber Lab für hilfreiche Diskussionen und Feedback. Wir danken der Unterstützung durch das NIH/NCI (1K08CA245024), CPRIT (RR200090) und die V Foundation (V2022-022).
Materialien
| Name | Company | Catalog Number | Comments |
| 4P CoA | Cayman Chemical | 10547 | Click chemistry co-factor |
| Amplex Red | Fisher Sci | A12222 | Fluorescence substrate |
| Biotin-PEG-Azide | Alfa Aesar | J64996MC | Click chemistry |
| Copper Sulfate | Sigma-aldrich | 7758-98-7 | Click chemistry |
| DMSO | Fisher Scientific | 67-68-5 | diluent |
| DTT | Acros Organics | 03-12-3483 | reducting agent |
| Forskolin | VWR | 102987-310 | Protein expression |
| Freestyle 293 Expression Medium | Thermo Fisher | 12338018 | Media |
| Freestyle 293-F cells | Thermo Fisher | R790-07 | Protein expression |
| H4-peptide/1-23-GGK-biotin | Anaspec | AS65097 | peptide substrate |
| HEPES | Sigma-aldrich | 7365-45-9 | EB buffer |
| Hydrogen peroxide 30% solution | Sigma-aldrich | Z00183-99-0 | initiator |
| M2 FLAG antibody slurry | Millipore-Sigma | A2220 | Protein purification |
| Macrosep 10K Filter (Pall Lab) | VWR | 89131-980 | Protein purification |
| Neutravidin Plate | Thermo Sci | 15127 | BSA-pre-blocked |
| NP40 (IGEPAL) | MP Biomedical | 198596 | 20x buffer |
| pHEK-293 plasmid | Takara Bio | 3390 | Protein expression |
| Phosphate Buffered Saline 10x | Alfa Aesar | Z00082-33-6 | wash buffer |
| Pra peptide | Genscript | Custom synthesis | biotinylated |
| Sodium Ascorbate | Sigma-aldrich | 134-03-2 | Click chemistry |
| Sodium chloride | Sigma-aldrich | 7647-14-5 | EB buffer |
| Sodium phosphate | VWR International | 7558-80-7 | buffer |
| Streptavidin | EMD Millipore | 189730 | competitor |
| Streptavidin-HRP | Cell Signaling | 3999S | enzyme |
| THPTA ligand | Fisher Sci | 1010-500 | Click chemistry |
| Tris base | Sigma-aldrich | 77-86-1 | 20x buffer |
| Triton-X 100 | VWR International | 9002-93-1 | EB buffer |
| Tween-20 | Sigma-aldrich | 9005-64-5 | Wash buffer |
| Urea | Sigma-Aldrich | 57-13-6 | quencher |
Referenzen
- Kleff, S., et al. Identification of a gene encoding a yeast histone H4 acetyltransferase. J Biol Chem. 270 (42), 24674-24677 (1995).
- Parthun, M. R., Widom, J., Gottschling, D. E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism. Cell. 87 (1), 85-94 (1996).
- Verreault, A., et al. Nucleosomal DNA regulates the core-histone-binding subunit of the human Hat1 acetyltransferase. Curr Biol. 8 (2), 96-108 (1998).
- Parthun, M. R. Histone acetyltransferase 1: more than just an enzyme. Biochim Biophys Acta. 1819 (3-4), 256-263 (2013).
- Gruber, J. J., et al. HAT1 coordinates histone production and acetylation via H4 promoter binding. Mol Cell. 75 (4), 711-724 e5 (2019).
- Fan, P., et al. Overexpressed histone acetyltransferase 1 regulates cancer immunity by increasing programmed death-ligand 1 expression in pancreatic cancer. J Exp Clin Cancer Res. 38 (1), 47 (2019).
- Xia, P., et al. MicroRNA-377 exerts a potent suppressive role in osteosarcoma through the involvement of the histone acetyltransferase 1-mediated Wnt axis. J Cell Physiol. 234 (12), 22787-22798 (2019).
- Yang, G., et al. Histone acetyltransferase 1 is a succinyltransferase for histones and non-histones and promotes tumorigenesis. EMBO Rep. 22 (2), e50967 (2021).
- Xue, L., et al. RNAi screening identifies HAT1 as a potential drug target in esophageal squamous cell carcinoma. Int J Clin Exp Pathol. 7 (7), 3898-3907 (2014).
- Zhang, W., et al. Structural plasticity of histones H3-H4 facilitates their allosteric exchange between RbAp48 and ASF1. Nat Struct Mol Biol. 20 (1), 29-35 (2013).
- Campos, E. I., et al. The program for processing newly synthesized histones H3.1 and H4. Nat Struct Mol Biol. 17 (11), 1343-1351 (2010).
- Agudelo Garcia, P. A., et al. Identification of multiple roles for histone acetyltransferase 1 in replication-coupled chromatin assembly. Nucleic Acids Res. 45 (16), 9319-9335 (2017).
- Nagarajan, P., et al. Histone acetyl transferase 1 is essential for mammalian development, genome stability, and the processing of newly synthesized histones H3 and H4. PLoS Genet. 9 (6), e1003518 (2013).
- Annunziato, A. T. Assembling chromatin: the long and winding road. Biochim Biophys Acta. 1819 (3-4), 196-210 (2013).
- Annunziato, A. T., Seale, R. L. Histone deacetylation is required for the maturation of newly replicated chromatin. J Biol Chem. 258 (20), 12675-12684 (1983).
- Dahlin, J. L., et al. Assay interference and off-target liabilities of reported histone acetyltransferase inhibitors. Nat Commun. 8 (1), 1527 (2017).
- Baell, J. B., Miao, W. Histone acetyltransferase inhibitors: where art thou. Future Med Chem. 8 (13), 1525-1528 (2016).
- Lasko, L. M., et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours. Nature. 550 (7674), 128-132 (2017).
- Baell, J. B., et al. Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. Nature. 560 (7717), 253-257 (2018).
- Falk, H., et al. An efficient high-throughput screening method for MYST family acetyltransferases, a new class of epigenetic drug targets. J Biomol Screen. 16 (10), 1196-1205 (2011).
- He, M., et al. Chemical biology approaches for investigating the functions of lysine acetyltransferases. Angew Chem Int Ed Engl. 57 (5), 1162-1184 (2018).
- Lipchik, A. M., et al. A peptide-based biosensor assay to detect intracellular Syk kinase activation and inhibition. Biochemistry. 51 (38), 7515-7524 (2012).
- Song, J., et al. Chemoproteomic profiling of protein substrates of a major lysine acetyltransferase in the native cellular context. ACS Chem Biol. 17 (5), 1092-1102 (2022).
- Gaddameedi, J. D., et al. Acetyl-click screening platform identifies small-molecule inhibitors of histone acetyltransferase 1 (HAT1). J Med Chem. 66 (8), 5774-5801 (2023).
- Ngo, L., Brown, T., Zheng, Y. G. Bisubstrate inhibitors to target histone acetyltransferase 1 (HAT1). Chem Biol Drug Des. 93 (5), 865-873 (2019).
- Parker, C. G., Pratt, M. R. Click chemistry in proteomic investigations. Cell. 180 (4), 605-632 (2020).
- Islam, K. The bump-and-hole tactic: Expanding the scope of chemical genetics. Cell Chem Biol. 25 (10), 1171-1184 (2018).
- Radziwon, K., Weeks, A. M. Protein engineering for selective proteomics. Curr Opin Chem Biol. 60, 10-19 (2021).
- Rich, R. L., Myszka, D. G. Survey of the year 2007 commercial optical biosensor literature. J Mol Recognit. 21 (6), 355-400 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten