JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
히스톤 아세틸전이효소 1 아세틸화를 측정하기 위한 아세틸 클릭 화학 분석
요약
특정 억제제를 스크리닝하기 위한 빠르고 정확한 화학 분석은 약물 개발 무기고에서 중요한 도구입니다. 여기에서는 HAT1 아세틸화 활성의 억제를 측정하기 위한 확장 가능한 아세틸 클릭 화학 분석을 제시합니다.
초록
히스톤 아세틸전이효소 1(Histone acetyltransferase 1)이라고도 하는 HAT1은 뉴클레오솜 조립 전에 초기 H4를 안정화하고 아세틸화하여 염색질 합성에 중요한 역할을 합니다. 다양한 시스템에서 종양 성장에 필요하므로 암 치료의 잠재적 표적이 될 수 있습니다. HAT1 효소 활성을 억제할 수 있는 화합물의 식별을 용이하게 하기 위해 당사는 신속한 스크리닝을 위한 아세틸 클릭 분석을 고안했습니다. 이 간단한 분석에서는 활성화된 인간 세포에서 정제된 재조합 HAT1/Rbap46을 사용합니다. 이 방법은 클릭 화학 접근 방식에서 아세틸-CoA 아날로그 4-펜티노일-CoA(4P)를 활용합니다. 여기에는 HAT1 의존성 아실화 반응을 통해 비오틴화된 H4 N-말단 펩타이드로 알카인 핸들의 효소 전달이 포함됩니다. 그런 다음 포획된 펩타이드를 뉴트라비딘 플레이트에 고정시킨 후 비오틴-아지드로 클릭 화학 기능화를 수행합니다. 그 후, 스트렙타비딘-퍼옥시다아제 모집을 사용하여 amplex red를 산화시켜 정량적 형광 출력을 생성합니다. 아실화 반응 중에 화학적 억제제를 도입하면 형광 신호의 감소를 기반으로 효소 억제를 정량화할 수 있습니다. 중요한 것은 이 반응이 확장 가능하여 HAT1 효소 활성에 대한 잠재적 억제제의 고처리량 스크리닝이 가능하다는 것입니다.
서문
수많은 진핵 아세틸 전이 효소 중에서, HAT1은 1,2,3 분리 될 초기 히스톤 아세틸 전이 효소였습니다. 후속 연구는 염색질 복제, 특히 S상4단계 동안 새로운 뉴클레오솜 합성에서 중추적인 역할을 확고하게 확립했습니다. 우리의 연구 노력은 HAT1이 유방 세포의 표피 성장 인자(EGF) 치료에 의해 매우 자극된다는 것을 인식하게 했다5. 또한, HAT1은 생체 내 빠른 세포 증식 및 종양 형성에 필요하다는 것이 밝혀졌습니다 6,7,8,9. 데이터에 따르면 HAT1은 세포 분열을 위한 동화 작용 및 후성유전학적 과정을 조정하여 종양 성장을 촉진하는 데 중요한 역할을 합니다.
HAT1은 히스톤을 결합하고 아미노 말단을 HAT1에 제시하는 샤페론 단백질 Rbap46과 복합체로 라이신 5 및 12에서 히스톤 H4의 아미노 말단 꼬리를 디 아세틸화합니다. 히스톤 사량체 또는 디솜(10)은 HAT1/Rbap46 및 다른 히스톤 샤페론(11)과 함께 핵으로 수입된다. 그런 다음 히스톤은 복제 포크 또는 유전자 활성화 또는 억제를 지원하기 위해 다른 부위에 기탁되도록 방출됩니다. 히스톤 H4에 대한 HAT1 디 아세틸화 표시의 기능은 완전히 이해되지 않았습니다. H15가 염색질에 삽입 된 후 히스톤 탈아 세틸 화제 12,13,14,15의 작용에 의해 30-4 분 이내에 빠르게 제거 될 가능성이 높습니다. 따라서, HAT1 디-아세틸화 표시는 염색질에서 증식되지 않으며, 초기 염색질에 대한 염색질 변형 효소의 모집에 대한 역할이 가정되었지만, 진정한 후성유전학적 역할을 하지 않을 수 있다12. 또한 HAT1은 염색질을 직접 아세틸화하지 않습니다. 그 활성은 용해성 히스톤으로 제한됩니다.
저분자 히스톤 아세틸전이효소 억제제의 개발은 비특이적이고 처리량이 낮은 분석에 의해 방해를 받아 종종 생물학적 반응성 화합물16,17의 생성을 초래합니다. 아세틸전이효소 활성을 측정하기 위한 골드 스탠다드 분석에는 처리량이 제한되고 방사선이 필요한 3H-아세틸-coA를 사용해야 합니다. 그럼에도 불구하고, 최근에는 CBP/p30018 및 KAT6A/B19,20을 표적으로 하는 특이적이고 매우 강력한 저분자 아세틸전이효소 억제제가 3 H-아세틸-CoA의 사용을 통해 기술되고 확인되었습니다. 앞으로 더 나은 처리량을 달성하고 실험실 위험을 피하기 위한 개선된 분석법이 고안되고 있습니다.
아세틸화 모니터링(acetylation monitoring)21 의 최근 발전은 효소 반응 모니터링을 가능하게 하기 위해 클릭 화학(click chemistry)을 사용하였다. 간단한 합성 경로로 접근하거나 효소 반응에 통합할 수 있는 구매가 가능한 다양한 클릭 기반 전구체가 있습니다. 이러한 반응은 전형적으로 재조합 시스템에서 수행되지만, 세포 기반 분석도 가능하다22. 클릭 가능 보조 인자 및 기질의 장점은 스크리닝이 종종 스크리닝 화합물에 의해 교란되고 추가 처리 단계가 필요한 결합된 판독 시스템 없이 효소 활성을 직접 측정할 수 있다는 것입니다. 이를 통해 효소 단계에서만 억제제 처리가 가능한 반면, 모든 다운스트림 기능화 및 검출 단계는 화합물을 제거하기 위해 광범위한 세척 후에 수행되므로 분석 간섭이 발생할 가능성이 제한됩니다. 이러한 장점으로 인해 클릭 기반 분석의 설계는 일반적으로 유리 코엔자임 A의 검출에 의존하는 결합 분석보다 선호됩니다.
한 가지 중요한 고려 사항은 클릭 가능 보조 인자를 효소 활성 부위에 수용하는 것입니다. 기존의 클릭 지원 보조 요인은 기본 보조 요인에 최적화된 활성 사이트와 완전히 호환되지 않을 수 있습니다. 구조 정보 및 모델링은 변형된 기질(23)을 통합하기 위해 활성 부위를 확대하기 위한 아미노산 치환을 설계하는데 사용될 수 있다. 이를 통해 향상된 효소 역학과 더 낮은 기질 및 효소 수준으로 스크리닝할 수 있습니다. 이 접근법의 단점은 변형된 촉매 포켓이 네이티브 효소와 강하게 상호 작용하는 억제제를 식별하지 못할 수 있다는 것입니다. 궁극적으로, 잠재적인 효소 억제제를 식별하고 검증하기 위해서는 다양한 접근법의 조합이 필요합니다.
여기에서는 클릭 보조 인자 4-펜티노일-CoA24를 사용하여 HAT1 효소 활성을 정제하고 분석하기 위해 개발된 방법을 설명합니다. 이 분석법(그림 1)은 효소 활성을 높이는 것으로 밝혀진 필수 파트너 단백질 Rbap46과 복합체의 네이티브 효소 서열을 사용합니다. 인간 세포에서 효소를 정제하면 셀룰로에서 효소가 활성화되어 완전한 효소 활성에 중요한 자극적인 번역 후 변형을 보존할 수 있습니다. 고처리량 화학 스크리닝을 위한 재조합 효소 분석의 설계 및 최적화는 HAT1 저분자 억제제를 식별하고 특성화하는 데 성공적으로 사용되었습니다.
프로토콜
1. 방법 1: 재조합 HAT1/Rbap46 복합체 생산 및 정제
- HEK293f 세포 해동, 회수 및 확장
- 1-1,000만 HEK293f 포유류 세포26 개를 100mL 플라스크에서 30mL 자유형 293 발현 배지로 해동합니다. 60 rpm으로 회전하면서 37 °C에서 8 % CO2 에서 배양합니다.
- 다음날 세포 생존율을 계산하고 확인한 다음 회전 속도를 120rpm으로 조정합니다. 1L 플라스크에서 300mL 배양액으로 확장하여 파종 밀도를 500,000 cells/mL로 유지하고 밀도가 3 x 106 cells/mL를 초과하기 전에 세포를 분할합니다.
- HEK293f 트랜스펙션
- 1L 플라스크에 300mL 배양액에 5 x 105 세포/mL를 파종하고 24시간 동안 배양합니다. 다음 날, 세포가 7.5 x 105 에서 1.2 x 106 cells/mL 사이인지 확인하기 위해 세포를 세십시오.
- 30mL의 PBS에 300μg의 pHEK-FLAG-HAT1과 300μg의 pHEK-Rbap46 플라스미드 DNA의 혼합물을 준비합니다. 폴리에틸렌민(PEI) 1.2mL를 DNA/PBS에 넣고 섞습니다. RT에서 20분 동안 배양합니다. DNA/PBS/PEI 혼합물을 300mL 배양액에 넣고 37°C에서 120rpm에서 48시간 동안 배양합니다.
- 세포 채취
- 세포가 1.7 x 106 에서 2.5 x 106 세포/mL 사이에 있는지 계산합니다. 이 세포 밀도에서 포스콜린을 12.5μM의 최종 농도에 첨가하여 HAT1을 활성화하고 30분, 37°C, 120rpm 동안 세포를 배양합니다.
- 세포를 300 x g 에서 5분 동안 펠렛화하고 30mL의 PBS로 한 번 세척한 다음 액체 N2에서 급속 냉동합니다. 단백질이 정제될 때까지 -80°C에서 보관하거나 바로 정제를 진행하십시오.
- HAT1/Rbap46 복합체 FLAG-비드 정제
- 세포를 용해하고 단백질 추출물을 준비합니다.
- 50mL의 RSB-500(20mM Tris-HCl pH 7.5, 500mM NaCl, 25mM MgCl2)에 프로테아제 억제제 칵테일(PIC) 1정을 첨가하여 용해 완충액을 준비합니다.
- 300mL 배양액의 세포 펠릿을 얼음 위에서 해동하고 0.1% Triton X-100을 사용하여 40mL의 얼음 저온 용해 완충액으로 용해합니다. 얼음 위에서 초음파 처리 한 다음 10,000 x g 에서 4 ° C에서 10 분 동안 회전시킵니다. 모든 상층액을 얼음 위의 50mL 튜브 하나에 모으면 단백질 추출물이 나옵니다.
- FLAG 비즈를 준비합니다.
- 5mL의 0.1M 글리신 pH 3.5, 0.01% Triton X-100을 포함하는 각 튜브에 400μL의 M2-FLAG 아가로스 비드 슬러리를 추가하여 FLAG 비드를 4개의 15mL 원뿔형 튜브로 분할합니다. 각 튜브를 손으로 5초 동안 세게 흔듭니다.
- 4°C에서 1000 x g 에서 30초 동안 펄스 원심분리기, 상층액을 흡입하고 RSB-500 + 0.1% Triton X-100(Tx-100, 단백질 분해 효소 억제제 없음) 5mL로 2회 세척하고 얼음에서 배양합니다.
- FLAG 면역침전을 수행합니다.
- 세척된 비드가 들어 있는 각 15mL 원뿔형 튜브에 단백질 추출물 10mL를 추가합니다. 튜브를 4°C에서 역회전하여 최소 90분 또는 하룻밤 동안 배양합니다.
- 결합된 비드를 RSB-500 + 0.1% Tx-100 10mL 10mL에 5회 세척합니다. 세척할 때마다 튜브를 2x-5x 뒤집어 비드를 재현탁한 다음 1000 x g 에서 4°C에서 1분 동안 펠릿합니다. 마지막으로 RSB-100 (20mM Tris-HCl pH 7.5, 100mM NaCl, 25mM MgCl2) + 0.1 % Tx-100에서 1x 세척합니다.
- FLAG 펩타이드 용출: 0.5mg/mL FLAG 펩타이드를 함유한 1.5mL의 용출 완충액(EB)에서 HAT1 복합체를 용리합니다. 4 °C에서 최소 1시간 또는 하룻밤 동안 배양합니다. 용리된 비드를 1000 x g 에서 4°C에서 30초 동안 펄스 원심분리하고 정제된 HAT1/Rbap46 복합체를 포함하는 상층액을 수집합니다.
- 단백질을 농축하고 FLAG 펩타이드를 제거합니다.
- 20mL, 10,000Dalton 차단 필터 튜브에 용리액을 추가합니다. EB를 사용하여 부피를 15mL까지 올립니다. 4°C에서 15분 동안 2500 x g 으로 회전하고 매번 15mL의 EB를 추가로 사용하여 두 번 반복합니다.
- 필터 튜브에서 최종 용리액을 회수하고 A260으로 단백질 농도를 확인합니다. EB 6mL에 총 단백질 1mg의 농도가 예상됩니다(시작 배양 300mL 기준). SDS-PAGE, Coomassie, immunoblotting을 통해 단백질 순도를 확인하고 분취액은 -80°C에서 보관합니다.
- 세포를 용해하고 단백질 추출물을 준비합니다.
2. 방법 2: HAT1 아세틸 클릭 표준 곡선
- 표준 곡선 준비
- SCRG[Pra]GGKGLG[Pra]GGAKRHRKVLRGG[Lys(Biotin)] 서열로 양성 대조군 H4 N-말단 펩타이드를 합성합니다.
- Pra 펩타이드를 DMSO에서 0.1mg/mL로 재현탁시킵니다. Pra 펩타이드를 비오틴화된 히스톤 H4 펩타이드(1-23-GGK-비오틴)와 혼합하여 표준 곡선을 만듭니다(그림 2, 표 1에서 찾을 수 있는 부피). 표준 곡선은 플레이트 바인딩 전에 새로 만들거나 미리 만든 경우 20μL의 8M 요소와 혼합하여 3.3단계까지 -20°C에서 보관할 수 있습니다.
3. 방법 3: HAT1 아세틸 클릭 분석
- 테스트 억제제를 사용한 아세틸화 반응 조립
- 다음 구성 요소에서 0.2mL PCR 튜브 또는 96-웰 PCR 플레이트에 아세틸화 반응을 중복으로 조립합니다: DMSO에서 0.1mg/mL(34.8μM)로 재현탁된 비오틴화 히스톤 H4 펩타이드(1-23-GGK-비오틴), EB에 미리 희석된 HAT1 효소, 20배 완충액(1M Tris pH 8.5, 0.1% NP40), 2mM DTT, 물에 1mg/mL(1mM)로 용해된 4-펜티노일-CoA. 20μL 반응은 10μL의 효소, 1μL의 H4 펩타이드, 1μL의 20x 완충액, 1μL의 DTT로 구성되며 사전 혼합되어 얼음 위의 96웰 PCR 플레이트의 웰에 분주됩니다.
- 웰당 1μL의 DMSO(음성 대조군) 또는 1-10μM에 용해된 테스트 화합물 또는 H4K12CoA25 (또는 적절한 양성 대조군 억제제)를 추가합니다. 부드러운 피펫팅으로 혼합하고 얼음 위에서 10분 동안 배양하여 효소: 억제제 복합체가 형성되도록 합니다.
- 아세틸화 반응 연속
- 2μL의 4-펜티노일-CoA를 4μL의 물과 결합한 다음 우물에 추가합니다. 피펫팅으로 부드럽게 혼합하고, 상온에서 300 x g 에서 30초 동안 원심분리하여 내용물을 수집하고, 37°C에서 뚜껑이 있는 튜브 또는 재밀봉 가능한 호일이 있는 플레이트에서 1시간 동안 배양합니다.
- 반응 생성물을 위해 내용물을 직접 처리하거나 20μL의 8M 요소로 담금질하고 처리할 때까지 -20°C에서 보관합니다.
- Neutravidin 플레이트에 대한 펩타이드 결합
- 웰당 80μL의 PBST(PBS + 0.1% Tween-20)를 포함하는 소 혈청 알부민(BSA) 사전 차단된 검은색 뉴트라비딘 코팅 96웰 플레이트에 요소 유무에 관계없이 반응 내용물을 추가합니다.
- 표준 곡선 펩타이드를 80μL의 PBST를 함유한 자체 웰에 중복으로 추가합니다. 실온(RT)에서 1시간 동안 부드러운 궤도 흔들림으로 펩타이드를 결합합니다.
- 세척: 결합이 완료된 후 웰에서 액체를 흡인하고 플레이트 와셔를 사용하여 200μL의 PBST 3x 15스트로크(180μL 스트로크 부피)로 웰을 세척합니다.
- 클릭 화학 반응
- 다음과 같이 클릭 반응을위한 시약을 준비한다 : 100 mM Tris (3- 하이드 록시 프로필 트리아 졸리 메틸) 아민 (THPTA) 리간드 물 및 20 mM CuSO4 물에서. THPTA는 Cu (II) 촉매 가수 분해를 방지하고 O2 / Cu / 아스코르브 산염에서 생성 된 라디칼 및 과산화물을 소멸시킵니다.
- THPTA/Cu 혼합물을 물에 있는 300mM 아스코르브산나트륨에 첨가하고 DMSO에 2.5mM 비오틴 아지드화물을 첨가합니다. 원클릭 반응에는 PBS 140μL, THPTA 10μL, CuSO4 10μL, 아스코르브산나트륨 10μL, 비오틴-아지드 20μL가 포함됩니다. 항상 클릭 시약을 신선하게 혼합한 다음 각 웰에 190μL를 분주하고 플레이트를 밀봉한 다음 37°C에서 1시간 동안 배양합니다.
- 세척: 웰에서 액체를 흡입하고 PBST(180μL 스트로크 부피, 세척 당 15스트로크)로 플레이트를 3회 세척합니다.
- 스트렙타비딘-고추냉이 과산화효소 결합: 스트렙타비딘-HRP(0.224mg/mL)를 스트렙타비딘(0.224mg/mL)에 1:10으로 희석한 다음 PBST에서 1:1000으로 더 희석합니다. Steptavidin-HRP: streptavidin 혼합물(웰당 100μL)을 추가하고 부드러운 궤도 흔들림으로 RT에서 1시간 동안 배양합니다.
- 이전 단계와 같이 PBST로 3번 세척합니다.
- Amplex 적색 산화
- Amplex 적색 검출 시약을 다음과 같이 결합합니다: 4.45mL의 NaHPO4 완충액(1x = 50mM 최종 농도), 50μL amplex 적색(DMSO에 희석된 20mM), 500μL의 희석된H2O2.
- 1x NaHPO4 완충액에H2O2를 30% 스톡에서 3%로 희석한 후, 22.7μL의 3%H2O2를 977μL의 1x NaHPO4 완충액(암플렉스 반응에 사용되는H2O2)에 첨가한다. 웰당 100μL의 amplex 적색 반응 혼합물을 추가하고 빛으로부터 보호된 상태에서 실온에서 30분 동안 배양한 다음 표준 형광 플레이트 리더를 사용하여 571/585nm의 형광 여기/방출을 검출합니다.
- 효소 억제 계산: 다음 공식에 따라 억제율(그림 3)을 계산합니다.
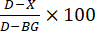 여기서 D 는 DMSO로만 처리한 대조 반응의 형광 값, X 는 테스트 화합물로 처리한 반응의 값, BG 는 배경 웰(H4 펩타이드 대조군, 효소 첨가 없음)의 값입니다.
여기서 D 는 DMSO로만 처리한 대조 반응의 형광 값, X 는 테스트 화합물로 처리한 반응의 값, BG 는 배경 웰(H4 펩타이드 대조군, 효소 첨가 없음)의 값입니다. - 용량 곡선: 화합물을 연속적으로 희석하여 반복 분석을 위해 HAT1 효소 활성을 억제하는 것으로 밝혀진 테스트 화합물을 선택합니다. 화합물 희석액을 경험적으로 측정한다. 예를 들어, 2mM 스톡 농도로 시작한 다음 8회 희석을 위해 1:3으로 연속적으로 희석합니다. 그런 다음 1:20을 효소 분석에 희석하여 100μM 최고 용량을 산출합니다.
- 곡선 그리기: 최소 제곱 회귀를 사용하여 데이터 분석 소프트웨어(GraphPad Prism)에서 용량-반응 억제 곡선(3.10단계의 억제 백분율 값 사용)을 피팅하고 각 화합물에 대한 IC50 값을 도출합니다(그림 4).
- 아세틸-CoA와의 아세틸화 반응:
- 이를 사용하여 4-펜티노일-CoA 대신 네이티브 보조 인자 아세틸-CoA로 효소 활성을 확인합니다. 3.1-3.2단계에서 설명한 대로 4-펜티노일-CoA 대신 아세틸-CoA를 사용하여 HAT4 아세틸화 분석을 수행합니다.
- 반응 생성물을 니트로셀룰로오스 멤브레인(1-2μL)에 놓고 건조시킨 다음 항-H4-라이신-12-아세틸 또는 항-H4-라이신-5-아세틸 항체로 도트 블롯합니다. 밀도 측정법으로 면역블롯 신호를 정량화합니다.
결과
중복된 표준 곡선(16웰)은 적절한 분석 성능을 보장하기 위해 모든 플레이트에 포함되어야 합니다. 표준 곡선 데이터는 용액 내 Pra 함유 펩타이드와 네이티브 H4 펩타이드의 비율에 따라 100%에서 0% 범위의 표 형식으로 설정해야 합니다(표 1). Amplex 적색 신호는 100% pra/0% 천연 H4 펩타이드 웰에서 가장 높고, 0% pra/100% 천연 H4 펩타이드 웰에서 가장 낮습니다. 형광이 검출되고 웰의 평균을 ?...
토론
지난 10년 동안 클릭 화학이 두각을 나타내면서 상호 작용하는 화학 구조의 정밀한 설계가 가능해졌습니다. 이러한 맥락에서, 다양한 생체 직교 공유 연결(bioorthogonal covalent connections)21 은 자연 환경에서 복합체를 형성하기 위한 유망한 옵션으로 등장했다. 클릭 화학은 일반적으로 "클릭 반응"으로 알려진 빠르고 선택적인 반응을 나타내는 작용기 쌍을 사용합니?...
공개
당사는 HAT1 아세틸 클릭 분석법(PCT/US20/29395)을 기술하는 특허를 출원했습니다. J.J.G.는 Sharma Therapeutics, LLC 및 Guidepoint와의 컨설팅 계약 및 Hummingbird Biosciences의 연구 지원(그의 기관)을 보고합니다.
감사의 말
H4K12CoA를 제공해 주신 George Zheng에게 감사드립니다. 유용한 토론과 피드백을 주신 Gruber Lab 구성원 여러분께 감사드립니다. NIH/NCI(1K08CA245024), CPRIT(RR200090) 및 V Foundation(V2022-022)의 지원에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 4P CoA | Cayman Chemical | 10547 | Click chemistry co-factor |
| Amplex Red | Fisher Sci | A12222 | Fluorescence substrate |
| Biotin-PEG-Azide | Alfa Aesar | J64996MC | Click chemistry |
| Copper Sulfate | Sigma-aldrich | 7758-98-7 | Click chemistry |
| DMSO | Fisher Scientific | 67-68-5 | diluent |
| DTT | Acros Organics | 03-12-3483 | reducting agent |
| Forskolin | VWR | 102987-310 | Protein expression |
| Freestyle 293 Expression Medium | Thermo Fisher | 12338018 | Media |
| Freestyle 293-F cells | Thermo Fisher | R790-07 | Protein expression |
| H4-peptide/1-23-GGK-biotin | Anaspec | AS65097 | peptide substrate |
| HEPES | Sigma-aldrich | 7365-45-9 | EB buffer |
| Hydrogen peroxide 30% solution | Sigma-aldrich | Z00183-99-0 | initiator |
| M2 FLAG antibody slurry | Millipore-Sigma | A2220 | Protein purification |
| Macrosep 10K Filter (Pall Lab) | VWR | 89131-980 | Protein purification |
| Neutravidin Plate | Thermo Sci | 15127 | BSA-pre-blocked |
| NP40 (IGEPAL) | MP Biomedical | 198596 | 20x buffer |
| pHEK-293 plasmid | Takara Bio | 3390 | Protein expression |
| Phosphate Buffered Saline 10x | Alfa Aesar | Z00082-33-6 | wash buffer |
| Pra peptide | Genscript | Custom synthesis | biotinylated |
| Sodium Ascorbate | Sigma-aldrich | 134-03-2 | Click chemistry |
| Sodium chloride | Sigma-aldrich | 7647-14-5 | EB buffer |
| Sodium phosphate | VWR International | 7558-80-7 | buffer |
| Streptavidin | EMD Millipore | 189730 | competitor |
| Streptavidin-HRP | Cell Signaling | 3999S | enzyme |
| THPTA ligand | Fisher Sci | 1010-500 | Click chemistry |
| Tris base | Sigma-aldrich | 77-86-1 | 20x buffer |
| Triton-X 100 | VWR International | 9002-93-1 | EB buffer |
| Tween-20 | Sigma-aldrich | 9005-64-5 | Wash buffer |
| Urea | Sigma-Aldrich | 57-13-6 | quencher |
참고문헌
- Kleff, S., et al. Identification of a gene encoding a yeast histone H4 acetyltransferase. J Biol Chem. 270 (42), 24674-24677 (1995).
- Parthun, M. R., Widom, J., Gottschling, D. E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism. Cell. 87 (1), 85-94 (1996).
- Verreault, A., et al. Nucleosomal DNA regulates the core-histone-binding subunit of the human Hat1 acetyltransferase. Curr Biol. 8 (2), 96-108 (1998).
- Parthun, M. R. Histone acetyltransferase 1: more than just an enzyme. Biochim Biophys Acta. 1819 (3-4), 256-263 (2013).
- Gruber, J. J., et al. HAT1 coordinates histone production and acetylation via H4 promoter binding. Mol Cell. 75 (4), 711-724 e5 (2019).
- Fan, P., et al. Overexpressed histone acetyltransferase 1 regulates cancer immunity by increasing programmed death-ligand 1 expression in pancreatic cancer. J Exp Clin Cancer Res. 38 (1), 47 (2019).
- Xia, P., et al. MicroRNA-377 exerts a potent suppressive role in osteosarcoma through the involvement of the histone acetyltransferase 1-mediated Wnt axis. J Cell Physiol. 234 (12), 22787-22798 (2019).
- Yang, G., et al. Histone acetyltransferase 1 is a succinyltransferase for histones and non-histones and promotes tumorigenesis. EMBO Rep. 22 (2), e50967 (2021).
- Xue, L., et al. RNAi screening identifies HAT1 as a potential drug target in esophageal squamous cell carcinoma. Int J Clin Exp Pathol. 7 (7), 3898-3907 (2014).
- Zhang, W., et al. Structural plasticity of histones H3-H4 facilitates their allosteric exchange between RbAp48 and ASF1. Nat Struct Mol Biol. 20 (1), 29-35 (2013).
- Campos, E. I., et al. The program for processing newly synthesized histones H3.1 and H4. Nat Struct Mol Biol. 17 (11), 1343-1351 (2010).
- Agudelo Garcia, P. A., et al. Identification of multiple roles for histone acetyltransferase 1 in replication-coupled chromatin assembly. Nucleic Acids Res. 45 (16), 9319-9335 (2017).
- Nagarajan, P., et al. Histone acetyl transferase 1 is essential for mammalian development, genome stability, and the processing of newly synthesized histones H3 and H4. PLoS Genet. 9 (6), e1003518 (2013).
- Annunziato, A. T. Assembling chromatin: the long and winding road. Biochim Biophys Acta. 1819 (3-4), 196-210 (2013).
- Annunziato, A. T., Seale, R. L. Histone deacetylation is required for the maturation of newly replicated chromatin. J Biol Chem. 258 (20), 12675-12684 (1983).
- Dahlin, J. L., et al. Assay interference and off-target liabilities of reported histone acetyltransferase inhibitors. Nat Commun. 8 (1), 1527 (2017).
- Baell, J. B., Miao, W. Histone acetyltransferase inhibitors: where art thou. Future Med Chem. 8 (13), 1525-1528 (2016).
- Lasko, L. M., et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours. Nature. 550 (7674), 128-132 (2017).
- Baell, J. B., et al. Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. Nature. 560 (7717), 253-257 (2018).
- Falk, H., et al. An efficient high-throughput screening method for MYST family acetyltransferases, a new class of epigenetic drug targets. J Biomol Screen. 16 (10), 1196-1205 (2011).
- He, M., et al. Chemical biology approaches for investigating the functions of lysine acetyltransferases. Angew Chem Int Ed Engl. 57 (5), 1162-1184 (2018).
- Lipchik, A. M., et al. A peptide-based biosensor assay to detect intracellular Syk kinase activation and inhibition. Biochemistry. 51 (38), 7515-7524 (2012).
- Song, J., et al. Chemoproteomic profiling of protein substrates of a major lysine acetyltransferase in the native cellular context. ACS Chem Biol. 17 (5), 1092-1102 (2022).
- Gaddameedi, J. D., et al. Acetyl-click screening platform identifies small-molecule inhibitors of histone acetyltransferase 1 (HAT1). J Med Chem. 66 (8), 5774-5801 (2023).
- Ngo, L., Brown, T., Zheng, Y. G. Bisubstrate inhibitors to target histone acetyltransferase 1 (HAT1). Chem Biol Drug Des. 93 (5), 865-873 (2019).
- Parker, C. G., Pratt, M. R. Click chemistry in proteomic investigations. Cell. 180 (4), 605-632 (2020).
- Islam, K. The bump-and-hole tactic: Expanding the scope of chemical genetics. Cell Chem Biol. 25 (10), 1171-1184 (2018).
- Radziwon, K., Weeks, A. M. Protein engineering for selective proteomics. Curr Opin Chem Biol. 60, 10-19 (2021).
- Rich, R. L., Myszka, D. G. Survey of the year 2007 commercial optical biosensor literature. J Mol Recognit. 21 (6), 355-400 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유