A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
توليد وتحليل صور الأنسجة عالية المعلمات باستخدام قياس التدفق الخلوي
In This Article
Summary
الموصوفة هنا هي طريقة يمكن استخدامها لتصوير خمسة أو أكثر من المعلمات الفلورية بواسطة المجهر المناعي. تم تحديد خط أنابيب تحليل لاستخراج الخلايا المفردة من هذه الصور وإجراء تحليل الخلية الواحدة من خلال استراتيجيات بوابات تشبه قياس التدفق الخلوي ، والتي يمكنها تحديد مجموعات فرعية من الخلايا في أقسام الأنسجة.
Abstract
إن استخدام علم الأنسجة للتحقيق في تنوع الخلايا المناعية في أقسام الأنسجة مثل تلك المشتقة من الجهاز العصبي المركزي (CNS) محدود للغاية بسبب عدد المعلمات الفلورية التي يمكن تصويرها في وقت واحد. تم تعريف معظم المجموعات الفرعية للخلايا المناعية باستخدام قياس التدفق الخلوي باستخدام مجموعات معقدة من علامات البروتين ، وغالبا ما تتطلب أربعة معلمات أو أكثر لتحديدها بشكل قاطع ، وهو ما يتجاوز قدرات معظم المجاهر التقليدية. نظرا لأن قياس التدفق الخلوي يفصل الأنسجة ويفقد المعلومات المكانية ، فهناك حاجة إلى تقنيات يمكنها الاحتفاظ بالمعلومات المكانية أثناء استجواب أدوار أنواع الخلايا المعقدة. تتم معالجة هذه القضايا هنا من خلال إنشاء طريقة لتوسيع عدد المعلمات الفلورية التي يمكن تصويرها عن طريق جمع إشارات الفلوروفورات المتداخلة طيفيا واستخدام عدم الخلط الطيفي لفصل إشارات كل فلوروفور على حدة. ثم تتم معالجة هذه الصور باستخدام خط أنابيب تحليل لالتقاط صور الأنسجة عالية المعلمات واستخراج خلايا مفردة من هذه الصور بحيث يمكن تحليل خصائص الفلورسنت الفريدة لكل خلية على مستوى خلية واحدة. باستخدام استراتيجيات البوابات الشبيهة بقياس التدفق الخلوي ، يمكن بعد ذلك تحديد ملامح الخلايا في مجموعات فرعية وتعيينها مرة أخرى على أقسام الأنسجة ليس فقط لتحديد وفرتها ، ولكن أيضا لتحديد كيفية تفاعلها مع بيئة الأنسجة. بشكل عام ، تم إثبات بساطة وإمكانية استخدام قياس التدفق الخلوي النسيجي لدراسة مجموعات المناعة المعقدة في أقسام الأنسجة.
Introduction
يمكن أن يساهم الالتهاب الناجم عن خلايا الجهاز المناعي والخلايا الدبقية في الاضطرابات المزمنة في الجهاز العصبي المركزي حيث يمكن لكل مجموعة تعزيز نشاط1،2،3 الأخرى. إن فهم كيفية تفاعل الجهاز المناعي مع هذه العناصر من الجهاز العصبي المركزي لتعزيز التهاب الجهاز العصبي المركزي هو حاليا موضوع رئيسي للاهتمام وقد تم تسهيله إلى حد كبير من خلال التقنيات عالية المعلمات مثل تسلسل الحمض النووي الريبي أحادي الخلية. من خلال تسلسل الحمض النووي الريبي أحادي الخلية ، اكتشفنا أن هناك اتصالا مكثفا يحدث بين الخلايا الدبقية والجهاز المناعي في العديد من اضطرابات الجهاز العصبي المركزي4،5،6. إن فهم كيفية تأثير هذه التفاعلات على هذه الاضطرابات سيكون أمرا حاسما لتوضيح بيولوجيا هذه الأمراض.
تتمثل إحدى مشكلات تحليلات تسلسل الخلية الواحدة في أن هذه التقنيات تتطلب تعطيل الأنسجة للحصول على خلايا أو نوى مفردة ، مما يؤدي إلى فقدان كامل للمعلومات المكانية. إن معرفة مكان وجود الخلية في الأنسجة أمر بالغ الأهمية لفهم دور الخلية في تحفيز الالتهاب. على سبيل المثال ، يمكن للخلايا المناعية مثل الخلايا البائية التركيز في الجهاز العصبي المركزي أثناء الالتهاب العصبي. ومع ذلك ، نادرا ما تدخل حمة الجهاز العصبي المركزي وبدلا من ذلك تركز في حواجز الجهاز العصبيالمركزي 7. نظرا لتوطينها ، فمن غير المحتمل أن تساهم هذه الخلايا في التهاب الجهاز العصبي المركزي من خلال التفاعل جسديا مع الخلايا الدبقية في حمة الجهاز العصبي المركزي ، مما يشير إلى أن أي تفاعلات قد تكون مع الخلايا الدبقية ستحدث من خلال عوامل مفرزة. بالإضافة إلى ذلك ، غالبا ما يكون لعلم الأمراض الذي يحدث في اضطرابات الجهاز العصبي المركزي بنية 8,9 بحيث يمكن لتوطين الخلية في الأنسجة أن يحدد بشكل حاسم ما إذا كانت تساهم بنشاط في الاضطراب أو أنها متفرج. وبالتالي ، فإن استخدام التوجه المكاني لتقييم دور الخلية في علم الأمراض أمر ضروري.
عادة ما يتم إنجاز دراسة الخلايا في الأنسجة باستخدام الكيمياء الهيستولوجية المناعية أو الفحص المجهري المناعي. تكمن مشكلة هذه التقنيات في أنها عادة ما يمكنها فقط تصوير ما يصل إلى أربعة معلمات في وقت واحد. وهذا قيد رئيسي على هذه التقنيات، كما نعلم من تحليلات قياس التدفق الخلوي وتسلسل الحمض النووي الريبي أحادي الخلية أن العديد من مجموعات الخلايا تتطلب معلمتين أو أكثر لتحديدها؛ أيضا ، يزداد عدد المعلمات المطلوبة عادة عند البحث عن مجموعات فرعية محددة من نوع الخلية10. وبالتالي ، من غير العملي استخدام تقنيات التصوير القياسية لدراسة كيفية تفاعل مجموعات فرعية من الخلايا داخل الأنسجة.
تم التغلب على هذه المشكلة جزئيا من خلال طرق أحدث عالية المعلمات يمكنها الاحتفاظ بالمعلومات المكانية ، مثل تسلسل الحمض النووي الريبي المكاني11 وتصوير قياس الكتلةالخلوية 12. في حين أن هذه التقنيات ذات قيمة ، إلا أنها تواجه العديد من المشكلات ، مثل عدم توفرها على نطاق واسع ، وتقليل البيانات ثلاثية الأبعاد إلى بعدين ، وتتطلب خبرة كبيرة للتنفيذ. تقنية أخرى تعرف باسم التلوين المتسلسل ، حيث يتم تلطيخ الأنسجة بمجموعة واحدة من الأجسام المضادة متبوعة بتعطيل المجموعة السابقة من الأجسام المضادة قبل تلطيخها بمجموعة أخرى من الأجسام المضادة ، يمكن أن تحقق أنسجة عالية المعلمات دون الحاجة إلى معدات أو خبرة متخصصة13،14،15. ومع ذلك ، يمكن أن يكون التلوين المتسلسل كثيف العمالة ويتطلب قدرا كبيرا من وقت الفحص المجهري ، والذي قد يكون غير عملي للمختبرات التي لا تمتلك مجهرا شخصيا. وبالتالي ، هناك حاجة إلى تقنيات يمكنها زيادة عدد المعلمات الفلورية التي يمكن تصويرها في وقت واحد على المجاهر المتاحة على نطاق واسع وفي الوقت المناسب.
بمجرد الحصول على البيانات عالية المعلمات ، تنشأ مشكلة أخرى: من غير المرجح أن تقوم طرق تحليل الصور التقليدية بتحليل البيانات بنجاح. لا تكون تقنيات مثل العد اليدوي أو العتبة قابلة للتطبيق إلا إذا كان التحليل يتكون من معلمة واحدة أو إذا كانت العلامات المتعددة لها نفس التوطين حيث يتم حساب الإشارات المتداخلة فقط. هذا القيد يجعل التحليل التقليدي غير كاف للعمل مع مجموعات البيانات عالية المعلمات. تم تحقيق تحليل ناجح لمجموعات البيانات هذه من خلال تقسيم الخلايا المفردة من صور الأنسجة ثم إجراء استراتيجيات بوابات تشبه قياس التدفق الخلوي لتحديد أنواع الخلايا16,17. ومع ذلك ، هناك مشكلة أخرى تؤثر على هذه التحليلات وهي أنها تعمل فقط مع مجموعات البيانات حيث يتم فصل جميع الخلايا ذات الأهمية ماديا عن بعضها البعض ، لأن هذه التقنيات لا تستخدم طرقا يمكنها فصل الخلايا التي هي على اتصال جسدي بدقة. وبالتالي ، هناك حاجة إلى طريقة أحدث يمكنها إجراء تحليلات خلية واحدة على أقسام الأنسجة حتى لو كانت الخلايا على اتصال جسدي.
في هذه المقالة ، تم وصف بروتوكول بسيط يسمى قياس التدفق الخلوي النسيجي الذي تم تقديمه سابقا18 والذي يوسع عدد المعلمات الفلورية التي يمكن تصويرها في وقت واحد باستخدام المجاهر المتاحة على نطاق واسع. يعمل هذا البروتوكول عن طريق تلطيخ الأنسجة بأصباغ متداخلة طيفيا ثم استخدام التعويض الطيفي لإزالة النزيف من القنوات المتداخلة للحصول على بقع مفردة واضحة. لتسهيل تحليل صور الأنسجة عالية المعلمات ، يتم وصف خط أنابيب تحليل مفصل يستخرج الخلايا المفردة من أقسام الأنسجة لغرض فرز الخلايا إلى مجموعات متميزة باستخدام استراتيجيات بوابات تشبه قياس التدفق الخلوي. يعمل هذا البروتوكول في الأنسجة التي توجد فيها الخلايا بشكل منتشر وفي الأنسجة التي تضغط فيها الخلايا معا بشكل وثيق، مما يجعل هذه التقنية متعددة الاستخدامات لدراسة الأنسجة مثل الجهاز العصبي المركزي في كل من الاتزان الداخلي والالتهاب العصبي. وبالتالي ، فإن قياس التدفق الخلوي النسيجي هو تقنية مفيدة لدراسة التفاعلات بين أنواع الخلايا المعقدة التي تتطلب علامات خلايا متعددة لتحديد الخلايا مع الحفاظ على المعلومات المكانية.
Protocol
لا يغطي هذا البروتوكول تقسيم الأنسجة للأنسجة. يرجى الاطلاع على Jain et al.18 أو19 للحصول على أوصاف لكيفية تقسيم الأنسجة للأنسجة. يمكن استخدام هذا البروتوكول مع أي مناديل مقسمة على شرائح زجاجية. تستخدم هذه المقالة الغدد الليمفاوية الأربية المعزولة من محصن كما هو موضح سابقا18. يتم تلخيص الإجراء والجدول الزمني لهذا البروتوكول في الشكل 1. تفاصيل الكواشف والمعدات المستخدمة في هذه الدراسة مدرجة في جدول المواد.
1. تلطيخ أقسام الأنسجة
ملاحظة: بالإضافة إلى تلطيخ الأنسجة بجميع ملصقات الأجسام المضادة التي يهتم بها الباحث ، يجب على الباحث أيضا إعداد عناصر تحكم أحادية اللون (واحدة لكل معلمة فلورية مخصصة للاستخدام) على شرائح الأنسجة المجاورة أو المتطابقة حيث يتم تلطيخ كل عنصر تحكم في اللون بالكواشف المطلوبة لإنتاج إشارة معلمة فلورية واحدة.
- تحضير بيرم ومنع العازلة وبيرم وتلطيخ العازلة (انظر الجدول 1 للتكوين).
- قم بإذابة أقسام الأنسجة على الشرائح الزجاجية في درجة حرارة الغرفة وامسح التكثيف باستخدام مسح. استخدم علامة كارهة للماء لتحديد حدود الأنسجة (الأنسجة) التي يجب تلطيخها وترك العلامات تجف.
- إعادة ترطيب الأنسجة عن طريق نقع أقسام الأنسجة في محلول ملحي مخزن بالفوسفات (PBS) لمدة 1 دقيقة. تخلص من برنامج تلفزيوني واستخدم منديلا لإزالة PBS الزائد دون لمس الأنسجة مباشرة.
- إصلاح الأنسجة عن طريق إضافة ما يكفي من 4 ٪ بارافورمالدهيد (PFA) لتغطية الأنسجة تماما. احتضان في درجة حرارة الغرفة لمدة 20 دقيقة.
تنبيه: يمكن أن يكون PFA ساما ويجب التعامل معه داخل غطاء الدخان.
ملاحظة: هذه الخطوة مطلوبة فقط للأنسجة الطازجة المجمدة. إذا تم إصلاح الأنسجة بالفعل ، فتجاهل هذه الخطوة. يتم وصف تثبيت PFA هنا ، ولكن يمكن إصلاح الأنسجة باستخدام طرق أخرى حسب احتياجات التجربة. - اغسل المناديل في حمام PBS لمدة 1 دقيقة مع هزاز لطيف ، ثم قم بإزالة الشريحة من الحمام. استبدل برنامج تلفزيوني وكرر الغسيل 3 مرات أكثر.
- تخلص من PBS الزائد واستخدم منديلا لإزالة PBS الزائد دون لمس الأنسجة مباشرة.
- حدد الأجسام المضادة الثانوية التي يجب استخدامها في الخطوة 1.12 من البروتوكول وأنواع التي تشتق منها الأجسام المضادة الثانوية. قم بتقوية بيرم ومنع العازلة عن طريق إضافة مصل من هذه إلى بيرم ومنع المخزن المؤقت إلى تركيز نهائي بنسبة 5٪ لكل نوع.
ملاحظة: يساعد استخدام الأجسام المضادة الثانوية على تضخيم شدة الإشارة. وبالتالي ، يوصى بإقران الأجسام المضادة الأولية التي تنتج إشارات ضعيفة بأجسام مضادة ثانوية لتضخيم إشاراتها. إذا كانت جميع أهداف الأجسام المضادة التي يتم تلطيخها ساطعة بما يكفي بحيث لا تتطلب أجساما مضادة ثانوية ، فيمكن تخطي الخطوات 1.7-1.12 والخطوة 1.15. - منع الارتباط غير المحدد للأجسام المضادة عن طريق إضافة ما يكفي من بيرم المحصنة ومنع العازلة لتغطية كل نسيج. احتضان الشرائح لمدة 1-8 ساعات في درجة حرارة الغرفة في غرفة رطبة محمية من الضوء.
- أضف أجساما مضادة أولية غير مقترنة إلى بيرم وعازل تلطيخ عند التخفيفات المحددة تجريبيا (يجب أن تكون الأنواع أو الأنماط المتماثلة للغلوبولين المناعي للأجسام المضادة الأولية مختلفة ويمكن تمييزها بواسطة الأجسام المضادة الثانوية التي ينوي الباحث استخدامها في الخطوة 1.12).
- قم بتفريغ Perm وحظر المخزن المؤقت واستخدم منديلا لإزالة المخزن المؤقت الزائد دون لمس الأنسجة مباشرة. أضف ما يكفي من بيرم وعازل تلطيخ مع الأجسام المضادة الأولية إلى الأنسجة لتغطيتها واحتضان الشريحة عند 4 درجات مئوية لمدة 12-48 ساعة.
ملاحظة: سيحتاج الباحث إلى تحديد الوقت الأمثل لحضانة الأجسام المضادة. - إعداد العازلة حجب وتلطيخ العازلة (الجدول 1).
- أضف أجساما مضادة ثانوية إلى المخزن المؤقت للتلطيخ المعد في الخطوة 1.11 عند التخفيفات المحددة تجريبيا. يجب أن تستهدف الأجسام المضادة الثانوية المستخدمة نفس الأنواع أو الأنماط المتماثلة للأجسام المضادة التي تم استخدامها في الخطوة 1.9.
- تحضير المخزن المؤقت للتبريد (كما في الجدول 1).
ملاحظة: تساعد هذه الخطوة في تقليل التألق الذاتي من الأنسجة. إذا لم تكن هناك حاجة إلى ذلك ، فيمكن تخطي الخطوتين 1.13 و 1.14. - اغسل المناديل كما هو موضح في الخطوات 1.5-1.6. ثم أضف ما يكفي من مخزن التبريد لتغطية الأنسجة على الشرائح واحتضان الشرائح في درجة حرارة الغرفة لمدة 1-2 دقيقة.
- اغسل المنديل كما هو موضح في الخطوات 1.5-1.6. بعد ذلك ، أضف الأجسام المضادة الثانوية المخففة في المخزن المؤقت للتلطيخ المحضر في الخطوة 1.12 إلى الأنسجة بحيث يتم تغطية الأنسجة بالكامل في المخزن المؤقت للتلطيخ. احتضان الشرائح عند 4 درجات مئوية لمدة 2-8 ساعات.
- قم بإعداد محلول الحجب عن طريق إضافة 50 ميكرولتر من المصل العادي من أي نوع من الأجسام المضادة التي سيتم استخدامها في الخطوة 1.18 (5٪ من كل نوع) إلى المخزن المؤقت للحجب المعد في الخطوة 1.11.
- اغسل المناديل كما هو موضح في الخطوات 1.5-1.6. ثم ، أضف محلول الحجب مع المصل العادي المحضر في الخطوة 1.16 إلى الأنسجة بحيث يتم تغطيتها بالكامل في محلول تلطيخ. احتضان الشرائح في درجة حرارة الغرفة لمدة 1-8 ساعات.
- قم بإعداد محلول التلوين عن طريق إضافة أجسام مضادة أولية مترافقة بالفلوروفور عند التخفيفات المحددة تجريبيا إلى المخزن المؤقت للتلطيخ المعد في الخطوة 1.11. أضف أيضا بقعة نووية ، مثل الأصفر النووي ، إلى محلول التلوين عند تخفيف محدد تجريبيا.
ملاحظة: الخطوات 1.18-1.19 مطلوبة فقط إذا احتاج الباحث إلى استخدام ملصقات إضافية للأجسام المضادة لن تتطلب أجساما مضادة ثانوية لتضخيم الإشارة. في حالة عدم استخدام الأجسام المضادة المقترنة الأولية ، يمكن إضافة بقع النوى في الخطوة 1.12. - تخلص من المخزن المؤقت المانع واستخدم منديلا لإزالة المخزن المؤقت الزائد دون لمس الأنسجة مباشرة. أضف ما يكفي من محلول التلوين المحضر في الخطوة 1.18 إلى الأنسجة لتغطيته واحتضانه عند 4 درجات مئوية لمدة 12-48 ساعة.
- اغسل المناديل كما هو موضح في الخطوات 1.5-1.6. أضف 5 قطرات من الحامل إلى كل شريحة دون إدخال فقاعات ، وقم بتوجيه غطاء زجاجي برفق وببطء فوق المنديل لتغطية الأنسجة تماما في الحامل واستبعاد جميع فقاعات الهواء. قم بتخزين الشرائح على حرارة 4 درجات مئوية في الظلام حتى تصبح جاهزة للتصوير.
2. تصوير أقسام الأنسجة
ملاحظة: في هذه المرحلة ، يتم تلطيخ أقسام الأنسجة بجميع الأجسام المضادة ذات الأهمية لجميع الحالات التي يقصد تصويرها ، ويتم تلطيخ عناصر التحكم أحادية اللون بمعلمة فلورية واحدة في كل قسم. يجب أن يتم التصوير على مجهر يحتوي على كاشفات متعددة يمكن ضبطها للكشف عن نطاقات معينة من الضوء ، ومن الناحية المثالية ، يجب أن يكون لديه إمكانية الوصول إلى أكبر عدد ممكن من خطوط الليزر للإثارة.
- أدخل إحدى الشرائح الملطخة بالكامل في المجهر وقم بإعداد المجهر لتصوير القسم باستخدام الهدف المراد استخدامه لجمع البيانات.
- بناء على الفلوروفورات التي تلطخ بها الأنسجة ، حدد خطوط ليزر الإثارة اللازمة لتحفيز تلك الفلوروفورات وحدد نطاقات الانبعاث لكل فلوروفور تحتوي على أكبر قدر من الإشارة.
ملاحظة: يمكن أن تكون أدوات عرض أطياف التألق عبر الإنترنت (على سبيل المثال ، انظر جدول المواد) مفيدة لتحديد الأطوال الموجية المثلى للإثارة للفلوروفورات وتحديد النطاقات المثلى للكشف عن الفلوروفور. يجب اختيار الفلوروفورات المستخدمة في قياس التدفق الخلوي النسيجي بحيث يختلف كل فلوروفور في خط الليزر الأمثل الذي يتم تحفيز الفلوروفور به أو يختلف عن النطاق الأمثل لأطوال موجات الانبعاث. سيكون من الصعب فصل الفلوروفورات التي لها أطوال موجية أقرب للإثارة / الانبعاث. يتم تفصيل الفلوروفورات المستخدمة في هذه الدراسة في قسم النتائج. - افتح البرنامج 1 (انظر جدول المواد) في مجهر متحد البؤر وحدد علامة تبويب التكوين في الأعلى. ثم حدد زر الجهاز على الجانب الأيسر وقم بتغيير عمق البت إلى 16 بت.
ملاحظة: يوصى باستخدام أعماق بتات أعلى، مثل بيانات 16 بت، لجمع البيانات، لأن ذلك سيحسن دقة التعويض الطيفي. بالإضافة إلى ذلك ، ستعمل كثافات البكسل الأعلى ، والأرقام الأكبر لمتوسطات الخطوط ، والمزيد من الخطوات في z-stack على تحسين جودة الفصل الطيفي. - حدد علامة التبويب اكتساب في الجزء العلوي من الشاشة وأدخل إعداد التنسيق لتضمين كثافة البكسل للصورة (عادة ما تنتج الدقة الأعلى نتائج أفضل).
- إذا كان ذلك متاحا ، فحدد اكتساب X ثنائي الاتجاه لتسريع وقت التصوير ، بشرط أن تكون هذه الميزة قد تمت معايرتها بشكل مناسب.
- اختر قيمة الثقب للمجهر متحد البؤر (عادة ما بين 400-600 نانومتر) حيث تنتج القيم الأصغر عادة نتائج قياس خلوي عالي الجودة للتدفق النسيجي ، على الرغم من أن قيم الثقب الأصغر ستجعل من الصعب الحصول على الصور وقد تتطلب وقت تصوير إضافي أو شدة إثارة أعلى لإنتاج الصور.
- حدد إظهار لوحة المسح المتسلسل في أعلى القائمة اليسرى لفتح خيار الفحص المتسلسل. استخدم علامة "+" لإعداد تسلسلات إضافية ، حيث سيحتوي كل تسلسل على طول موجي واحد للإثارة تم تحديده في الخطوة 2.2.
- قم بتشغيل جميع أجهزة الليزر المطلوبة من خلال النقر على أزرار التشغيل المرتبطة بالليزر ذي الأهمية في وسط الشاشة لتحويلها إلى حالة تشغيل. انتقل إلى كل تسلسل محدد وقم بزيادة طاقة الليزر في وسط الشاشة إلى أي قيمة مرغوبة ، حيث سيكون لكل تسلسل طول موجي واحد للإثارة يتم استخدامه.
- قم بتشغيل العديد من أجهزة الكشف حسب الحاجة في الجزء السفلي من الشاشة في كل تسلسل لتصوير جميع الفلوروفورات التي سيتم تصويرها على خط الليزر هذا. اضبط نطاقات أجهزة الكشف عن طريق النقر المزدوج على نطاقاتها وإدخال النطاقات العلوية والسفلية للأطوال الموجية التي يجب جمعها.
ملاحظة: يوصى باستخدام أجهزة الكشف عالية الحساسية حيثما أمكن ، ومن الناحية المثالية ، يجب جمع جميع القنوات على نفس أنواع أجهزة الكشف ، على الرغم من أن هذا غير مطلوب. - اضغط على الزر المباشر ، ثم ركز على العينة. استخدم زر Auto Scale الموجود على يسار الصورة في البرنامج لتقييم ما إذا كانت القنوات مشبعة بشكل كاف بالإشارة. يجب على المستخدم ضبط قيم الكسب وشدة الليزر في كل تسلسل حتى لا يصل القياس التلقائي إلى القيمة القصوى (65535 لبيانات 16 بت) ولكنه قريب جدا من التشبع لجميع القنوات.
ملاحظة: نظرا لأن الفلوروفورات المختلفة يمكن أن تختلف في سطوعها ويمكن أن تختلف أهداف الأجسام المضادة المختلفة في وفرتها ، فستحتاج حساسية الكاشفات إلى التعديل لتكون ذات حساسية منخفضة للإشارات الساطعة وحساسية عالية للإشارات الخافتة إذا تم تحفيز كلا الفلوروفورين بواسطة نفس الليزر (لا يمكن أن تتغير شدة الليزر في هذا السيناريو حيث سيتأثر كلا الفلوروفور). للحصول على أفضل النتائج ، يجب موازنة إشارة التألق من كل قناة في التجربة بحيث تحتوي كل قناة على كميات مكافئة تقريبا من الإشارة ، مما يقلل من النزيف الطيفي إلى قنوات أخرى ويؤدي إلى إشارات أقوى. نظرا لأن العينات المختلفة قد تختلف في وفرة العلامات المثيرة للاهتمام الملطخة ، فمن المستحسن اختبار إعدادات المجهر في ظروف تجريبية متعددة للتأكد من أن التبييض لا يحدث في ظل أي من الظروف التجريبية. - بمجرد الانتهاء من إعدادات المجهر ، قم بتحميل المجهر بشرائح التحكم في اللون الفردي وابحث عن تلطيخ تمثيلي لكل فلوروفور. قم بإعداد مكدس Z من خلال التركيز أسفل العينة والضغط على زر البدء في قائمة مكدس z ، ثم التركيز فوق العينة والضغط على زر النهاية .
- اختر عدد الخطوات والمسافة بين الخطوات (واستخدم نفس عدد الخطوات والمعلمات مثل كثافة البكسل وعمق البت التي سيتم استخدامها لتصوير الشرائح الملطخة بالكامل) ، ثم اضغط على زر البدء.
ملاحظة: من الناحية المثالية ، يجب أن تتضمن هذه الصور ألمع الإشارات من تلطيخ حقيقي يمكن العثور عليه على الشريحة. - بعد تصوير عناصر التحكم أحادية اللون ، تابع استخدام نفس إعدادات المجهر لتصوير جميع العينات الملطخة بالكامل.
3. تعويض مضان
- افتح ملفات .lif في البرنامج 1 وانقر فوق علامة تبويب العملية.
- اختر وحدة فصل الصبغة ثم اختر خيار فصل الصبغة التلقائي . حدد الطريقة اليدوية لفصل الصبغة واختر عدم إعادة القياس.
- افتح إحدى صور التحكم أحادية اللون. افحص يدويا عناصر التحكم أحادية اللون بحثا عن التسييل الطيفي من قناة إلى أخرى من خلال مراقبة النوافذ ذات التدرج الرمادي لكل قناة بحثا عن دليل على وجود إشارة في قنوات غير مناسبة. استخدم زر القياس الآلي إذا لزم الأمر لرؤية التسييل الضعيف.
- ابدأ في إزالة التسييل عن طريق إدخال الأرقام يدويا (عادة 0-1 ، حيث يمثل 0 عدم حدوث تسييل و 1 يمثل تسييل 100٪) في المصفوفة على الشاشة ، ثم الضغط على تطبيق لاختبار ما إذا كان قد تمت إزالة التسييل بشكل كاف. يجب تكرار هذا حتى يتم تخفيض الإشارات في جميع القنوات الأخرى باستثناء قناة الاهتمام إلى مستويات الخلفية.
ملاحظة: سيؤدي التعويض الزائد إلى نتائج غير دقيقة ، لذلك يجب تقليل التألق إلى مستويات الخلفية ولكن ليس أقل منها. ستظهر البيانات ذات التعويض الزائد كقناة واحدة بها إشارة وقنوات أخرى يبدو أنها بها ثقوب سوداء حيث تكون إشارة الاهتمام (الشكل 2). - بمجرد اكتمال أحد عناصر التحكم أحادية اللون، قم بتسجيل القيم في المصفوفة ثم أعد تعيين المصفوفة، وانتقل إلى عنصر التحكم أحادي اللون التالي. كرر هذه العملية لكل عنصر تحكم بالألوان.
ملاحظة: ستكون الصبغة الأولى في صف الصبغة 1 ، وستكون كمية التألق التي يجب إزالتها من الصبغة 1 في الأعمدة المجاورة. - قم بتجميع جميع القيم المكتسبة من كل عنصر تحكم في مصفوفة واحدة وقم بتطبيق هذه المصفوفة على عينة ملطخة بالكامل. إذا كان الفصل يبدو دقيقا، قم بتطبيق الإعدادات على كل الصور في التجربة، وإلا، أعد تقييم عناصر التحكم أحادية اللون.
ملاحظة: يجب أن يزيل التعويض الفعال معظم النزيف عبر القنوات ، كما هو ملخص وموضح في الشكل 3. - احفظ جميع الملفات التي تم تعويضها في ملف .lif منفصل.
4. تحديد النوى والخلايا باستخدام الإيلاستيك
- افتح ملفات .lif المعوضة في FIJI20. قم بتمكين عرض الحزم المكدسة في مكدس فائق وحدد صورة.
- (اختياري) استخدم أداة تحديد مثل أداة المستطيل لتضمين أجزاء الصورة التي سيتم تحليلها واستبعاد المساحات التي لن يتم تحليلها. اقتصاص الصورة باستخدام الصورة > وظيفة الاقتصاص.
ملاحظة: يعد تقليل حجم الصورة مفيدا في الخطوات اللاحقة لتقليل استهلاك ذاكرة الوصول العشوائي (RAM) وزيادة سرعة المعالجة. - احفظ الصورة كملف .tif. أيضا ، احفظ الصورة كملف hdf5 باستخدام المكون الإضافي ilastik ل FIJI بالنقر فوق المكونات الإضافية > ilastik > Export hdf5. احفظ الملف ك "اسم الاختيار" .h5 مع ضغط 0.
- افتح Ilastik21 وقم بعمل مشروع تصنيف بكسل. استخدم وظيفة إضافة > جديد إضافة صور منفصلة لإضافة 3 صور تمثيلية على الأقل بالانتقال إلى إضافة بيانات إدخال جديدة.
- انقر بزر الماوس الأيمن فوق إحدى الصور ، وحدد تحرير الخصائص ، وقم بتغيير وضع العرض إلى تدرج الرمادي.
- على يمين الشاشة، حدد علامة التبويب تحديد الميزة . انقر فوق تحديد الميزات وحدد جميع الميزات حتى أعلى قيمة سيجما التي سيسمح لك البرنامج باستخدامها ، ثم اضغط على موافق.
- الآن حدد علامة التبويب التدريب. استخدم أداة فرشاة الرسم (أثناء تحديد التسمية الأولى ) لتمييز الخلايا الفردية (الشكل 4) ذات الأهمية بحيث يتم تضمين الجزء الداخلي (السيتوبلازم والنواة) وغشاء الخلية في التمييز.
- كرر هذا عبر جميع عينات التدريب حيث يتم اختيار جميع أنواع الخلايا ذات الأهمية ، ويجب اختيار خلايا متعددة متفاوتة الشدة والتشكل والتوطينات في الأنسجة من كل عينة.
ملاحظة: اعتمادا على العلامات المختارة لمجموعة فردية من البقع ، قد تكون هناك أو لا توجد بقع داخل الخلايا أو بقع غشاء الخلية للخلايا ذات الأهمية. مهما كانت العلامات المستخدمة ، تأكد من أن التمييز يشمل البقعة الخارجية على الخلايا ذات الاهتمام ويتضمن داخل الخلية بغض النظر عما إذا كان يتم استخدام بقعة داخل الخلايا. - بعد ذلك ، استخدم التسمية الثانية لتمييز كل شيء ليس خلايا الاهتمام (الشكل 4). يمكن أن يشمل ذلك مساحة فارغة أو نوى غير مسماة أو أنسجة لا تحتوي على علامات اهتمام. تأكد من تضمين المساحة المجاورة مباشرة للخلايا ذات الأهمية لتعزيز تعلم حدود الخلايا الدقيقة.
- استخدم وظيفة Live Update لتقييم جودة التدريب على الصور ذات الأهمية. إذا كان التدريب غير كاف ، فتابع التدريب في الخطوات من 4.7 إلى 4.9. خلاف ذلك ، إذا كان مرضيا ، انقر فوق علامة التبويب تصدير التنبؤ .
- قم بتعيين المصدر ليكون احتمالات. انقر فوق الزر اختيار تصدير إعدادات الصورة ، ثم قم بتعيين المعلمة c للتصدير 0-1 (بافتراض أن التسمية 1 تسلط الضوء على المعلمات ذات الأهمية).
- قم بتحويل نوع البيانات إلى عدد صحيح 16 بت ، وقم بتصديره كملف hdf5 إلى دليل الاهتمام باسم الاهتمام (نوصي باستخدام {nickname} mask.h5) ، ثم اضغط على موافق.
- انقر فوق علامة التبويب معالجة الدفعات . اضغط على الزر تحديد ملفات البيانات الأولية ، وحدد كافة ملفات .h5 ذات الأهمية التي تتطلب معالجة، ثم اضغط على فتح. انقر فوق معالجة جميع الملفات.
- كرر الخطوات من 4.4 إلى 4.13 ، باستثناء بدلا من تحديد الخلايا ، حدد الآن النوى واستبعد كل شيء آخر (الشكل 4). تصدير هذا كتنبؤ منفصل لتحديد نوى الخلية.
- افتح Ilastik وأنشئ مشروع تحويل بيانات جديد. أضف ملف hdf5 تمثيليا إلى علامة التبويب بيانات الإدخال باستخدام وظيفة إضافة > جديد إضافة صورة منفصلة .
- انقر فوق علامة التبويب تصدير البيانات واختر زر اختيار إعدادات تصدير الصورة مع تعيين المصدر على الإدخال. قم بتغيير التنسيق إلى tiff متعدد الصفحات ، واختر موقع حفظ باستخدام وظيفة التحديد ، وقم بتعيين اسم الملف إلى {nickname}. اضغط على موافق.
- انقر فوق علامة التبويب معالجة الدفعات وحدد جميع ملفات hdf5 الخاصة بك باستخدام ملف حدد ملفات بيانات الإدخال زر واضغط على معالجة جميع الملفات زر.
- افتح البرنامج 2 (انظر جدول المواد) وأضف جميع ملفات tif/tiff التي تم إنشاؤها في الخطوتين 4.3 و4.17 باستخدام الزر إضافة ملفات . اختر موقعا لمخرجات الملفات واضغط على الزر Start All لتحويل كافة الملفات إلى ملفات .ims. الملفات جاهزة الآن للتحليل في البرنامج 3 (انظر جدول المواد).
5. التحليل في البرنامج 3
- افتح إحدى الصور التي تحتوي على جميع بقع الفلورسنت. حدد تحرير-> إضافة قنوات وأضف ملفات .ims المقابلة للأقنعة المصنوعة على أجسام النوى والخلايا. كرر هذا لجميع العينات.
- حدد الزر إضافة خلايا جديدة . حدد خيار الكشف عن النواة والخلية والمضي قدما.
- حدد القناع فوق النوى ليكون قناة المصدر للكشف عن النوى وحدد الخيار المتقدم لتقسيم النوى بنقاط البذور. اختر قيمة لقطر النواة) والمضي قدما.
ملاحظة: يجب اختبار القيمة المثلى لقطر النواة عن طريق تشغيل المحاكاة والتقييم اليدوي لما إذا كان البرنامج لا يقسم نوى كافية (يتم تجميع نوى متعددة معا) أو ما إذا كان هناك انقسام زائد (يتم تقسيم نواة واحدة إلى أكثر من واحدة). - اختر قيمة لتقييم الجودة، ثم تابع. عادة ما يكون من الأفضل تضمين جميع النوى ، لأن إغفال النوى يمكن أن يؤدي إلى اندماج خلايا متعددة في خلية واحدة.
- اختر قيمة عتبة لإنشاء أسطح نووية ثم تابع. تأكد من أن جميع النوى ، حتى تلك التي تكون باهتة ، لا تزال مدرجة في العتبة. لا تجعل العتبة منخفضة للغاية ، لأن هذا قد يبالغ في تقدير حجم النوى أو يحتمل أن يشمل الحطام على أنه تلطيخ نووي.
- حدد قيمة لعدد الحد الأدنى لعدد Voxels لكل نواة وتابع. عادة ، من الأفضل الاحتفاظ بكل شيء ما لم يكن من الممكن استبعاد الحطام أو غير النوى باستخدام هذه المعلمة.
- اختر خيار حدود الخلية والسيتوبلازم ، وحدد القناع فوق الخلايا ليكون القناة المصدر لاكتشاف أجسام الخلايا ، ثم تابع.
- اختر عتبة خلية تلتقط بدقة جميع الخلايا ذات الأهمية ولكنها ليست منخفضة جدا بحيث تتضمن الخلفية أو أي اكتشاف غير محدد للخلايا. حدد خيار تقسيم الخلايا الملامسة بحيث توجد نواة واحدة لكل خلية ، ويتم فصلها بالمسافة من النوى. اختر أيضا توسيع الخلايا على النوى ثم تابع.
- حدد قيمة للحد الأدنى لعدد voxels لكل خلية وتابع. عادة ، من الأفضل الاحتفاظ بكل شيء ما لم يكن من الممكن استبعاد الحطام أو غير الخلايا باستخدام هذه المعلمة.
- قم بتقييم ما إذا كانت الأسطح التي تم إنشاؤها فوق الخلايا دقيقة من خلال النظر فيما إذا كانت خلايا متعددة قد تم دمجها معا أو إذا كانت الخلايا مفقودة. إذا كانت الأسطح غير كافية ، فارجع إلى الخطوة 5.2 وجرب معلمات جديدة حتى يتم تحديد الإعدادات المثلى.
- بمجرد إنشاء أفضل الإعدادات ، انقر فوق كائن الخلايا التي تم إنشاؤها في القائمة اليسرى ، وانقر فوق علامة التبويب إنشاء ، وانقر فوق زر تخزين المعلمات للدفعة لحفظ الإعدادات المستخدمة.
- افتح نوافذ متعددة من البرنامج 3 وافتح صورا منفصلة في كل نافذة. اضغط على زر الخلايا الجديد في كل نافذة ، واختر الإعدادات المحفوظة في قائمة معلمات الإنشاء المفضلة ، واطلب من كل نافذة أن تعمل حتى الانتهاء. كرر هذا عدة مرات حسب الحاجة حتى يتم تحليل جميع العينات.
ملاحظة: سيؤدي تشغيل مثيلات متعددة من البرنامج 3 إلى متابعة هذا التحليل بشكل أسرع، ولكن سيعتمد عدد الإطارات التي يمكن تشغيلها بالتوازي على سرعة معالجة وحدة المعالجة المركزية (CPU) ومقدار ذاكرة الوصول العشوائي التي يتم تشغيل الكمبيوتر الذي يتم تشغيل التحليل عليه. - بمجرد اكتمال جميع العينات ، انقر فوق كائن الخلايا التي تم إنشاؤها في القائمة اليسرى في كل صورة وانقر فوق علامة التبويب إحصائيات. انقر فوق الزر تكوين قائمة قيم الإحصائيات المرئية ، وقم بإلغاء تحديد كافة الإحصائيات باستثناء قيمة "متوسط كثافة الخلية"، ثم اضغط على موافق.
- لكل صورة تحتاج إلى تحليل ، انقر فوق تصدير جميع الإحصائيات إلى ملف زر في علامة التبويب إحصائيات واحفظ الملفات في موقع.
6. تحليل قياس التدفق الخلوي النسيجي ورسم خرائط السكان على أقسام الأنسجة
- افتح أناكوندا وجوبيتر لاب داخل أناكوندا. بعد ذلك ، افتح البرنامج النصي المقدم في ملف الترميز التكميلي 1. قم بتشغيل التعليمة البرمجية باستخدام قائمة التشغيل وحدد تشغيل كافة الخلايا ، أو قم بتشغيل الخلايا بشكل فردي بالترتيب عن طريق تحديد تشغيل الخلايا المحددة.
- عند مطالبتك بإدخال الملف المصدر، أدخل دليل الملف لقيم الفلورسنت المصدرة التي تم إنشاؤها في الخطوة 5.14. يمكن الحصول على دليل الملف من خلال النظر في خصائص أحد ملفات .csv المصدرة ونسخ مسار الملف إلى المطالبة.
- بعد ذلك ، سيطلب البرنامج موقع إخراج للملفات المعالجة. أدخل دليل الملف لأي موقع مناسب لحفظ الملفات التي تمت معالجتها. سيكتشف البرنامج تلقائيا عدد قنوات الفلورسنت الموجودة في المجلد.
- قم بتشغيل القسم التالي من التعليمات البرمجية لإضافة تعليقات توضيحية إلى البيانات بأسماء كل قناة. يمكن تعديل هذا الرمز حسب الحاجة لتغيير المعلمة "numberOfChannels" لمطابقة عدد القنوات المكتشفة في الخطوة 6.3. بالإضافة إلى ذلك ، يمكن تعديل أسماء القنوات لأي عدد ممكن من القنوات حسب الحاجة عن طريق تكرار سطر "ChX": "Marker" عدة مرات حسب الحاجة وتعديل أسماء العلامات.
- سيؤدي تشغيل السطر التالي من التعليمات البرمجية إلى إنشاء ملف .csv في الموقع المحدد الذي يتم التعليق عليه ويحتوي على جميع معلمات الفلورسنت في مجموعة البيانات. كرر الخطوات من 6.1 إلى 6.5 لكل مجموعة من عمليات تصدير البرامج 3 لإنشاء ملفات .csv لكافة البيانات.
- قم بتحويل ملفات .csv إلى ملفات .fcs عن طريق فتح البرنامج 4 (انظر جدول المواد) وسحب ملفات .csv إلى نافذة البرنامج. سيبدأ البرنامج 4 تلقائيا في تحويل الملفات إلى ملفات .fcs.
- انقر نقرا مزدوجا فوق إحدى العينات الموجودة في البرنامج 4 لفتح نافذة على تلك العينة. استخدم أدوات البوابات في البرنامج 4 لإنشاء استراتيجية بوابة. يوصى بالبدء ببوابة على علامة تعريف النسب تحتوي على جميع الخلايا ذات الأهمية ، ثم الانتقال إلى البوابات التي تزيل المجموعات الفرعية النادرة أولا ، ثم الانتهاء من أنواع الخلايا الأكثر وفرة.
- بمجرد إنشاء استراتيجية البوابات ، اختبر دقة البوابة بالنقر بزر الماوس الأيمن على تلك المجموعة في القائمة واختيار تصدير. تصدير جميع المعلمات كملف .csv مع تضمين الرؤوس.
- افتح JupyterLab في أناكوندا. بعد ذلك ، افتح البرنامج النصي المقدم في ملف الترميز التكميلي 2 ، وقم بتشغيل الكود. عند مطالبتك بإدخال موقع ملف Software 4 ، أدخل دليل الملف لملف .csv الذي تم تصديره والذي تم إنشاؤه في الخطوة السابقة. يمكن الحصول على دليل الملف من خلال النظر في خصائص ملف .csv المصدر ونسخ مسار الملف إلى المطالبة.
- عند مطالبتك بإضافة موقع الإخراج ، اختر أي دليل ملف من اختيارك ، وتأكد من أن دليل الملف يتضمن اسم الملف في النهاية ('دليل الملف' \ filename.txt). قم بتشغيل بقية التعليمات البرمجية.
- انسخ النص في ملف .txt وافتح ملف .ims المقابل في البرنامج 3 لنفس العينة التي تستند إليها البيانات المصدرة. انقر فوق كائن الخلية في الملف ، وقم بالتبديل إلى علامة التبويب الإحصائيات ، والصق النص في شريط البحث ، وابدأ البحث. سيتم تسليط الضوء على جميع الخلايا ذات الاهتمام.
- تحقق يدويا مما إذا كانت جميع الخلايا ذات الاهتمام قد تم التقاطها باستخدام استراتيجية البوابة. إذا تم التقليل من عدد الخلايا أو المبالغة في تقديرها (الأمثلة الموضحة في الشكل 5) ، فارجع إلى الخطوة 6.7 ، واضبط البوابة ، وأعد تقييم ما إذا كانت الخلايا قد تم التقاطها بدقة باستخدام البوابة الجديدة. كرر هذا مع جميع البوابات في استراتيجية البوابة وقم بتقييم البوابة على عينات متعددة.
- عندما يتم التحقق من صحة جميع البوابات ، قم بتصدير البيانات من البرنامج 4 باستخدام وظيفة محرر الجدول وتحليل البيانات باستخدام طريقة التحليل المفضلة.
ملاحظة: يمكن تعيين مجموعات الخلايا مرة أخرى على أقسام الأنسجة في البرنامج 3 عن طريق تحديد السكان المسورة كما في الخطوات 6.8-6.11 ثم إنشاء سطح بناء على الخلايا المحددة. يمكن القيام بذلك لكل مجموعة مسورة ، كما هو موضح في الشكل 6 والشكل 7.
النتائج
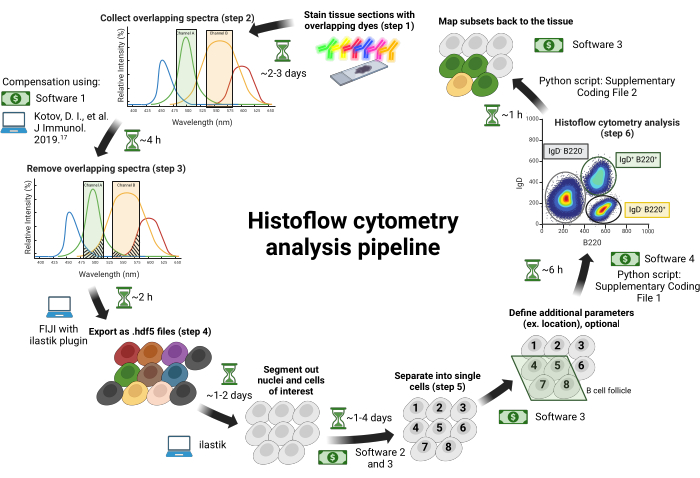
الشكل 1: سير عمل قياس التدفق الخلوي للنسيج. أقسام الأنسجة ملطخة بأصباغ متداخلة طيفيا (الخطوة 1). يتم جمع الصور عبر ليزر الإثارة الفردي المقترن بمرشحات تمرير النطاق القابلة للضبط لتقليل النزيف ال?...
Discussion
هنا ، يتم وصف استخدام قياس التدفق الخلوي النسيجي ، وهي تقنية تم التحقق من صحتها سابقا18. ثبت أنه عند تلطيخ أقسام الأنسجة بأصباغ متداخلة طيفيا ، يمكن إزالة هذا النزيف عبر القنوات باستخدام التعويض الطيفي ، مما يؤدي إلى حل عدد أكبر من معلمات الفلورسنت بوضوح أكثر مما هو ممكن عادة م?...
Disclosures
ليس لدى المؤلفين أي تضارب في المصالح المالية.
Acknowledgements
نشكر منصة الفحص المجهري المتقدمة لمعهد هوتشكيس للدماغ على البنية التحتية للتصوير والخبرة. تم دعم RWJ من خلال تمويل زمالة ما بعد الدكتوراه من برنامج جامعة كالجاري Eyes High ومن قبل جمعية التصلب المتعدد في كندا وزمالة Roche Canada التعليمية غير المقيدة. تلقت VWY دعم الراتب من برنامج كرسي الأبحاث الكندي من المستوى 1. تم دعم هذا العمل من خلال أموال التشغيل من المعاهد الكندية للبحوث الصحية Grant 1049959 ، ومنحة جمعية التصلب المتعدد الكندية 3236 ، ووزارة الدفاع الأمريكية لبرنامج أبحاث التصلب المتعدد الموجه من الكونغرس. يتم إنشاء الشكل 1 باستخدام BioRender.com. تم نشر الأرقام التي تم تكييفها في هذا المنشور في الأصل في مجلة علم المناعة. راجيف دبليو جاين وديفيد أ. إليوت و في وي يونغ. 2023. تحليل الخلية المفردة لصور الأنسجة عالية المعلمات باستخدام قياس التدفق الخلوي النسيجي. J. المناعي. 210: 2038-2049. حقوق النشر © [2023]. الرابطة الأمريكية لعلماء المناعة ، Inc.
Materials
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
References
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved