Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Создание и анализ гистологических изображений с высокими параметрами с помощью гистопрофлоуцитометрии
В этой статье
Резюме
Здесь описан метод, который может быть использован для визуализации пяти или более флуоресцентных параметров с помощью иммунофлуоресцентной микроскопии. Описан конвейер анализа для извлечения отдельных клеток из этих изображений и проведения анализа отдельных клеток с помощью стратегий проточной цитометрии, подобных гейтингу, которые могут идентифицировать подмножества клеток в срезах тканей.
Аннотация
Использование гистологии для исследования разнообразия иммунных клеток в срезах тканей, таких как срезы центральной нервной системы (ЦНС), критически ограничено количеством флуоресцентных параметров, которые могут быть визуализированы за один раз. Большинство подмножеств иммунных клеток были определены с помощью проточной цитометрии с использованием сложных комбинаций белковых маркеров, часто требующих четырех или более параметров для окончательной идентификации, что выходит за рамки возможностей большинства обычных микроскопов. Поскольку проточная цитометрия диссоциирует ткани и теряет пространственную информацию, существует потребность в методах, которые могут сохранять пространственную информацию при исследовании ролей сложных типов клеток. Эти проблемы решаются путем создания метода расширения числа флуоресцентных параметров, которые могут быть отображены путем сбора сигналов спектрально перекрывающихся флуорофоров и использования спектрального расмешивания для разделения сигналов каждого отдельного флуорофора. Затем эти изображения обрабатываются с помощью конвейера анализа для получения гистологических изображений с высокими параметрами и извлечения отдельных клеток из этих изображений, чтобы уникальные флуоресцентные свойства каждой клетки могли быть проанализированы на уровне одной клетки. Используя стратегии проточной цитометрии, клетки затем могут быть профилированы на подмножества и отображены обратно в гистологические разделы, чтобы не только количественно оценить их распространенность, но и установить, как они взаимодействуют с тканевой средой. В целом, продемонстрирована простота и потенциал использования гистопроточной цитометрии для изучения сложных иммунных популяций в гистологических срезах.
Введение
Воспаление, вызванное клетками иммунной системы и глиальными клетками, может способствовать хроническим заболеваниям ЦНС, где каждая популяция может способствовать активности другой 1,2,3. Понимание того, как иммунная система взаимодействует с этими элементами ЦНС, способствуя воспалению ЦНС, в настоящее время является основной темой интереса и в значительной степени облегчается методами с высокими параметрами, такими как секвенирование РНК одиночных клеток. С помощью секвенирования РНК одиночных клеток мы обнаружили, что существует обширная коммуникация между глиальными клетками и иммунной системой при нескольких заболеваниях ЦНС 4,5,6. Понимание того, как эти взаимодействия влияют на эти расстройства, будет иметь решающее значение для выяснения биологии этих заболеваний.
Одна из проблем с анализом секвенирования одиночных клеток заключается в том, что эти методы требуют разрушения ткани для получения отдельных клеток или ядер, что приводит к полной потере пространственной информации. Знание того, где в ткани находится клетка, имеет решающее значение для понимания роли клетки в развитии воспаления. Например, иммунные клетки, такие как В-клетки, могут концентрироваться в ЦНС во время нейровоспаления; однако они редко проникают в паренхиму ЦНС и вместо этого концентрируются в барьерах ЦНС7. Учитывая их локализацию, маловероятно, что эти клетки способствуют воспалению ЦНС, физически взаимодействуя с глиальными клетками в паренхиме ЦНС, предполагая, что любые взаимодействия, которые они могут иметь с глиальными клетками, будут происходить через секретируемые факторы. Кроме того, патология, возникающая при заболеваниях ЦНС, часто имеет структуру 8,9 таким образом, что локализация клетки в ткани может критически определить, активно ли она способствует заболеванию или является сторонним наблюдателем. Таким образом, использование пространственной ориентации для оценки роли клетки в патологии имеет важное значение.
Изучение клеток в тканях обычно осуществляется с помощью иммуногистохимии или иммунофлуоресцентной микроскопии. Проблема с этими методами заключается в том, что они обычно могут отображать только до четырех параметров одновременно. Это является основным ограничением этих методов, поскольку мы знаем из анализа проточной цитометрии и секвенирования РНК отдельных клеток, что многим популяциям клеток требуются два или более параметров для их идентификации; Кроме того, количество необходимых параметров обычно увеличивается при поиске конкретных подмножеств ячейки типа10. Таким образом, нецелесообразно использовать стандартные методы визуализации для изучения того, как подмножества клеток могут взаимодействовать в ткани.
Эта проблема была частично решена с помощью новых методов с высокими параметрами, которые могут сохранять пространственную информацию, таких как пространственное секвенирование РНК11 и визуализационная массовая цитометрия12. Несмотря на то, что эти методы ценны, у них есть несколько проблем, таких как недостаточная доступность, сведение трехмерных данных к двум измерениям и требование значительного опыта для реализации. Другой метод, известный как последовательное окрашивание, при котором ткани окрашиваются одним набором антител с последующей инактивацией предыдущего набора антител перед окрашиванием другим набором антител, позволяет достичь гистологии с высокими параметрами без необходимости использования специализированного оборудования или опыта 13,14,15. Тем не менее, последовательное окрашивание может быть чрезвычайно трудоемким и требует большого количества времени на микроскопию, что может быть непрактично для лабораторий, не владеющих персональным микроскопом. Таким образом, существует потребность в методах, которые могут расширить количество флуоресцентных параметров, которые могут быть визуализированы за один раз на микроскопах, которые широко доступны и своевременно.
После того, как данные с высокими параметрами были получены, возникает другая проблема: обычные методы анализа изображений вряд ли смогут успешно проанализировать данные. Такие методы, как ручной подсчет или пороговое значение, жизнеспособны только в том случае, если анализ состоит из одного параметра или если несколько маркеров имеют одинаковую локализацию, где подсчитываются только перекрывающиеся сигналы. Это ограничение делает традиционный анализ неадекватным для работы с наборами данных с высокими параметрами. Успешный анализ этих наборов данных был достигнут путем сегментации отдельных клеток по гистологическим изображениям и последующего проведения стратегий гейтирования, подобных проточной цитометрии, для идентификации типов клеток16,17. Тем не менее, еще одна проблема, влияющая на эти анализы, заключается в том, что они работают только для наборов данных, в которых все интересующие клетки физически отделены друг от друга, поскольку эти методы не используют методы, которые могут точно разделить клетки, находящиеся в физическом контакте. Таким образом, требуется более новый метод, который может проводить анализ отдельных клеток на срезах гистологии, даже если клетки находятся в физическом контакте.
В данной статье описывается простой протокол, называемый гистопотоковой цитометрией, которыйбыл введен ранее и расширяет число флуоресцентных параметров, которые могут быть одновременно визуализированы с помощью широко доступных микроскопов. Этот протокол работает путем окрашивания тканей спектрально перекрывающимися красителями, а затем с использованием спектральной компенсации для удаления просачивания из перекрывающихся каналов для получения прозрачных одиночных пятен. Для облегчения анализа гистологических изображений с высокими параметрами описывается подробный конвейер анализа, который извлекает отдельные клетки из срезов тканей с целью сортировки клеток в отдельные популяции с использованием стратегий гейтирования, подобных проточной цитометрии. Этот протокол работает в тканях, где клетки присутствуют диффузно, и в тканях, где клетки плотно сжаты друг с другом, что делает этот метод универсальным для изучения тканей, таких как ЦНС, как в гомеостазе, так и в нейровоспалении. Таким образом, гистопроточная цитометрия является полезным методом для изучения взаимодействий между сложными типами клеток, которым требуется несколько клеточных маркеров для определения клеток, сохраняя при этом пространственную информацию.
протокол
Этот протокол не распространяется на срезы тканей для гистологии; пожалуйста, обратитесь к Jain et al.18 или19 для описания того, как делать срезы тканей для гистологии. Этот протокол можно использовать с любыми разрезанными тканями на предметных стеклах. В данной статье используются паховые лимфатические узлы, выделенные от иммунизированного животного, как описано ранее18. Процедура и график работы с этим протоколом кратко изложены на рисунке 1. Подробная информация о реагентах и оборудовании, использованных в этом исследовании, приведена в Таблице материалов.
1. Окрашивание участков ткани
Примечание: В дополнение к окрашиванию тканей всеми метками антител, которые интересуют исследователя, исследователь также должен подготовить один цветовой контроль (по одному для каждого флуоресцентного параметра, который предполагается использовать) на смежных или иным образом идентичных тканевых предметных стеклах, при этом каждый отдельный цветовой контроль окрашивается реагентами, необходимыми для получения сигнала одного флуоресцентного параметра.
- Приготовьте Perm и блокирующий буфер и Perm и окрашивающий буфер (состав см. в Таблице 1 ).
- Разморозьте срезы салфеток на предметных стеклах при комнатной температуре и сотрите конденсат с помощью салфетки. С помощью гидрофобного маркера очертите границу тканей, которые необходимо окрасить, и дайте следам высохнуть.
- Регидратируйте ткани, замочив участки тканей в фосфатно-солевом буфере (PBS) на 1 минуту. Стряхните PBS и с помощью салфетки удалите излишки PBS, не касаясь непосредственно ткани.
- Зафиксируйте ткани, добавив достаточное количество 4% параформальдегида (PFA), чтобы полностью покрыть ткань. Выдерживать при комнатной температуре в течение 20 минут.
ВНИМАНИЕ: PFA может быть токсичным, и с ним следует обращаться в вытяжном шкафу.
ПРИМЕЧАНИЕ: Этот шаг требуется только для свежезамороженных тканей. Если ткань уже зафиксирована, то игнорируйте этот шаг. Фиксация PFA описана здесь, но ткань может быть зафиксирована другими методами в зависимости от потребностей эксперимента. - Постирайте салфетки в ванне PBS в течение 1 минуты с легким покачиванием, а затем снимите горку с ванны. Замените PBS и повторите стирку еще 3 раза.
- Стряхните излишки PBS и с помощью салфетки удалите излишки PBS, не касаясь непосредственно ткани.
- Определите, какие вторичные антитела необходимо использовать на шаге 1.12 протокола, а также виды животных, от которых получены вторичные антитела. Укрепите завивку и блокирующий буфер, добавив сыворотку от этих животных в завивку и блокирующий буфер до конечной концентрации 5% для каждого вида.
ПРИМЕЧАНИЕ: Использование вторичных антител помогает усилить интенсивность сигнала. Таким образом, рекомендуется, чтобы первичные антитела, продуцирующие слабые сигналы, сочетались со вторичными антителами для усиления их сигналов. Если все окрашиваемые мишени антител достаточно яркие, чтобы не требовать вторичных антител, шаги 1.7-1.12 и 1.15 можно пропустить. - Блокируйте неспецифическое связывание антител путем добавления достаточного количества обогащенной химической завивки и блокирующего буфера, чтобы покрыть каждую ткань. Выдержите предметные стекла в течение 1-8 часов при комнатной температуре во увлажненной камере, защищенной от света.
- Добавляйте неконъюгированные первичные антитела к пермскому и окрашивающему буферу в экспериментально определенных разведениях (все виды или изотипы иммуноглобулинов первичных антител должны быть разными и различимыми по вторичным антителам, которые исследователь намерен использовать на шаге 1.12).
- Слейте буфер для химической завивки и блокировки и используйте салфетку, чтобы удалить излишки буфера, не касаясь непосредственно ткани. Добавьте к ткани достаточное количество перманентной завивки и окрашивающего буфера с первичными антителами, чтобы покрыть ее, и инкубируйте предметное стекло при температуре 4 °C в течение 12-48 часов.
Примечание: Исследователь должен будет определить оптимальное время для инкубации антител. - Подготовьте буфер блокировки и буфер для окрашивания (табл. 1).
- Добавляйте вторичные антитела в окрашивающий буфер, приготовленный на шаге 1.11 при экспериментально определенных разведениях. Используемые вторичные антитела должны быть нацелены на те же виды или изотипы антител, которые были использованы на этапе 1.9.
- Подготовьте буфер для закалки (как показано в таблице 1).
ПРИМЕЧАНИЕ: Этот шаг помогает снизить аутофлуоресценцию от тканей. Если в этом нет необходимости, шаги 1.13 и 1.14 можно пропустить. - Промойте салфетки, как описано в шагах 1.5-1.6. Затем добавьте достаточное количество буфера для гашения, чтобы покрыть ткани на предметных стеклах, и инкубируйте стекла при комнатной температуре в течение 1-2 минут.
- Промойте салфетку, как описано в пунктах 1.5-1.6. Затем добавляют вторичные антитела, разведенные в окрашивающем буфере, приготовленном на стадии 1.12, в ткани таким образом, чтобы ткани были полностью покрыты окрашивающим буфером. Выдерживать предметные стекла при температуре 4 °C в течение 2-8 часов.
- Приготовьте блокирующий раствор путем добавления 50 мкл нормальной сыворотки из любого вида антител, которые будут использоваться на шаге 1.18 (5% каждого вида) в блокирующий буфер, приготовленный на шаге 1.11.
- Промойте салфетки, как описано в шагах 1.5-1.6. Затем добавьте блокирующий раствор с обычной сывороткой, приготовленной на шаге 1.16, на ткани так, чтобы они были полностью покрыты окрашивающим буфером. Инкубируйте предметные стекла при комнатной температуре в течение 1-8 часов.
- Окрашивающий раствор готовят путем добавления флуорофор-конъюгированных первичных антител в экспериментально определенных разведениях в окрашивающий буфер, приготовленный на шаге 1.11. Кроме того, добавьте ядерное красило, например, ядерный желтый, в раствор для окрашивания при экспериментально определенном разбавлении.
ПРИМЕЧАНИЕ: Шаги 1.18-1.19 требуются только в том случае, если исследователю необходимо использовать дополнительные метки антител, которые не требуют вторичных антител для усиления сигнала. Если не используются первичные конъюгированные антитела, на шаге 1.12 можно добавить красители для ядер. - Стряхните блокирующий буфер и с помощью салфетки удалите излишки буфера, не касаясь непосредственно ткани. Добавьте в ткань достаточное количество раствора для окрашивания, приготовленного на шаге 1.18, чтобы покрыть ее, и инкубируйте при температуре 4 °C в течение 12-48 часов.
- Промойте салфетки, как описано в шагах 1.5-1.6. Добавьте 5 капель монтанта на каждое стекло, не вводя пузырьков, и осторожно и медленно наклоните стеклянный покровный стекло на ткань, чтобы полностью покрыть ткань монтантом и исключить все пузырьки воздуха. Храните предметные стекла при температуре 4 °C в темноте до тех пор, пока они не будут готовы к съемке.
2. Визуализация срезов тканей
Примечание: На этой стадии срезы ткани окрашивают всеми антителами, представляющими интерес для всех состояний, которые предполагается визуализировать, а одноцветные срезы окрашивают одним флуоресцентным параметром на каждом участке. Визуализация должна быть сделана на микроскопе, который имеет несколько детекторов, которые могут быть настроены на обнаружение определенных диапазонов света, и в идеале он должен иметь доступ к как можно большему количеству лазерных линий для возбуждения.
- Вставьте в микроскоп один из полностью окрашенных предметных стекол и настройте микроскоп так, чтобы он получил изображение среза с помощью объектива, который предназначен для сбора данных.
- Основываясь на флуорофорах, которыми окрашена ткань, определите возбуждающие лазерные линии, необходимые для стимуляции этих флуорофоров, и определите диапазоны излучения для каждого флуорофора, которые содержат наибольшее количество сигнала.
ПРИМЕЧАНИЕ: Онлайн-инструменты просмотра флуоресцентных спектров (например, см. Таблицу материалов) могут быть полезны для определения оптимальных длин волн возбуждения флуорофоров и определения оптимальных диапазонов для обнаружения флуорофоров. Флуорофоры, используемые для гистопроточной цитометрии, должны быть подобраны таким образом, чтобы каждый флуорофор отличался оптимальной лазерной линией, которой стимулируется флуорофор, или отличался оптимальным диапазоном длин волн излучения. Флуорофоры, которые имеют более близкие длины волн возбуждения/излучения, будет труднее разделить. Флуорофоры, использованные в этом исследовании, подробно описаны в разделе результатов. - Откройте программное обеспечение 1 (см. Таблицу материалов) в конфокальном микроскопе и выберите вкладку конфигурации вверху. Затем выберите аппаратную кнопку слева и измените битовую глубину на 16-бит.
ПРИМЕЧАНИЕ: Для сбора данных рекомендуется использовать более высокие битовые глубины, такие как 16-битные данные, так как это повысит точность спектральной компенсации. Кроме того, более высокая плотность пикселей, большие числа для средних линий и большее количество ступеней в z-стеке улучшат качество спектрального разделения. - Выберите вкладку «Захват » в верхней части экрана и введите настройку формата, чтобы включить плотность изображения в пикселях (как правило, чем выше разрешение, тем лучше результаты).
- Если это возможно, выберите двунаправленное X-сканирование , чтобы ускорить время получения изображения, при условии, что эта функция была должным образом откалибрована.
- Выберите значение точечного отверстия для конфокального микроскопа (обычно в диапазоне 400-600 нм), при котором меньшие значения обычно дают более качественные результаты гистопроточной цитометрии, хотя меньшие значения точечных отверстий затрудняют получение изображений и могут потребовать дополнительного времени визуализации или более высокой интенсивности возбуждения для получения изображений.
- Выберите «Показать панель последовательного сканирования » в верхней части левого меню, чтобы открыть параметр «Последовательное сканирование». Используйте знак '+' для настройки дополнительных последовательностей, где каждая последовательность будет содержать одну длину волны возбуждения, указанную на шаге 2.2.
- Включите все лазеры, которые необходимы, нажав на кнопки ВКЛ, связанные с интересующими лазерами в центре экрана, чтобы перевести их в состояние ВКЛ. Перейдите к каждой конкретной последовательности и увеличьте мощность лазера в центре экрана до любого желаемого значения, где каждая последовательность будет иметь одну длину волны возбуждения.
- Включите столько детекторов, сколько необходимо в нижней части экрана в каждой последовательности, чтобы отобразить все флуорофоры, которые будут отображаться на этой лазерной линии. Отрегулируйте диапазоны детекторов, дважды щелкнув по их диапазонам и введя верхний и нижний диапазоны длин волн, которые необходимо собрать.
ПРИМЕЧАНИЕ: Рекомендуется по возможности использовать высокочувствительные детекторы, а в идеале все каналы должны собираться на одних и тех же типах детекторов, хотя это и не обязательно. - Нажмите кнопку реального времени, затем сосредоточьтесь на образце. Используйте кнопку «Автомасштабирование» слева от изображения в программном обеспечении, чтобы оценить, достаточно ли насыщены каналы сигналом. Пользователь должен регулировать значения усиления и интенсивности лазера в каждой последовательности до тех пор, пока автомасштабирование не достигнет максимального значения (65535 для 16-битных данных), но будет очень близко к насыщению для всех каналов.
Примечание: Поскольку различные флуорофоры могут различаться по своей яркости, а различные мишени антител могут различаться по их количеству, чувствительность детекторов должна быть отрегулирована таким образом, чтобы она была низкой для ярких сигналов и высокой чувствительностью для тусклых сигналов, если оба флуорофора стимулируются одним и тем же лазером (интенсивность лазера не может измениться в этом сценарии, так как будут затронуты оба флуорофора). Для достижения наилучших результатов флуоресцентный сигнал от каждого канала в эксперименте должен быть сбалансирован таким образом, чтобы каждый канал имел примерно одинаковое количество сигнала, что уменьшит спектральное просачивание в другие каналы и приведет к усилению сигналов. Поскольку различные образцы могут различаться по обилию окрашиваемых маркеров, рекомендуется протестировать настройки микроскопа в нескольких экспериментальных условиях, чтобы подтвердить, что обесцвечивание не происходит ни в одном из экспериментальных условий. - После того, как настройки микроскопа будут завершены, загрузите в микроскоп одинарные стекла для управления цветом и найдите репрезентативное окрашивание для каждого флуорофора. Настройте Z-стек, сфокусировавшись под образцом и нажав кнопку начала в меню стека Z, затем сфокусируйтесь над образцом и нажав кнопку конца .
- Выберите количество шагов и расстояние между шагами (и используйте такое же количество шагов и параметров, таких как плотность пикселей и битовая глубина, которые будут использоваться для изображения полностью окрашенных слайдов), затем нажмите кнопку запуска.
ПРИМЕЧАНИЕ: В идеале эти изображения должны включать в себя самые яркие сигналы от реального окрашивания, которые можно найти на предметном стекле. - После того, как элементы управления одним цветом будут изображены, продолжайте использовать те же настройки микроскопа для изображения всех полностью окрашенных образцов.
3. Компенсация флуоресценции
- Откройте файлы .lif в программе Software 1 и перейдите на вкладку процесса.
- Выберите модуль «Разделение красителей», а затем выберите опцию «Автоматическое разделение красителей». Выберите ручной метод для разделения красителя и выберите «Без изменения масштаба».
- Откройте одно из одноцветных изображений элемента управления. Вручную проверьте одноцветные элементы управления на предмет проникновения спектра из одного канала в другой, наблюдая за серыми окнами для каждого канала на наличие сигнала в неподходящих каналах. При необходимости используйте кнопку «Автомасштабирование », чтобы увидеть слабое просвечивание.
- Начните удаление просвечивания, вручную введя числа (обычно 0-1, где 0 означает отсутствие просвечивания, а 1 — 100% просвечивания) в матрицу на экране, а затем нажмите кнопку «Применить », чтобы проверить, адекватно ли удалено просвечивание. Это следует повторять до тех пор, пока сигналы во всех других каналах, кроме интересующего канала, не будут снижены до фоновых уровней.
ПРИМЕЧАНИЕ: Чрезмерная компенсация приведет к неточным результатам, поэтому флуоресценция должна быть снижена до фоновых уровней, но не ниже их. Данные с избыточной компенсацией будут отображаться как один канал с сигналом, а другие каналы будут иметь черные дыры там, где находится интересующий сигнал (рис. 2). - После завершения работы над одним из одноцветных элементов управления запишите значения в матрицу, а затем сбросьте матрицу и перейдите к следующему одноцветному элементу управления. Повторите этот процесс для каждого отдельного элемента управления цветом.
ПРИМЕЧАНИЕ: Первый краситель будет находиться в ряду Краситель 1, а количество флуоресценции, которое необходимо удалить из Красителя 1, будет находиться в соседних столбцах. - Соберите все значения, полученные от каждого элемента управления, в одну матрицу и примените эту матрицу к полностью окрашенному образцу. Если разделение выглядит точным, примените настройки ко всем изображениям в эксперименте, в противном случае повторно оцените элементы управления одним цветом.
ПРИМЕЧАНИЕ: Эффективная компенсация должна устранять большую часть утечки через каналы, как показано на рисунке 3. - Сохраните все компенсированные файлы в отдельный файл .lif.
4. Идентификация ядер и клеток с помощью ilastik
- Откройте компенсированные файлы .lif в FIJI20. Включите просмотр стеков в гиперстеке и выберите изображение.
- (Дополнительный) Используйте инструмент выделения, такой как инструмент «Прямоугольник», чтобы включить части изображения, которые будут проанализированы, и исключить области, которые не будут анализироваться. Обрежьте изображение с помощью функции обрезки изображения >.
ПРИМЕЧАНИЕ: Уменьшение размера изображения полезно на последующих этапах для уменьшения потребления оперативной памяти (ОЗУ) и увеличения скорости обработки. - Сохраните изображение как файл .tif. Кроме того, сохраните изображение как файл hdf5 с помощью плагина ilastik для FIJI, нажав на Плагины > ilastik > Экспорт hdf5. Сохраните файл как "name of choice".h5 со сжатием 0.
- Откройте Ilastik21 и создайте проект по классификации пикселей. Используйте функцию Добавить новое > Добавить отдельные изображения , чтобы добавить как минимум 3 репрезентативных изображения, перейдя к добавлению новых входных данных.
- Щелкните правой кнопкой мыши одно из изображений, выберите «Редактировать свойства» и измените режим отображения на оттенки серого.
- В левой части экрана выберите вкладку выбора функций. Нажмите на Select features и выберите все функции до максимального значения sigma, которое программа позволит вам использовать, затем нажмите OK.
- Теперь выберите вкладку Обучение. С помощью инструмента «Кисть» (когда выбрана метка номер один ) выделите отдельные клетки (рис. 4) таким образом, чтобы внутренняя часть (цитоплазма и ядро) и клеточная мембрана были включены в выделение.
- Повторите это для всех обучающих образцов, где выбраны все типы клеток, представляющие интерес, и из каждого образца должно быть выбрано несколько клеток различной интенсивности, морфологии и локализации в ткани.
ПРИМЕЧАНИЕ: В зависимости от маркеров, выбранных для индивидуального набора красителей, могут быть или не быть внутриклеточные окрашивания или окрашивания клеточных мембран для интересующих клеток. Какие бы маркеры ни использовались, убедитесь, что выделение включает в себя самую крайнюю краску на интересующих клетках и внутреннюю часть клетки, независимо от того, используется ли внутриклеточное окрашивание. - Затем используйте метку два , чтобы выделить все, что не является интересующими вас ячейками (рисунок 4). Это может быть пустое пространство, немеченые ядра или ткань, которая не имеет маркеров, представляющих интерес. Обязательно включите пространство, непосредственно прилегающее к интересующим клеткам, чтобы способствовать изучению точных границ клеток.
- Используйте функцию Live Update для оценки качества обучения на интересующих вас изображениях. Если обучение является недостаточным, продолжайте обучение с шагов 4.7 - 4.9. В противном случае, если все устраивает, перейдите на вкладку «Экспорт прогнозов ».
- Задайте в качестве источника значение вероятностей. Нажмите кнопку «Выбрать параметры экспорта изображения », затем установите параметр c на экспорт 0-1 (предполагая, что метка 1 выделяет интересующие параметры).
- Преобразуйте тип данных в 16-битное целое число и экспортируйте как файл hdf5 в интересующий каталог с именем интересующего вас (мы рекомендуем использовать {nickname} mask.h5), затем нажмите OK.
- Перейдите на вкладку Пакетная обработка . Нажмите кнопку Select Raw Data files , выберите все интересующие вас файлы .h5, требующие обработки, а затем нажмите кнопку Open. Нажмите « Обработать все файлы».
- Повторите шаги с 4.4 по 4.13, за исключением того, что вместо идентификации клеток теперь определите ядра и исключите все остальное (рисунок 4). Экспортируйте это как отдельный прогноз для идентификации ядра клеток.
- Откройте Ilastik и создайте новый проект преобразования данных. Добавьте репрезентативный файл hdf5 на вкладку Входные данные с помощью функции Добавить новый > Добавить отдельное изображение .
- Перейдите на вкладку «Экспорт данных» и нажмите кнопку «Выбрать настройки экспорта изображения», установив источник в качестве ввода. Измените формат на многостраничный tiff, выберите место сохранения с помощью функции выбора и установите имя файла на {nickname}. Нажмите OK.
- Перейдите на вкладку «Пакетная обработка» и выберите все файлы hdf5 с помощью кнопки « Выбрать файлы входных данных » и нажмите кнопку «Обработать все файлы ».
- Откройте программу Software 2 (см. Таблицу материалов) и добавьте все файлы tif/tiff, созданные на шагах 4.3 и 4.17, с помощью кнопки « Добавить файлы ». Выберите место для вывода файлов и нажмите кнопку «Начать все », чтобы преобразовать все файлы в файлы .ims. Теперь файлы готовы к анализу в программном обеспечении 3 (см. Таблицу материалов).
5. Анализ в программном обеспечении 3
- Откройте одно из изображений, содержащее все флуоресцентные пятна. Выберите Edit-> Add Channels и добавьте файлы .ims, соответствующие маскам, созданным над ядрами и телами клеток. Повторите это для всех образцов.
- Нажмите кнопку « Добавить новые ячейки ». Выберите опцию «Обнаружить ядро и клетку » и продолжайте двигаться дальше.
- Выберите маску над ядрами в качестве исходного канала для обнаружения ядер и выберите расширенную опцию разделения ядер по начальным точкам. Выберите значение диаметра ядра) и продолжайте двигаться дальше.
ПРИМЕЧАНИЕ: Оптимальное значение диаметра ядра необходимо проверить, запустив моделирование и вручную оценив, не расщепляет ли программа достаточное количество ядер (несколько ядер группируются вместе) или имеет место чрезмерное расщепление (одно ядро разбивается на более чем одно). - Выберите значение для оценки качества и продолжите. Как правило, лучше всего включать все ядра, так как пропуск ядер может привести к слиянию нескольких клеток в одну.
- Выберите пороговое значение для создания ядерных поверхностей и продолжите. Убедитесь, что все ядра, даже более тусклые, все еще включены в порог. Не делайте порог слишком низким, так как это может привести к завышению размера ядер или потенциальному включению обломков в качестве ядерного окрашивания.
- Выберите значение минимального количества вокселей на ядро и продолжайте. Как правило, лучше всего сохранить все, если только с помощью этого параметра нельзя исключить мусор или неядерные частицы.
- Выберите опцию Граница клетки и цитоплазма , выберите маску над клетками , которая будет исходным каналом для обнаружения клеточных тел, а затем продолжите.
- Выберите пороговое значение ячейки, которое точно захватывает все интересующие клетки, но не настолько низкое, чтобы включать фоновое или неспецифическое обнаружение клеток. Выберите вариант разделения соприкасающихся клеток таким образом, чтобы в каждой клетке было одно ядро, и они были отделены расстоянием от ядер. Кроме того, выберите расширение клеток на ядрах , а затем продолжите.
- Выберите значение минимального количества вокселей в ячейке и продолжайте. Как правило, лучше всего сохранить все, если только с помощью этого параметра нельзя исключить мусор или неклетки.
- Оцените, являются ли созданные поверхности над ячейками точными, посмотрев, не были ли несколько ячеек слиты вместе или отсутствуют ячейки. Если поверхности неадекватны, вернитесь к шагу 5.2 и попробуйте новые параметры, пока не будут определены оптимальные настройки.
- После того, как наилучшие настройки будут установлены, нажмите на созданный объект ячеек в левом меню, перейдите на вкладку создания и нажмите кнопку «Сохранить параметры для пакета », чтобы сохранить использованные настройки.
- Откройте несколько окон Программы 3 и откройте отдельные изображения в каждом окне. Нажмите новую кнопку «Ячейки » в каждом окне, выберите сохраненные настройки в меню «Параметры создания избранного » и укажите каждому окну работать до завершения. Повторяйте это столько раз, сколько необходимо, пока все образцы не будут проанализированы.
ПРИМЕЧАНИЕ: Запуск нескольких экземпляров программного обеспечения 3 ускорит выполнение анализа, но количество окон, которые могут быть запущены параллельно, будет зависеть от скорости обработки центрального процессора (ЦП) и объема оперативной памяти компьютера, на котором выполняется анализ. - После того, как все образцы будут завершены, нажмите на объект созданных ячеек в левом меню на каждом изображении и перейдите на вкладку «Статистика». Нажмите кнопку «Настроить список видимых значений статистики», отмените выбор всех статистических данных, кроме значения «Среднее значение интенсивности ячейки», а затем нажмите OK.
- Для каждого изображения, которое необходимо проанализировать, нажмите кнопку Экспортировать всю статистику в файл на вкладке статистики и сохраните файлы в нужном месте.
6. Анализ гистопотоковой цитометрии и картирование популяций на срезы тканей
- Откройте Anaconda и JupyterLab в Anaconda. Затем откройте скрипт, представленный в Файле дополнительного кодирования 1. Запустите код с помощью меню «Выполнить » и выберите «Запустить все ячейки» или запустите ячейки по отдельности, выбрав «Запустить выбранные ячейки».
- При появлении запроса на ввод исходного файла введите каталог файла для экспортированных значений флуоресценции, сгенерированных на шаге 5.14. Каталог файлов можно получить, посмотрев свойства одного из экспортируемых файлов .csv и скопировав путь к файлу в командной строке.
- Далее программа запросит место вывода обработанных файлов. Введите каталог файлов в любое место, подходящее для сохранения обработанных файлов. Программа автоматически определит количество флуоресцентных каналов, содержащихся в папке.
- Выполните следующий раздел кода, чтобы аннотировать данные с именами каждого канала. Этот код можно изменить по мере необходимости, чтобы изменить параметр "numberOfChannels" в соответствии с количеством каналов, обнаруженных на шаге 6.3. Кроме того, имена каналов могут быть изменены для необходимого количества каналов, повторяя строку 'ChX':'Marker' столько раз, сколько необходимо, и изменяя имена маркеров.
- Выполнение следующей строки кода создаст файл .csv в указанном месте, который будет аннотирован и содержит все флуоресцентные параметры в наборе данных. Повторите шаги 6.1-6.5 для каждого набора экспортируемых данных из программного обеспечения 3, чтобы создать .csv файлы для всех данных.
- Преобразуйте файлы .csv в файлы .fcs, открыв программу Software 4 (см. Таблицу материалов) и перетащив файлы .csv в окно программы. Программное обеспечение 4 автоматически начнет преобразование файлов в файлы .fcs.
- Дважды щелкните один из примеров в программе Software 4, чтобы открыть окно с этим образцом. Используйте инструменты стробирования в программе 4 для создания стратегии стробирования. Рекомендуется начать с гейтирования на маркере, определяющем родословную, содержащем все интересующие клетки, затем перейти к гейтам, которые сначала удаляют более редкие подмножества, а затем закончить на более распространенных типах клеток.
- После того, как стратегия стробирования установлена, проверьте ее точность, щелкнув правой кнопкой мыши по этой популяции в меню и выбрав «Экспорт». Экспортируйте все параметры в виде файла .csv с включенными заголовками.
- Откройте JupyterLab в Анаконде. Затем откройте скрипт, представленный в файле дополнительного кодирования 2, и запустите код. При появлении запроса на ввод местоположения файла Software 4 введите каталог для экспортируемого файла .csv, созданного на предыдущем шаге. Каталог файлов можно получить, посмотрев на свойства экспортируемого файла .csv и скопировав путь к файлу в командной строке.
- Когда появится запрос на добавление места вывода, выберите любой каталог файлов по выбору и убедитесь, что каталог файлов содержит имя файла в конце («каталог файлов»\filename.txt). Выполните оставшуюся часть кода.
- Скопируйте текст в файле .txt и откройте соответствующий файл .ims в программе Software 3 для того же образца, на котором основаны экспортируемые данные. Нажмите на объект Cell в файле, переключитесь на вкладку статистики, вставьте текст в строку поиска и начните поиск. Все интересующие ячейки будут подсвечены.
- Вручную проверьте, все ли интересующие клетки были захвачены с помощью стратегии стробирования. Если количество ячеек недооценено или переоценено (примеры показаны на рисунке 5), вернитесь к шагу 6.7, отрегулируйте вентиль и повторно оцените, точно ли ячейки захватываются новым вентом. Повторите то же самое со всеми стробами в стратегии стробирования и оцените стробирование на нескольких образцах.
- Когда все вентили будут проверены, экспортируйте данные из программного обеспечения 4 с помощью функции редактора таблиц и проанализируйте данные с использованием предпочтительного метода анализа.
Примечание: Клеточные популяции могут быть отображены обратно на участки ткани в программном обеспечении 3 путем идентификации закрытой популяции, как показано на этапах 6.8-6.11, а затем создания поверхности на основе выбранных клеток. Это можно сделать для каждой закрытой популяции, как показано на рисунках 6 и 7.
Результаты
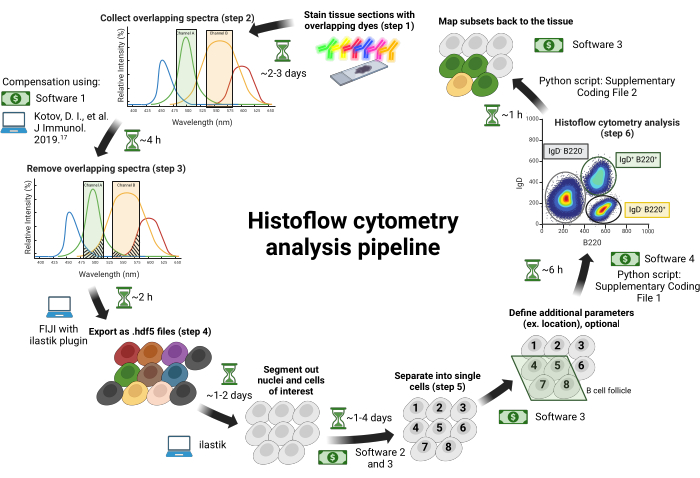
Рисунок 1: Рабочий процесс гистопроточной цитометрии. Срезы тканей окрашиваются спектрально перекрывающимися красителями (шаг 1). Изображения собираются с помощью отдельных возбуждающих лаз...
Обсуждение
Здесь описывается использование гистопроточной цитометрии, метода, который был валидирован ранее18. Показано, что при окрашивании срезов тканей спектрально перекрывающимися красителями это просвечивание через каналы может быть удалено с помощью спектральной компенсаци?...
Раскрытие информации
У авторов отсутствует финансовый конфликт интересов.
Благодарности
Мы благодарим передовую платформу микроскопии Института мозга Гочкиса за инфраструктуру визуализации и опыт. RWJ был поддержан стипендией для постдокторантуры в рамках программы Университета Калгари Eyes High и неограниченной образовательной стипендией Канадского общества рассеянного склероза и Roche Canada. VWY получал заработную плату в рамках программы Canada Research Chair Tier 1. Эта работа была поддержана операционными фондами из гранта Канадского института исследований в области здравоохранения 1049959, гранта 3236 Канадского общества рассеянного склероза и Программы исследований рассеянного склероза, направляемой Конгрессом Министерства обороны США. Рисунок 1 создан с помощью BioRender.com. Цифры, адаптированные в этой публикации, были первоначально опубликованы в The Journal of Immunology. Раджив В. Джейн, Дэвид А. Эллиотт и В. Ви Йонг. 2023. Одноклеточный анализ высокопараметрических гистологических изображений с использованием гистопрофлоуцитометрии. J. Immunol. 210: 2038-2049. Авторское право © [2023]. Американская ассоциация иммунологов, Inc.
Материалы
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
Ссылки
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены